カスタム関数を使用した信号のラベル付けの自動化
この例では、信号ラベラーのカスタム自動ラベル付け関数を使用して、心電図 (ECG) 信号の QRS 群と R ピークにラベル付けする方法を示します。1 つのカスタム関数は、学習済みの再帰型深層学習ネットワークを使用し、QRS 群を特定して検索します。もう 1 つのカスタム関数は、単純なピークの検索を使用して R ピークを検索します。この例では、ネットワークは、ネットワークの学習とテストのプロセスから完全に独立した 2 つの信号の QRS 群にラベル付けします。
ECG 波形の 3 つの偏位で構成される QRS 群は、心臓の左右の心室の脱分極を反映しています。また、QRS は人間の心拍の最大振幅セグメントでもあります。QRS 群の調査は、人の心臓の全体的な健康状態と異常の有無を評価するために役立ちます [1]。特に、QRS 群内の R ピークを検出し、連続するピークの時間間隔を調べることで、診断医は患者の心拍変動を計算して、心不整脈を見つけることができます。
この例の深層学習ネットワークは深層学習を使用した波形セグメンテーションで導入されており、公開されている QT データベースの ECG 信号を使用して学習させています [2] [3]。データは、合計 105 人の患者からの約 15 分間の ECG の記録で構成され、250 Hz でサンプリングされています。各記録を取得するために、検査員は患者の胸部の異なる場所に 2 つの電極を配置して、2 チャネル信号にしています。データベースは、自動化されたエキスパート システムによって生成される信号領域ラベルを提供します [1]。追加されたラベルにより、深層学習ネットワークにデータを使用できるようになっています。
信号ラベラーへのデータの読み込み、リサンプリング、およびインポート
この例では、MIT-BIH Arrhythmia Database の信号をラベル付けします [4]。データベース内の各信号は、平均 360 Hz のレートで不規則にサンプリングされ、2 人の心臓専門医によって注釈が付けられ、結果の検証が可能になっています。
記録 200 および 203 に対応する 2 つの MIT データベース信号を読み込みます。信号をサンプル時間 1/250 秒の等間隔グリッドにリサンプリングします。これは、QT データベース データの定格サンプル レートに対応します。
load mit200 y200 = resample(ecgsig,tm,250); load mit203 y203 = resample(ecgsig,tm,250);
信号ラベラーを開きます。[ラベラー] タブで [インポート] をクリックし、[メンバー] リストの From Workspace を選択します。ダイアログ ボックスで、信号 y200 および y203 を選択します。時間情報を追加します。ドロップダウン リストから Time を選択し、サンプル レートを 250 Hz に指定します。[インポートして閉じる] をクリックします。[ラベル付き信号セットのメンバー] ブラウザーに信号が表示されます。名前の隣にあるチェック ボックスをオンにして信号をプロットします。
ラベルの定義
ラベルを定義して、信号に付加します。
QRS 群のカテゴリカル関心領域 (ROI) ラベルを定義します。[ラベラー] タブの [定義の追加] をクリックします。[ラベル名] として
QRSregionsを指定し、[ラベル タイプ] にROIを選択して、[データ型] としてcategoricalと入力し、2 つの [カテゴリ]、QRSおよびn/aを 1 行に 1 つずつ追加します。QRSregionsのサブラベルを R ピークの数値の点ラベルとして定義します。[ラベルの定義] ブラウザーでQRSregionsをクリックして選択します。[定義の追加] をクリックし、Add sublabel definitionを選択します。[ラベル名] にRpeaksを指定し、[ラベル タイプ] にPointを選択し、[データ型] にnumericを入力します。

カスタム自動ラベル付け関数の作成
2 つのカスタム ラベル付け関数を作成します。1 つは QRS 群を検索してラベル付けし、もう 1 つは各 QRS 群内の R ピークを検索してラベル付けします。(関数 findQRS および findRpeaks のコードは、後ほどこの例で示します)。各関数を作成するために、[ラベラー] タブの [値の自動処理] ギャラリーを展開して、[カスタム関数の追加] を選択します。信号ラベラーにより、関数の名前、説明、およびラベル タイプを求めるダイアログ ボックスが表示されます。
QRS 群を検索する関数については、名前フィールドに
findQRSと入力し、[ラベル タイプ] としてROIを選択します。説明フィールドは空のままにすることも、独自の説明を入力することもできます。R ピークを検索する関数については、名前フィールドに
findRpeaksと入力し、[ラベル タイプ] としてPointを選択します。説明フィールドは空のままにすることも、独自の説明を入力することもできます。
関数を書き込み済みでその関数が現在のフォルダーまたは MATLAB® パスにある場合、信号ラベラーはその関数をギャラリーに追加します。関数をまだ書き込んでいない場合、コードの入力または貼り付けができるように、信号ラベラーはエディターで空白のテンプレートを開きます。ファイルを保存します。ファイルを保存すると、関数がギャラリーに表示されます。
QRS 群と R ピークのラベル付け
入力信号の QRS 群を見つけてラベル付けします。
[ラベル付き信号セットのメンバー] ブラウザーで、
y200の横にあるチェック ボックスをオンにします。[ラベルの定義] ブラウザーで
QRSregionsを選択します。[値の自動処理] ギャラリーで、
findQRSを選択します。[自動ラベル付け] をクリックし、
Auto-Label All Signalsを選択します。表示されるダイアログ ボックスで、[引数] フィールドに250Hz のサンプル レートを入力し、[OK] をクリックします。
信号ラベラーは、すべての信号の QRS 群を検索してラベル付けしますが、チェック ボックスをオンにした信号のラベルのみを表示します。QRS 群が、プロットとラベル ビューアーの座標軸に影付きの領域として表示されます。[表示] タブの [パナー] をクリックしてパナーをアクティブにし、ラベル付き信号の領域を拡大します。

QRS 群に対応する R ピークを見つけてラベル付けします。
[ラベルの定義] ブラウザーで
Rpeaksを選択します。[ラベラー] タブに戻ります。[値の自動処理] ギャラリーで、
findRpeaksを選択します。[自動ラベル付け] をクリックし、
Auto-Label All Signalsを選択します。表示されるダイアログ ボックスで [OK] をクリックします。
ラベルとその数値が、プロットとラベル ビューアーの座標軸に表示されます。

ラベル付き信号のエクスポートと心拍変動の計算
ラベル付き信号をエクスポートして、各患者の心拍変動を比較します。[ラベラー] タブで [エクスポート] をクリックし、[ラベル付き信号セット] リストで To File を選択します。表示されるダイアログ ボックスで、ラベル付き信号セットに HeartRates.mat という名前を付け、オプションで簡単な説明を追加します。[エクスポート] をクリックします。
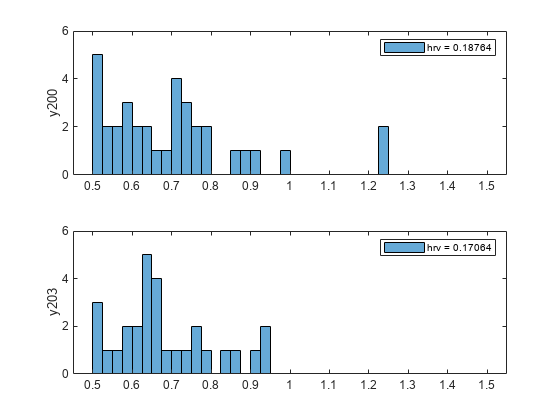
MATLAB® コマンド ウィンドウに戻ります。ラベル付き信号セットを読み込みます。セット内の各信号について、連続する心拍間の時間差の標準偏差として心拍変動を計算します。差のヒストグラムをプロットし、心拍変動を表示します。
load HeartRates nms = getMemberNames(heartrates); for k = 1:heartrates.NumMembers v = getLabelValues(heartrates,k,["QRSregions" "Rpeaks"]); hr = diff(cellfun(@(x) x.Location,v)); subplot(2,1,k) histogram(hr,0.5:0.025:1.5) legend("hrv = " + std(hr)) ylabel(nms(k)) ylim([0 6]) end
関数 findQRS: QRS 群の検索
関数 findQRS は、入力信号の QRS 群を見つけてラベル付けします。
この関数は、補助関数 computeFSST を使用して、入力データの形状を変更し、フーリエ シンクロスクイーズド変換 (FSST) を計算します。computeFSST を同じディレクトリ内の別のファイルに格納するか、最後の end ステートメントの前に挿入して、findQRS 内で入れ子にすることができます。
findQRS は関数classify (Deep Learning Toolbox)と学習済みの深層学習ネットワーク net を使用して、QRS 領域を特定します。深層学習ネットワークは、入力信号のすべての点を、P 領域、QRS 群、T 領域に属するとして、またはいずれにも属さないとしてラベル付けする categorical 配列を出力します。この関数は、signalMask を使用して QRS 群に対応する点ラベルを QRS 関心領域ラベルに変換し、残りを破棄します。df パラメーターは、持続時間が 20 サンプルよりも長い QRS 群のみを関心領域として選択します。サンプル レートを指定しない場合、関数は既定値の 250 Hz を使用します。
function [labelVals,labelLocs] = findQRS(x,t,parentLabelVal,parentLabelLoc,varargin) % This is a template for creating a custom function for automated labeling % % x is a matrix where each column contains data corresponding to a % channel. If the channels have different lengths, then x is a cell array % of column vectors. % % t is a matrix where each column contains time corresponding to a % channel. If the channels have different lengths, then t is a cell array % of column vectors. % % parentLabelVal is the parent label value associated with the output % sublabel or empty when output is not a sublabel. % parentLabelLoc contains an empty vector when the parent label is an % attribute, a vector of ROI limits when parent label is an ROI or a point % location when parent label is a point. % % labelVals must be a column vector with numeric, logical or string output % values. % labelLocs must be an empty vector when output labels are attributes, a % two column matrix of ROI limits when output labels are ROIs, or a column % vector of point locations when output labels are points. labelVals = cell(2,1); labelLocs = cell(2,1); if nargin<5 Fs = 250; else Fs = varargin{1}; end df = 20; load("trainedQTSegmentationNetwork","net") for kj = 1:size(x,2) sig = x(:,kj); % Reshape input and compute Fourier synchrosqueezed transforms mitFSST = computeFSST(sig,Fs); % Use trained network to predict which points belong to QRS regions netPreds = classify(net,mitFSST,MiniBatchSize=50); % Create a signal mask for QRS regions and specify minimum sequence length QRS = categorical([netPreds{1} netPreds{2}]',"QRS"); msk = signalMask(QRS,MinLength=df,SampleRate=Fs); r = roimask(msk); % Label QRS complexes as regions of interest labelVals{kj} = r.Value; labelLocs{kj} = r.ROILimits; end labelVals = vertcat(labelVals{:}); labelLocs = cell2mat(labelLocs); % Insert computeFSST here if you want to nest it inside findQRS. end
関数 computeFSST: 入力の形状変更とフーリエ シンクロスクイーズド変換の計算
この関数は、関数fsstを使用して、入力のフーリエ シンクロスクイーズド変換 (FSST) を計算します。深層学習を使用した波形セグメンテーションで説明されているように、ネットワークのパフォーマンスは、各学習信号またはテスト信号の時間-周波数マップが入力として与えられた場合に最も高くなります。FSST では、元の入力と同じ時間分解能で変換が行われるため、再帰型ネットワークで特に有用な特徴セットが得られます。関数は以下を実行します。
入力データに乱数をパディングし、
netが期待する 2 行 5000 列の cell 配列スタックに形状変更します。適切な周波数分解能を与えるために、長さが 128 のカイザー ウィンドウと既定の形状係数 を指定します。
0.5 Hz ~ 40 Hz の周波数範囲でデータを抽出します。
FSST の実数部と虚数部を個別の特徴として扱います。
平均を減算し、標準偏差で除算して、データを正規化。
function signalsFsst = computeFSST(xd,Fs) xd = reshape([xd;randn(10000-length(xd),1)/100],5000,2); signalsFsst = cell(1,2); for k = 1:2 [ss,ff] = fsst(xd(:,k),Fs,kaiser(128)); sp = ss(ff>0.5 & ff<40,:); signalsFsst{k} = normalize([real(sp);imag(sp)],2); end end
関数 findRpeaks: R ピークの検索
この関数は、findQRS により見つかった QRS 関心領域の最も突出したピークを検索します。この関数は、MATLAB® 関数 islocalmax を、findQRS により見つかった区間の信号の絶対値に適用します。
function [labelVals,labelLocs] = findRpeaks(x,t,parentLabelVal,parentLabelLoc,varargin) labelVals = zeros(size(parentLabelLoc,1),1); labelLocs = zeros(size(parentLabelLoc,1),1); for kj = 1:size(parentLabelLoc,1) tvals = t>=parentLabelLoc(kj,1) & t<=parentLabelLoc(kj,2); ti = t(tvals); xi = x(tvals); lc = islocalmax(abs(xi),MaxNumExtrema=1); labelVals(kj) = xi(lc); labelLocs(kj) = ti(lc); end end
参考文献
[1] Laguna, Pablo, Raimon Jané, and Pere Caminal."Automatic Detection of Wave Boundaries in Multilead ECG Signals: Validation with the CSE Database." Computers and Biomedical Research. Vol. 27, No. 1, 1994, pp. 45–60.
[2] Goldberger, Ary L., Luis A. N. Amaral, Leon Glass, Jeffery M. Hausdorff, Plamen Ch. Ivanov, Roger G. Mark, Joseph E. Mietus, George B. Moody, Chung-Kang Peng, and H. Eugene Stanley. "PhysioBank, PhysioToolkit, and PhysioNet: Components of a New Research Resource for Complex Physiologic Signals." Circulation. Vol. 101, No. 23, 2000, pp. e215–e220. [Circulation Electronic Pages: http://circ.ahajournals.org/content/101/23/e215.full].
[3] Laguna, Pablo, Roger G. Mark, Ary L. Goldberger, and George B. Moody. "A Database for Evaluation of Algorithms for Measurement of QT and Other Waveform Intervals in the ECG." Computers in Cardiology. Vol. 24, 1997, pp. 673–676.
[4] Moody, George B., and Roger G. Mark. "The impact of the MIT-BIH Arrhythmia Database." IEEE Engineering in Medicine and Biology Magazine. Vol. 20, No. 3, May–June 2001, pp. 45–50.