教師なし異常検出
このトピックでは、Statistics and Machine Learning Toolbox™ で使用できる多変量の標本データ向けの教師なし異常検出機能を紹介し、それらの機能による外れ値検出 (学習データ中の異常を検出) と新規性の検出 (汚染されていない学習データで新規のデータの異常を検出) のワークフローについて説明します。
ラベル付けされていない多変量の標本データについて、孤立森、ロバスト ランダム カット フォレスト、局所外れ値因子、1 クラス サポート ベクター マシン (SVM)、およびマハラノビス距離を使用して異常を検出できます。これらの手法では、モデルに学習させるかパラメーターを学習することで外れ値を検出します。新規性の検出では、汚染されていない学習データ (外れ値がないデータ) でモデルに学習させるかパラメーターを学習し、学習させたモデルまたは学習したパラメーターを使用して新規のデータの異常を検出します。
孤立森 — 孤立森アルゴリズムでは、孤立木のアンサンブルを使用して異常を正常な点から分離することで異常を検出します。関数
iforestを使用して外れ値を検出し、オブジェクト関数isanomalyを使用して新規性を検出します。ロバスト ランダム カット フォレスト — ロバスト ランダム カット フォレストアルゴリズムでは、点によって生じるモデルの複雑度の変化に基づいて、その点を正常な点または異常として分類します。孤立森アルゴリズムと同様に、ロバスト ランダム カット フォレスト アルゴリズムでは木のアンサンブルを作成します。2 つのアルゴリズムには、木の分岐変数の選択方法と異常スコアの定義方法に違いがあります。関数
rrcforestを使用して外れ値を検出し、オブジェクト関数isanomalyを使用して新規性を検出します。局所外れ値因子 — ローカルの外れ値因子 (LOF) アルゴリズムでは、周囲の近傍に対する観測値の相対的な密度に基づいて異常を検出します。関数
lofを使用して外れ値を検出し、オブジェクト関数isanomalyを使用して新規性を検出します。1 クラス サポート ベクター マシン (SVM) — 1 クラス SVM (教師なし SVM) では、変換された高次元予測子空間で原点からのデータの分離を試みます。関数
ocsvmを使用して外れ値を検出し、オブジェクト関数isanomalyを使用して新規性を検出します。マハラノビス距離 — 標本データが多変量正規分布に従っている場合、標本から分布までのマハラノビス平方距離はカイ二乗分布に従います。したがって、この距離を使用してカイ二乗分布の棄却限界値に基づいて異常を検出できます。外れ値の検出では、関数
robustcovを使用してロバストなマハラノビス距離を計算します。新規性の検出では、関数robustcovとpdist2を使用してマハラノビス距離を計算できます。
インクリメンタル学習の実行時に異常を検出するには、incrementalRobustRandomCutForest、incrementalOneClassSVM、およびIncremental Anomaly Detection Overviewを参照してください。
外れ値検出
この例では、5 つの教師なし異常検出法 (孤立森、ロバスト ランダム カット フォレスト、局所外れ値因子、1 クラス SVM、およびマハラノビス距離) による外れ値の検出のワークフローを示します。
データの読み込み
変数 feat と actid を含む humanactivity データ セットを読み込みます。変数 feat には、24,075 個の観測値に関する 60 個の特徴量から成る予測子データ行列が格納され、応答変数 actid には、観測値の身体動作 ID が整数として格納されています。この例では、変数 feat を異常検出に使用します。
load humanactivity変数 feat のサイズを調べます。
[N,D] = size(feat)
N = 24075
D = 60
データに含まれる外れ値の比率が 0.05 であると仮定します。
contaminationFraction = 0.05;
孤立森
関数iforestを使用して外れ値を検出します。
関数 iforest を使用して孤立森モデルに学習させます。外れ値の比率 (ContaminationFraction) を 0.05 と指定します。
rng("default") % For reproducibility [forest,tf_forest,s_forest] = iforest(feat, ... ContaminationFraction=contaminationFraction);
forest はIsolationForestオブジェクトです。iforest は、データ (feat) の異常インジケーター (tf_forest) および異常スコア (s_forest) も返します。iforest は、指定した比率の観測値が外れ値として検出されるようにスコアのしきい値 (forest.ScoreThreshold) を決定します。
スコア値のヒストグラムをプロットします。指定した比率に対応するスコアのしきい値に垂直線を作成します。
figure histogram(s_forest,Normalization="probability") xline(forest.ScoreThreshold,"k-", ... join(["Threshold =" forest.ScoreThreshold])) title("Histogram of Anomaly Scores for Isolation Forest")
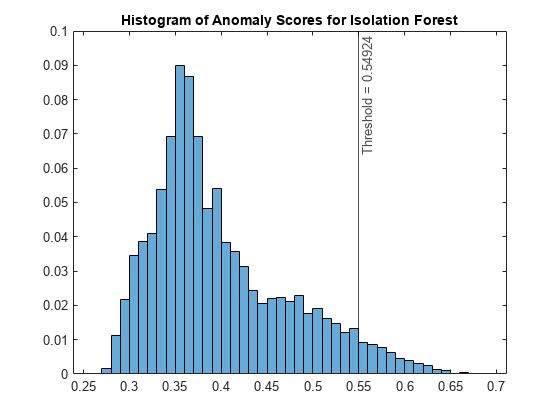
データ中の検出された異常の比率を確認します。
OF_forest = sum(tf_forest)/N
OF_forest = 0.0496
しきい値におけるスコアが同じになる値があると、外れ値の比率が指定した比率 (0.05) よりも小さくなることがあります。
ロバスト ランダム カット フォレスト
関数rrcforestを使用して外れ値を検出します。
関数 rrcforest を使用してロバスト ランダム カット フォレスト モデルに学習させます。外れ値の比率 (ContaminationFraction) を 0.05 と指定し、StandardizeData を true と指定して入力データを標準化します。
rng("default") % For reproducibility [rforest,tf_rforest,s_rforest] = rrcforest(feat, ... ContaminationFraction=contaminationFraction,StandardizeData=true);
rforest はRobustRandomCutForestオブジェクトです。rrcforest は、データ (feat) の異常インジケーター (tf_rforest) および異常スコア (s_rforest) も返します。rrcforest は、指定した比率の観測値が外れ値として検出されるようにスコアのしきい値 (rforest.ScoreThreshold) を決定します。
スコア値のヒストグラムをプロットします。指定した比率に対応するスコアのしきい値に垂直線を作成します。
figure histogram(s_rforest,Normalization="probability") xline(rforest.ScoreThreshold,"k-", ... join(["Threshold =" rforest.ScoreThreshold])) title("Histogram of Anomaly Scores for Robust Random Cut Forest")

データ中の検出された異常の比率を確認します。
OF_rforest = sum(tf_rforest)/N
OF_rforest = 0.0500
ローカルの外れ値因子
関数lofを使用して外れ値を検出します。
関数 lof を使用して局所外れ値因子モデルに学習させます。外れ値の比率 (ContaminationFraction) を 0.05 と指定し、500 個の最近傍とマハラノビス距離を指定します。
[LOFObj,tf_lof,s_lof] = lof(feat, ... ContaminationFraction=contaminationFraction, ... NumNeighbors=500,Distance="mahalanobis");
LOFObj はLocalOutlierFactorオブジェクトです。lof は、データ (feat) の異常インジケーター (tf_lof) および異常スコア (s_lof) も返します。lof は、指定した比率の観測値が外れ値として検出されるようにスコアのしきい値 (LOFObj.ScoreThreshold) を決定します。
スコア値のヒストグラムをプロットします。指定した比率に対応するスコアのしきい値に垂直線を作成します。
figure histogram(s_lof,Normalization="probability") xline(LOFObj.ScoreThreshold,"k-", ... join(["Threshold =" LOFObj.ScoreThreshold])) title("Histogram of Anomaly Scores for Local Outlier Factor")

データ中の検出された異常の比率を確認します。
OF_lof = sum(tf_lof)/N
OF_lof = 0.0500
1 クラス SVM
関数ocsvmを使用して外れ値を検出します。
関数 ocsvm を使用して 1 クラス SVM モデルに学習させます。外れ値の比率 (ContaminationFraction) を 0.05 と指定します。また、KernelScale を "auto" に設定して、関数でヒューリスティック手法を使用して適切なカーネル スケール パラメーターを選択できるようにし、StandardizeData を true に指定して入力データを標準化します。
[Mdl,tf_OCSVM,s_OCSVM] = ocsvm(feat, ... ContaminationFraction=contaminationFraction, ... KernelScale="auto",StandardizeData=true);
Mdl はOneClassSVMオブジェクトです。ocsvm は、データ (feat) の異常インジケーター (tf_OCSVM) および異常スコア (s_OCSVM) も返します。ocsvm は、指定した比率の観測値が外れ値として検出されるようにスコアのしきい値 (Mdl.ScoreThreshold) を決定します。
スコア値のヒストグラムをプロットします。指定した比率に対応するスコアのしきい値に垂直線を作成します。
figure histogram(s_OCSVM,Normalization="probability") xline(Mdl.ScoreThreshold,"k-", ... join(["Threshold =" Mdl.ScoreThreshold])) title("Histogram of Anomaly Scores for One-Class SVM")

データ中の検出された異常の比率を確認します。
OF_OCSVM = sum(tf_OCSVM)/N
OF_OCSVM = 0.0500
マハラノビス距離
関数robustcovを使用して、ロバストなマハラノビス距離、およびデータの平均と共分散のロバスト推定値を計算します。
関数 robustcov を使用して、feat から feat の分布までのマハラノビス距離を計算します。外れ値の比率 (OutlierFraction) を 0.05 と指定します。robustcov は、95% を超える観測値の共分散行列式を最小化します。
[sigma,mu,s_robustcov,tf_robustcov_default] = robustcov(feat, ...
OutlierFraction=contaminationFraction);robustcov は、ロバスト共分散行列の推定値 (sigma) とロバスト平均の推定値 (mu) を求めます。これらは、関数 cov および mean からの推定値よりも外れ値の影響が少なくなります。関数 robustcov は、マハラノビス距離 (s_robustcov) と外れ値インジケーター (tf_robustcov_default) も計算します。関数は、既定ではデータ セットが多変量正規分布に従うものと仮定し、カイ二乗分布の棄却限界値に基づいて入力観測値の 2.5% を外れ値として識別します。
データ セットが正規性の仮定を満たす場合、マハラノビス平方距離は自由度 D のカイ二乗分布に従います。ここで、D はデータの次元です。その場合、関数 chi2inv を使用して、指定した比率の観測値を外れ値として検出する新しいしきい値を見つけることができます。
s_robustcov_threshold = sqrt(chi2inv(1-contaminationFraction,D)); tf_robustcov = s_robustcov > s_robustcov_threshold;
距離-距離プロット (DD プロット) を作成して、データの多変量正規性を確認します。
figure d_classical = pdist2(feat,mean(feat),"mahalanobis"); gscatter(d_classical,s_robustcov,tf_robustcov,"kr",".x") xline(s_robustcov_threshold,"k-") yline(s_robustcov_threshold,"k-", ... join(["Threshold = " s_robustcov_threshold])); l = refline([1 0]); l.Color = "k"; xlabel("Mahalanobis Distance") ylabel("Robust Distance") legend("Normal Points","Outliers",Location="northwest") title("Distance-Distance Plot")
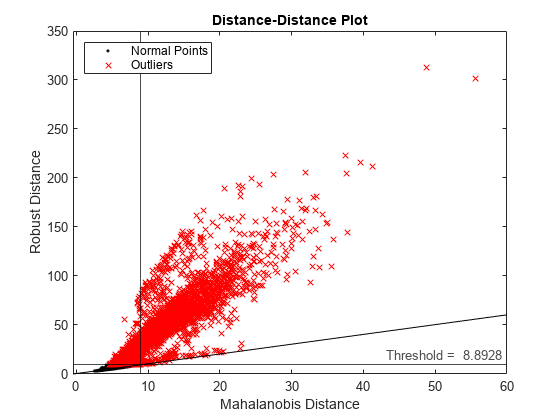
正常な点が見えるように座標軸を拡大します。
xlim([0 10]) ylim([0 10])
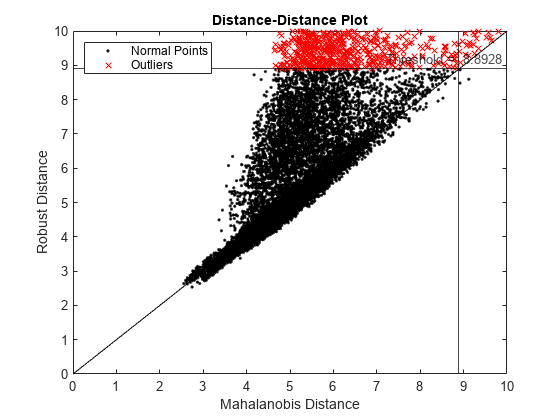
データ セットが多変量正規分布に従っていれば、データ点が 45 度の基準線の周囲に密集します。この DD プロットは、データ セットが多変量正規分布に従っていないことを示しています。
データ セットが正規性の仮定を満たさないため、累積確率に対する距離の値の分位数 (1 — contaminationFraction) を使用してしきい値を見つけます。
s_robustcov_threshold = quantile(s_robustcov,1-contaminationFraction);
新しいしきい値 s_robustcov_threshold を使用して feat の異常インジケーターを取得します。
tf_robustcov = s_robustcov > s_robustcov_threshold;
データ中の検出された異常の比率を確認します。
OF_robustcov = sum(tf_robustcov)/N
OF_robustcov = 0.0500
検出された外れ値の比較
検出された外れ値を可視化するには、関数tsneを使用してデータの次元を削減します。
rng("default") % For reproducibility T = tsne(feat,Standardize=true,Perplexity=100,Exaggeration=20);
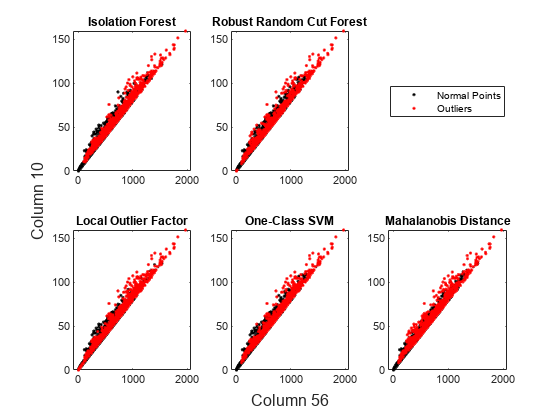
削減した次元で正常な点と外れ値をプロットします。孤立森アルゴリズム、ロバスト ランダム カット フォレスト アルゴリズム、局所外れ値因子アルゴリズム、1 クラス SVM モデル、および robustcov からのロバストなマハラノビス距離の 5 つの手法について結果を比較します。
figure tiledlayout(2,3) nexttile gscatter(T(:,1),T(:,2),tf_forest,"kr",[],[],"off") title("Isolation Forest") nexttile gscatter(T(:,1),T(:,2),tf_rforest,"kr",[],[],"off") title("Robust Random Cut Forest") nexttile(4) gscatter(T(:,1),T(:,2),tf_lof,"kr",[],[],"off") title("Local Outlier Factor") nexttile(5) gscatter(T(:,1),T(:,2),tf_OCSVM,"kr",[],[],"off") title("One-Class SVM") nexttile(6) gscatter(T(:,1),T(:,2),tf_robustcov,"kr",[],[],"off") title("Robust Mahalanobis Distance") l = legend("Normal Points","Outliers"); l.Layout.Tile = 3;
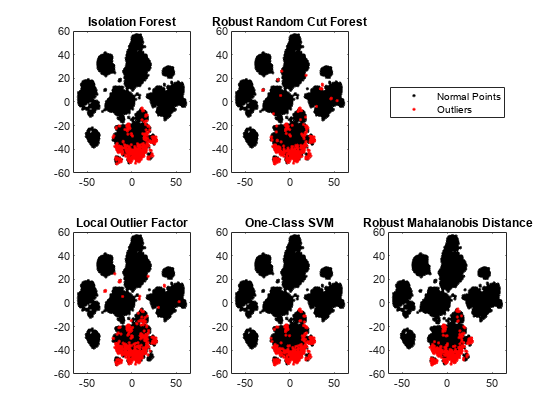
5 つの手法で識別された新規性は、削減した次元では互いに近い位置にあります。
また、関数fsulaplacianで選択された最も重要な 2 つの特徴量を使用して観測値を可視化することもできます。
idx = fsulaplacian(feat); figure t = tiledlayout(2,3); nexttile gscatter(feat(:,idx(1)),feat(:,idx(2)),tf_forest,"kr",[],[],"off") title("Isolation Forest") nexttile gscatter(feat(:,idx(1)),feat(:,idx(2)),tf_rforest,"kr",[],[],"off") title("Robust Random Cut Forest") nexttile(4) gscatter(feat(:,idx(1)),feat(:,idx(2)),tf_lof,"kr",[],[],"off") title("Local Outlier Factor") nexttile(5) gscatter(feat(:,idx(1)),feat(:,idx(2)),tf_OCSVM,"kr",[],[],"off") title("One-Class SVM") nexttile(6) gscatter(feat(:,idx(1)),feat(:,idx(2)),tf_robustcov,"kr",[],[],"off") title("Mahalanobis Distance") l = legend("Normal Points","Outliers"); l.Layout.Tile = 3; xlabel(t,join(["Column" idx(1)])) ylabel(t,join(["Column" idx(2)]))
新規性の検出
この例では、5 つの教師なし異常検出法 (孤立森、ロバスト ランダム カット フォレスト、局所外れ値因子、1 クラス SVM、およびマハラノビス距離) による新規性の検出のワークフローを示します。
データの読み込み
変数 feat と actid を含む humanactivity データ セットを読み込みます。変数 feat には、24,075 個の観測値に関する 60 個の特徴量から成る予測子データ行列が格納され、応答変数 actid には、観測値の身体動作 ID が整数として格納されています。この例では、変数 feat を異常検出に使用します。
load humanactivity関数cvpartitionを使用して、データを学習セットとテスト セットに分割します。観測値の 50% を学習データとして使用し、観測値の 50% を新規性の検出用のテスト データとして使用します。
rng("default") % For reproducibility c = cvpartition(actid,Holdout=0.50); trainingIndices = training(c); % Indices for the training set testIndices = test(c); % Indices for the test set XTrain = feat(trainingIndices,:); XTest = feat(testIndices,:);
学習データは汚染されていない (外れ値がない) ものと仮定します。
学習セットとテスト セットのサイズを調べます。
[N,D] = size(XTrain)
N = 12038
D = 60
NTest = size(XTest,1)
NTest = 12037
孤立森
関数iforestを使用して孤立森モデルに学習させた後、オブジェクト関数isanomalyを使用して新規性を検出します。
孤立森モデルに学習させます。
[forest,tf_forest,s_forest] = iforest(XTrain);
forest はIsolationForestオブジェクトです。iforest は、学習データ (XTrain) の異常インジケーター (tf_forest) および異常スコア (s_forest) も返します。既定では、iforest はすべての学習観測値を正常な観測値として扱い、スコアのしきい値 (forest.ScoreThreshold) を最大のスコア値に設定します。
学習済み孤立森モデルとオブジェクト関数 isanomaly を使用して、XTest 内の新規性を調べます。関数 isanomaly は、スコアがしきい値 (forest.ScoreThreshold) を超える観測値を新規性として識別します。
[tfTest_forest,sTest_forest] = isanomaly(forest,XTest);
関数 isanomaly は、テスト データの異常インジケーター (tfTest_forest) および異常スコア (sTest_forest) を返します。
スコア値のヒストグラムをプロットします。スコアのしきい値の位置に垂直線を作成します。
figure histogram(s_forest,Normalization="probability") hold on histogram(sTest_forest,Normalization="probability") xline(forest.ScoreThreshold,"k-", ... join(["Threshold =" forest.ScoreThreshold])) legend("Training data","Test data",Location="southeast") title("Histograms of Anomaly Scores for Isolation Forest") hold off
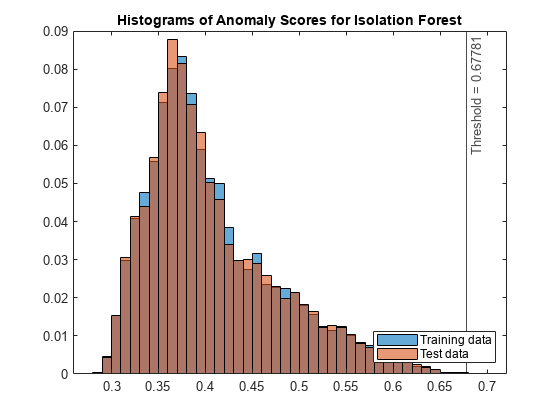
テスト データの異常スコア分布が学習データの異常スコア分布と類似しているため、isanomaly で検出されたテスト データ中の異常が少ないことがわかります。
テスト データ中の検出された異常の比率を確認します。
NF_forest = sum(tfTest_forest)/NTest
NF_forest = 8.3077e-05
テスト データ中にある異常の観測値のインデックスを表示します。
idx_forest = find(tfTest_forest)
idx_forest = 3422
ロバスト ランダム カット フォレスト
関数rrcforestを使用してロバスト ランダム カット フォレスト モデルに学習させた後、オブジェクト関数isanomalyを使用して新規性を検出します。
ロバスト ランダム カット フォレスト モデルに学習させます。StandardizeData を true と指定して入力データを標準化します。
[rforest,tf_rforest,s_rforest] = rrcforest(XTrain,StandardizeData=true);
rforest はRobustRandomCutForestオブジェクトです。rrcforest は、学習データ (XTrain) の異常インジケーター (tf_rforest) および異常スコア (s_rforest) も返します。既定では、rrcforest はすべての学習観測値を正常な観測値として扱い、スコアのしきい値 (rforest.ScoreThreshold) を最大のスコア値に設定します。
学習済みロバスト ランダム カット フォレスト モデルとオブジェクト関数 isanomaly を使用して、XTest 内の新規性を調べます。関数 isanomaly は、スコアがしきい値 (rforest.ScoreThreshold) を超える観測値を新規性として識別します。
[tfTest_rforest,sTest_rforest] = isanomaly(rforest,XTest);
関数 isanomaly は、テスト データの異常インジケーター (tfTest_rforest) および異常スコア (sTest_rforest) を返します。
スコア値のヒストグラムをプロットします。スコアのしきい値の位置に垂直線を作成します。
figure histogram(s_rforest,Normalization="probability") hold on histogram(sTest_rforest,Normalization="probability") xline(rforest.ScoreThreshold,"k-", ... join(["Threshold =" rforest.ScoreThreshold])) legend("Training data","Test data",Location="southeast") title("Histograms of Anomaly Scores for Robust Random Cut Forest") hold off
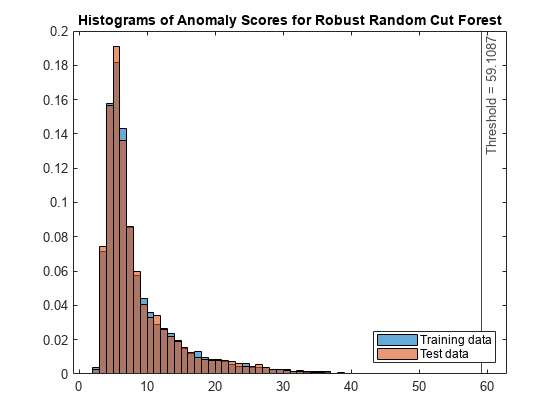
テスト データの異常スコア分布が学習データの異常スコア分布と類似しているため、isanomaly で検出されたテスト データ中の異常が少ないことがわかります。
テスト データ中の検出された異常の比率を確認します。
NF_rforest = sum(tfTest_rforest)/NTest
NF_rforest = 0
テスト データの異常スコア分布は学習データの異常スコア分布と類似しているため、isanomaly はテスト データ中にある異常を検出しません。
ローカルの外れ値因子
関数lofを使用して局所外れ値因子モデルに学習させた後、オブジェクト関数isanomalyを使用して新規性を検出します。
局所外れ値因子モデルに学習させます。
[LOFObj,tf_lof,s_lof] = lof(XTrain);
LOFObj はLocalOutlierFactorオブジェクトです。lof は、学習データ (XTrain) の異常インジケーター (tf_lof) および異常スコア (s_lof) を返します。既定では、lof はすべての学習観測値を正常な観測値として扱い、スコアのしきい値 (LOFObj.ScoreThreshold) を最大のスコア値に設定します。
学習済み局所外れ値因子モデルとオブジェクト関数 isanomaly を使用して、XTest 内の新規性を調べます。関数 isanomaly は、スコアがしきい値 (LOFObj.ScoreThreshold) を超える観測値を新規性として識別します。
[tfTest_lof,sTest_lof] = isanomaly(LOFObj,XTest);
関数 isanomaly は、テスト データの異常インジケーター (tfTest_lof) および異常スコア (sTest_lof) を返します。
スコア値のヒストグラムをプロットします。スコアのしきい値の位置に垂直線を作成します。
figure histogram(s_lof,Normalization="probability") hold on histogram(sTest_lof,Normalization="probability") xline(LOFObj.ScoreThreshold,"k-", ... join(["Threshold =" LOFObj.ScoreThreshold])) legend("Training data","Test data",Location="southeast") title("Histograms of Anomaly Scores for Local Outlier Factor") hold off
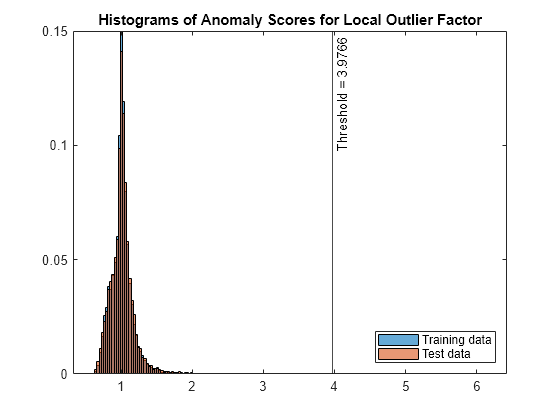
テスト データの異常スコア分布が学習データの異常スコア分布と類似しているため、isanomaly で検出されたテスト データ中の異常が少ないことがわかります。
テスト データ中の検出された異常の比率を確認します。
NF_lof = sum(tfTest_lof)/NTest
NF_lof = 8.3077e-05
テスト データ中にある異常の観測値のインデックスを表示します。
idx_lof = find(tfTest_lof)
idx_lof = 8704
1 クラス SVM
関数ocsvmを使用して 1 クラス SVM モデルに学習させた後、オブジェクト関数isanomalyを使用して新規性を検出します。
1 クラス SVM モデルに学習させます。KernelScale を "auto" に設定して、関数でヒューリスティック手法を使用して適切なカーネル スケール パラメーターを選択できるようにし、StandardizeData を true に指定して入力データを標準化します。
[Mdl,tf_OCSVM,s_OCSVM] = ocsvm(XTrain, ... KernelScale="auto",StandardizeData=true);
Mdl はOneClassSVMオブジェクトです。ocsvm は、学習データ (XTrain) の異常インジケーター (tf_OCSVM) および異常スコア (s_OCSVM) を返します。既定では、ocsvm はすべての学習観測値を正常な観測値として扱い、スコアのしきい値 (Mdl.ScoreThreshold) を最大のスコア値に設定します。
学習済み 1 クラス SVM モデルとオブジェクト関数 isanomaly を使用して、テスト データ (XTest) 内の新規性を調べます。関数 isanomaly は、スコアがしきい値 (Mdl.ScoreThreshold) を超える観測値を新規性として識別します。
[tfTest_OCSVM,sTest_OCSVM] = isanomaly(Mdl,XTest);
関数 isanomaly は、テスト データの異常インジケーター (tfTest_OCSVM) および異常スコア (sTest_OCSVM) を返します。
スコア値のヒストグラムをプロットします。スコアのしきい値の位置に垂直線を作成します。
figure histogram(s_OCSVM,Normalization="probability") hold on histogram(sTest_OCSVM,Normalization="probability") xline(Mdl.ScoreThreshold,"k-", ... join(["Threshold =" Mdl.ScoreThreshold])) legend("Training data","Test data",Location="southeast") title("Histograms of Anomaly Scores for One-Class SVM") hold off
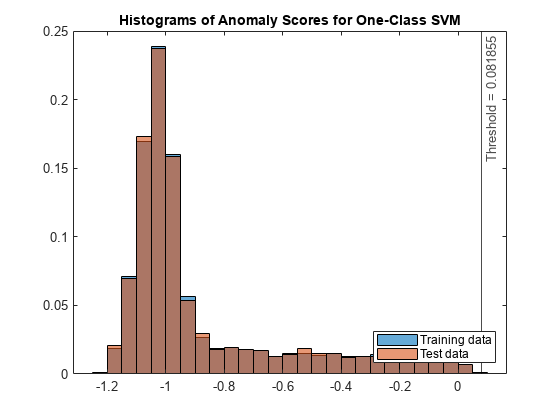
テスト データ中の検出された異常の比率を確認します。
NF_OCSVM = sum(tfTest_OCSVM)/NTest
NF_OCSVM = 1.6615e-04
テスト データ中にある異常の観測値のインデックスを表示します。
idx_OCSVM = find(tfTest_OCSVM)
idx_OCSVM = 2×1
3560
8316
マハラノビス距離
関数robustcovを使用して学習データのマハラノビス距離を計算し、関数pdist2を使用してテスト データのマハラノビス距離を計算します。
関数 robustcov を使用して、XTrain から XTrain の分布までのマハラノビス距離を計算します。外れ値の比率 (OutlierFraction) を 0 と指定します。
[sigma,mu,s_mahal] = robustcov(XTrain,OutlierFraction=0);
robustcov は、共分散行列の推定値 (sigma) と平均の推定値 (mu) も返します。これらを使用してテスト データの距離を計算できます。
s_mahal の最大値を新規性の検出用のスコアのしきい値として使用します。
s_mahal_threshold = max(s_mahal);
関数pdist2を使用して、XTest から XTrain の分布までのマハラノビス距離を計算します。
sTest_mahal = pdist2(XTest,mu,"mahalanobis",sigma);XTest の異常インジケーターを取得します。
tfTest_mahal = sTest_mahal > s_mahal_threshold;
スコア値のヒストグラムをプロットします。
figure histogram(s_mahal,Normalization="probability"); hold on histogram(sTest_mahal,Normalization="probability"); xline(s_mahal_threshold,"k-", ... join(["Threshold =" s_mahal_threshold])) legend("Training data","Test Data",Location="southeast") title("Histograms of Mahalanobis Distances") hold off
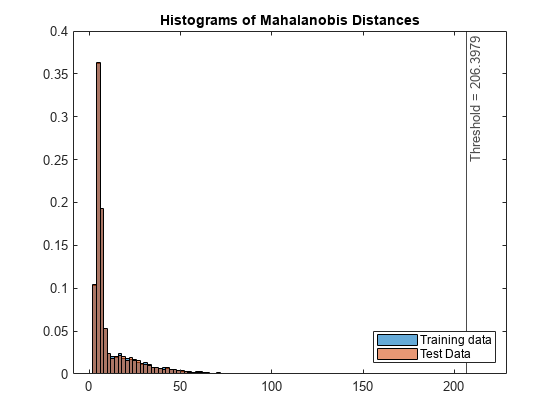
テスト データ中の検出された異常の比率を確認します。
NF_mahal = sum(tfTest_mahal)/NTest
NF_mahal = 8.3077e-05
テスト データ中にある異常の観測値のインデックスを表示します。
idx_mahal = find(tfTest_mahal)
idx_mahal = 3654
参考
iforest | isanomaly
(IsolationForest) | rrcforest | isanomaly
(RobustRandomCutForest) | lof | isanomaly
(LocalOutlierFactor) | ocsvm | isanomaly
(OneClassSVM) | robustcov | pdist2
トピック
- 孤立森による異常検出
- モデル固有の異常検出
- 3 軸振動データを使用した産業機械の異常の検出 (Predictive Maintenance Toolbox)