isanomaly
構文
説明
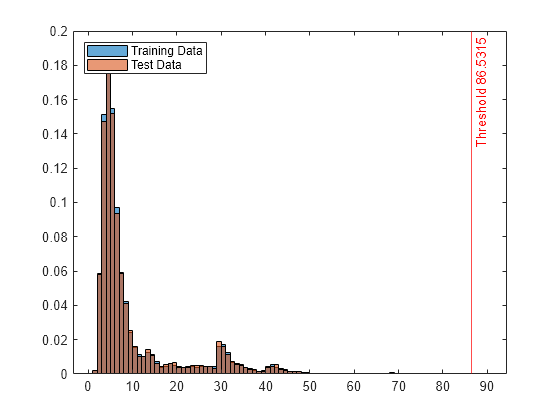
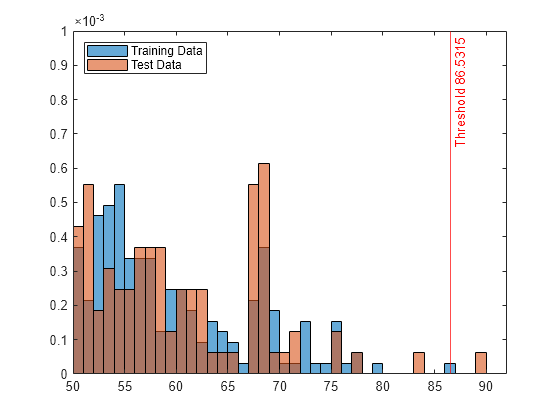
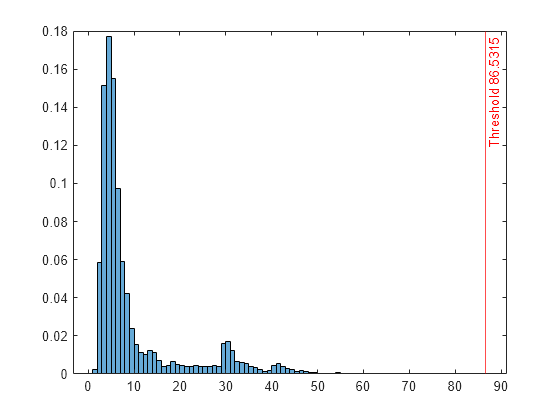
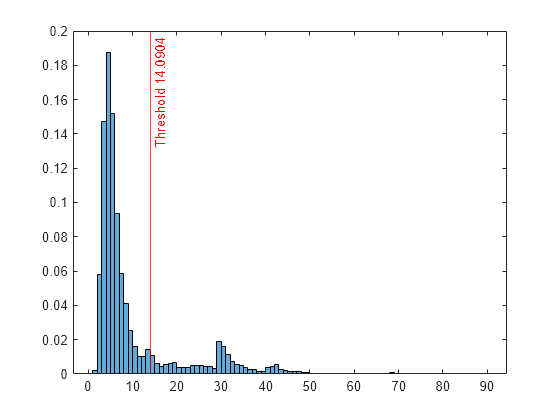
tf = isanomaly(forest,Tbl)RobustRandomCutForest モデル オブジェクト forest を使用して table Tbl 内の異常を検出し、Tbl の対応する行で異常が検出された場合に要素が true になる logical 配列 tf を返します。関数 rrcforest に table を渡して forest を作成した場合、この構文を使用する必要があります。
tf = isanomaly(___,Name=Value)ScoreThreshold=0.5
例
入力引数
名前と値の引数
出力引数
詳細
アルゴリズム
isanomaly は、Tbl に含まれている NaN、'' (空の文字ベクトル)、"" (空の string)、<missing>、<undefined> の値と X に含まれている NaN 値を欠損値と見なします。
isanomaly は、欠損値を含む観測値を使用して、それらの観測値が有効な値をもつ変数の分岐を特定します。それらの観測値は、葉ノードではなく枝ノードに配置されることがあります。その場合、isanomaly は、それぞれの木について、枝ノードからルート ノードまで移動して比率 (Disp(x,C)/|C|) を計算します。すべての値が欠損値である観測値はルート ノードに配置されます。そのため、比率および異常スコアは、それぞれの木の学習観測値の数になります。これは、学習済みのロバスト ランダム カット フォレスト モデルで求められる異常スコアの最大値に相当します。それぞれの木の学習観測値の数は、名前と値の引数 NumObservationsPerLearner を使用して指定できます。
参照
[1] Guha, Sudipto, N. Mishra, G. Roy, and O. Schrijvers. "Robust Random Cut Forest Based Anomaly Detection on Streams," Proceedings of The 33rd International Conference on Machine Learning 48 (June 2016): 2712–21.
[2] Bartos, Matthew D., A. Mullapudi, and S. C. Troutman. "rrcf: Implementation of the Robust Random Cut Forest Algorithm for Anomaly Detection on Streams." Journal of Open Source Software 4, no. 35 (2019): 1336.
拡張機能
バージョン履歴
R2023a で導入