predict
ガウス過程回帰モデルの予測応答
構文
説明
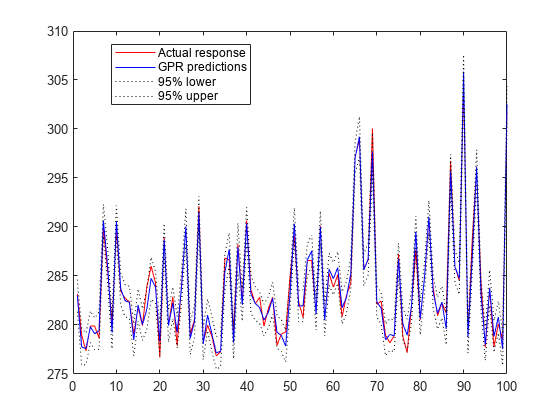
例
入力引数
名前と値の引数
出力引数
ヒント
fitrgpで名前と値のペアの引数PredictMethodを使用すると、GPR モデルを学習させるときの予測方法を選択できます。既定の予測方法は、n ≤ 10000 の場合は'exact'、それ以外の場合は'bcd'(ブロック座標降下) です。n は、学習データに含まれている観測値の数です。PredictMethodが'bcd'の場合、標準偏差ysdと予測区間yintの計算はサポートされません。gprMdlがCompactRegressionGPオブジェクトの場合、PredictMethodが'sr'または'fic'に等しいと、標準偏差ysdと予測区間yintを計算できません。PredictMethodが'sr'または'fic'に等しい場合にysdとyintを計算するには、完全な回帰オブジェクト (RegressionGP) を使用します。
代替方法
resubPredict を使用すると、学習データに含まれている観測値における学習済み GPR モデルの予測応答を計算できます。
Simulink ブロック
Simulink® にガウス過程回帰モデルの予測を統合するには、Statistics and Machine Learning Toolbox™ ライブラリにある RegressionGP Predict ブロックを使用するか、MATLAB® Function ブロックを関数 predict と共に使用します。例については、RegressionGP Predict ブロックの使用による応答の予測とMATLAB Function ブロックの使用によるクラス ラベルの予測を参照してください。
使用するアプローチを判断する際は、以下を考慮してください。
Statistics and Machine Learning Toolbox ライブラリ ブロックを使用する場合、固定小数点ツール (Fixed-Point Designer)を使用して浮動小数点モデルを固定小数点に変換できます。
MATLAB Function ブロックを関数
predictと共に使用する場合は、可変サイズの配列に対するサポートを有効にしなければなりません。MATLAB Function ブロックを使用する場合、予測の前処理や後処理のために、同じ MATLAB Function ブロック内で MATLAB 関数を使用することができます。
拡張機能
バージョン履歴
R2015b で導入参考
fitrgp | RegressionGP | CompactRegressionGP | compact | resubPredict | loss