predict
説明
例
学習済みの回帰ニューラル ネットワーク モデルを使用してテスト セットの応答値を予測します。
patients データ セットを読み込みます。データ セットから table を作成します。各行が 1 人の患者に対応し、各列が診断の変数に対応します。変数 Systolic を応答変数として使用し、残りの変数を予測子として使用します。
load patients
tbl = table(Diastolic,Height,Smoker,Weight,Systolic);非層化ホールドアウト分割を使用して、データを学習セット tblTrain とテスト セット tblTest に分割します。観測値の約 30% がテスト データ セット用に予約され、残りの観測値が学習データ セットに使用されます。
rng("default") % For reproducibility of the partition c = cvpartition(size(tbl,1),"Holdout",0.30); trainingIndices = training(c); testIndices = test(c); tblTrain = tbl(trainingIndices,:); tblTest = tbl(testIndices,:);
学習セットを使用して回帰ニューラル ネットワーク モデルに学習させます。tblTrain の列 Systolic を応答変数として指定します。数値予測子を標準化するための指定を行い、反復制限を 50 に設定します。既定では、ニューラル ネットワーク モデルには、最終全結合層を除き、出力数が 10 の全結合層が 1 つあります。
Mdl = fitrnet(tblTrain,"Systolic", ... "Standardize",true,"IterationLimit",50);
テスト セットの患者の収縮期血圧レベルを予測します。
predictedY = predict(Mdl,tblTest);
基準線を示した散布図を使用して結果を可視化します。予測値を縦軸に、実際の応答値を横軸に沿ってプロットします。基準線上にある点は予測が正しいことを示します。
plot(tblTest.Systolic,predictedY,".") hold on plot(tblTest.Systolic,tblTest.Systolic) hold off xlabel("True Systolic Blood Pressure Levels") ylabel("Predicted Systolic Blood Pressure Levels")

多くの点が基準線から離れた位置にあるため、サイズ 10 の全結合層をもつ既定のニューラル ネットワーク モデルは、収縮期血圧レベルの最適な予測子ではないようです。
テスト セットの損失と予測を比較することにより、特徴選択を実行します。すべての予測子を使用して学習させた回帰ニューラル ネットワーク モデルのテスト セット メトリクスを予測子のサブセットのみを使用して学習させたモデルのテスト セット メトリクスと比較します。
標本ファイル fisheriris.csv を読み込みます。これには、アヤメについてのがく片の長さ、がく片の幅、花弁の長さ、花弁の幅、種の種類などのデータが格納されています。ファイルを table に読み込みます。
fishertable = readtable('fisheriris.csv');非層化ホールドアウト分割を使用して、データを学習セット trainTbl とテスト セット testTbl に分割します。観測値の約 30% がテスト データ セット用に予約され、残りの観測値が学習データ セットに使用されます。
rng("default") c = cvpartition(size(fishertable,1),"Holdout",0.3); trainTbl = fishertable(training(c),:); testTbl = fishertable(test(c),:);
学習セット内のすべての予測子を使用して 1 つの回帰ニューラル ネットワーク モデルに学習させ、PetalWidth を除くすべての予測子を使用してもう 1 つのモデルに学習させます。両方のモデルについて、PetalLength を応答変数として指定し、予測子を標準化します。
allMdl = fitrnet(trainTbl,"PetalLength","Standardize",true); subsetMdl = fitrnet(trainTbl,"PetalLength ~ SepalLength + SepalWidth + Species", ... "Standardize",true);
2 つのモデルのテスト セットの平均二乗誤差 (MSE) を比較します。MSE の値が小さいほど、パフォーマンスが優れていることを示します。
allMSE = loss(allMdl,testTbl)
allMSE = 0.0834
subsetMSE = loss(subsetMdl,testTbl)
subsetMSE = 0.0884
各モデルについて、テスト セットの予測される花弁の長さと実際の花弁の長さを比較します。予測される花弁の長さを縦軸に、実際の花弁の長さを横軸に沿ってプロットします。基準線上にある点は予測が正しいことを示します。
tiledlayout(2,1) % Top axes ax1 = nexttile; allPredictedY = predict(allMdl,testTbl); plot(ax1,testTbl.PetalLength,allPredictedY,".") hold on plot(ax1,testTbl.PetalLength,testTbl.PetalLength) hold off xlabel(ax1,"True Petal Length") ylabel(ax1,"Predicted Petal Length") title(ax1,"All Predictors") % Bottom axes ax2 = nexttile; subsetPredictedY = predict(subsetMdl,testTbl); plot(ax2,testTbl.PetalLength,subsetPredictedY,".") hold on plot(ax2,testTbl.PetalLength,testTbl.PetalLength) hold off xlabel(ax2,"True Petal Length") ylabel(ax2,"Predicted Petal Length") title(ax2,"Subset of Predictors")
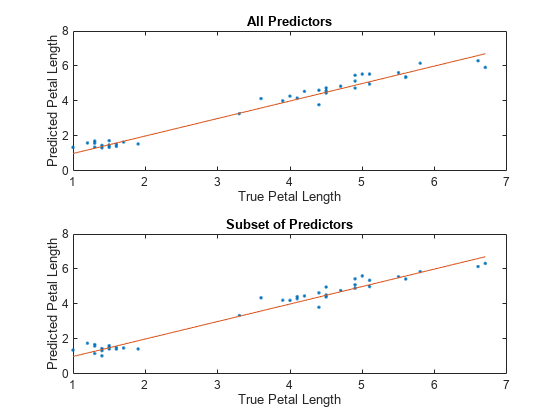
予測が基準線の近くに分布しており、両方のモデルが適切に機能しているようなので、PetalWidth を除くすべての予測子を使用して学習させたモデルを使用することを検討します。
単一の観測値の応答値を予測するために回帰ニューラル ネットワーク モデルの層がどのように連携するかを調べます。
標本ファイル fisheriris.csv を読み込みます。これには、アヤメについてのがく片の長さ、がく片の幅、花弁の長さ、花弁の幅、種の種類などのデータが格納されています。ファイルを table に読み込み、table の最初の数行を表示します。
fishertable = readtable('fisheriris.csv');
head(fishertable) SepalLength SepalWidth PetalLength PetalWidth Species
___________ __________ ___________ __________ __________
5.1 3.5 1.4 0.2 {'setosa'}
4.9 3 1.4 0.2 {'setosa'}
4.7 3.2 1.3 0.2 {'setosa'}
4.6 3.1 1.5 0.2 {'setosa'}
5 3.6 1.4 0.2 {'setosa'}
5.4 3.9 1.7 0.4 {'setosa'}
4.6 3.4 1.4 0.3 {'setosa'}
5 3.4 1.5 0.2 {'setosa'}
データ セットを使用して回帰ニューラル ネットワーク モデルに学習させます。変数 PetalLength を応答として指定し、他の数値変数を予測子として使用します。
Mdl = fitrnet(fishertable,"PetalLength ~ SepalLength + SepalWidth + PetalWidth");データ セットから 15 番目の観測値を選択します。ニューラル ネットワークの層が観測値をどのように扱い、予測応答値 newPointResponse をどのように返すかを調べます。
newPoint = Mdl.X{15,:}newPoint = 1×3
5.8000 4.0000 0.2000
firstFCStep = (Mdl.LayerWeights{1})*newPoint' + Mdl.LayerBiases{1};
reluStep = max(firstFCStep,0);
finalFCStep = (Mdl.LayerWeights{end})*reluStep + Mdl.LayerBiases{end};
newPointResponse = finalFCStepnewPointResponse = 1.6716
オブジェクト関数 predict で返される予測と一致することを確認します。
predictedY = predict(Mdl,newPoint)
predictedY = 1.6716
isequal(newPointResponse,predictedY)
ans = logical
1
2 つの結果は一致しています。
R2024b 以降
複数の応答変数をもつ回帰ニューラル ネットワークを作成します。
carbig データ セットを読み込みます。このデータ セットには、1970 年代と 1980 年代初期に製造された自動車の測定値が格納されています。Displacement や Horsepower などの予測子変数と応答変数の Acceleration および MPG が格納された table を作成します。table の最初の 8 行を表示します。
load carbig cars = table(Displacement,Horsepower,Model_Year, ... Origin,Weight,Acceleration,MPG); head(cars)
Displacement Horsepower Model_Year Origin Weight Acceleration MPG
____________ __________ __________ _______ ______ ____________ ___
307 130 70 USA 3504 12 18
350 165 70 USA 3693 11.5 15
318 150 70 USA 3436 11 18
304 150 70 USA 3433 12 16
302 140 70 USA 3449 10.5 17
429 198 70 USA 4341 10 15
454 220 70 USA 4354 9 14
440 215 70 USA 4312 8.5 14
cars から table に欠損値がある行を削除します。
cars = rmmissing(cars);
米国製かどうかに基づいて、自動車を分類します。
cars.Origin = categorical(cellstr(cars.Origin)); cars.Origin = mergecats(cars.Origin,["France","Japan",... "Germany","Sweden","Italy","England"],"NotUSA");
データを学習セットとテスト セットに分割します。観測値の約 85% をニューラル ネットワーク モデルの学習に使用し、観測値の約 15% を学習済みモデルの新しいデータでの性能のテストに使用します。cvpartition を使用してデータを分割します。
rng("default") % For reproducibility c = cvpartition(height(cars),"Holdout",0.15); carsTrain = cars(training(c),:); carsTest = cars(test(c),:);
学習データ carsTrain を fitrnet 関数に渡して、多重応答ニューラル ネットワーク回帰モデルに学習させます。より良い結果を得るために、予測子データを標準化するように指定します。
Mdl = fitrnet(carsTrain,["Acceleration","MPG"], ... Standardize=true)
Mdl =
RegressionNeuralNetwork
PredictorNames: {'Displacement' 'Horsepower' 'Model_Year' 'Origin' 'Weight'}
ResponseName: {'Acceleration' 'MPG'}
CategoricalPredictors: 4
ResponseTransform: 'none'
NumObservations: 334
LayerSizes: 10
Activations: 'relu'
OutputLayerActivation: 'none'
Solver: 'LBFGS'
ConvergenceInfo: [1×1 struct]
TrainingHistory: [1000×7 table]
Properties, Methods
Mdl は学習させた RegressionNeuralNetwork モデルです。ドット表記を使用して Mdl のプロパティにアクセスできます。たとえば、Mdl.ConvergenceInfo と指定すると、モデルの収束についての詳細情報を取得できます。
検定の平均二乗誤差 (MSE) を計算して、テスト セットで回帰モデルの性能を評価します。MSE の値が小さいほど、パフォーマンスが優れていることを示します。名前と値の引数 OutputType を "per-response" に設定して、各応答変数の損失を個別に返します。
testMSE = loss(Mdl,carsTest,["Acceleration","MPG"], ... OutputType="per-response")
testMSE = 1×2
1.5341 4.8245
テスト セット内の観測値に対する応答値を予測します。予測された応答値を table として返します。
predictedY = predict(Mdl,carsTest,OutputType="table")predictedY=58×2 table
Acceleration MPG
____________ ______
9.3612 13.567
15.655 21.406
17.921 17.851
11.139 13.433
12.696 10.32
16.498 17.977
16.227 22.016
12.165 12.926
12.691 12.072
12.424 14.481
16.974 22.152
15.504 24.955
11.068 13.874
11.978 12.664
14.926 10.134
15.638 24.839
⋮
入力引数
学習させた回帰ニューラル ネットワーク。fitrnet によって返される RegressionNeuralNetwork モデル オブジェクト、または compact によって返される CompactRegressionNeuralNetwork モデル オブジェクトとして指定します。
応答の生成に使用する予測子データ。数値行列または table を指定します。
既定では、X の各行は 1 つの観測値に対応し、各列は 1 つの変数に対応します。
数値行列の場合
Xの列に含まれている変数の順序は、Mdlに学習させた予測子変数の順序と同じでなければなりません。table (たとえば
Tbl) を使用してMdlに学習をさせる場合、Tblに含まれている予測子変数が数値のみであれば、Xを数値行列にすることができます。学習時にTbl内の数値予測子をカテゴリカルとして扱うには、fitrnetの名前と値の引数CategoricalPredictorsを使用してカテゴリカル予測子を指定します。Tblに種類の異なる予測子変数 (数値および categorical データ型など) が混在し、Xが数値行列である場合、predictでエラーがスローされます。
table の場合
predictは、文字ベクトルの cell 配列ではない cell 配列や複数列の変数をサポートしません。table (たとえば
Tbl) を使用してMdlに学習をさせた場合、X内のすべての予測子変数は変数名およびデータ型が、Mdlに学習させた (Mdl.PredictorNamesに格納されている) 変数と同じでなければなりません。ただし、Xの列の順序がTblの列の順序に対応する必要はありません。また、TblとXに追加の変数 (応答変数や観測値の重みなど) を含めることができますが、predictはこれらを無視します。数値行列を使用して
Mdlに学習をさせる場合、Mdl.PredictorNames内の予測子名とX内の対応する予測子変数名が同じでなければなりません。学習時に予測子の名前を指定するには、fitrnetの名前と値の引数PredictorNamesを使用します。X内の予測子変数はすべて数値ベクトルでなければなりません。Xに追加の変数 (応答変数や観測値の重みなど) を含めることができますが、predictはこれらを無視します。
Mdl に学習させるときに fitrnet で Standardize=true を設定した場合、予測子データの数値列が対応する平均 (Mdl.Mu) および標準偏差 (Mdl.Sigma) を使用して標準化されます。
メモ
観測値が列に対応するように予測子行列を配置して ObservationsIn="columns" を指定すると、計算時間が大幅に短縮される可能性があります。table の予測子データや多重応答回帰に対して ObservationsIn="columns" を指定することはできません。
データ型: single | double | table
名前と値の引数
オプションの引数のペアを Name1=Value1,...,NameN=ValueN として指定します。ここで、Name は引数名で、Value は対応する値です。名前と値の引数は他の引数の後に指定しなければなりませんが、ペアの順序は重要ではありません。
例: predict(Mdl,X,ObservationsIn="columns") は、予測子データの列が観測値に対応することを示します。
予測子データにおける観測値の次元。"rows" または "columns" として指定します。
メモ
観測値が列に対応するように予測子行列を配置して ObservationsIn="columns" を指定すると、計算時間が大幅に短縮される可能性があります。table の予測子データや多重応答回帰に対して ObservationsIn="columns" を指定することはできません。
データ型: char | string
R2023b 以降
予測子に欠損値がある観測値に使用する予測した応答値。"median"、"mean"、または数値スカラーとして指定します。
| 値 | 説明 |
|---|---|
"median" | predict は、予測子に欠損値がある観測値について予測した応答値として、学習データ内の観測された応答値の中央値を使用します。 |
"mean" | predict は、予測子に欠損値がある観測値について予測した応答値として、学習データ内の観測された応答値の平均値を使用します。 |
| 数値スカラー | predict は、予測子に欠損値がある観測値について予測した応答値として、この値を使用します。 |
例: PredictionForMissingValue="mean"
例: PredictionForMissingValue=NaN
データ型: single | double | char | string
出力引数
代替機能
Simulink ブロック
Simulink® にニューラル ネットワーク回帰モデルの予測を統合するには、Statistics and Machine Learning Toolbox™ ライブラリにある RegressionNeuralNetwork Predict ブロックを使用するか、MATLAB® Function ブロックを関数 predict と共に使用します。例については、RegressionNeuralNetwork Predict ブロックの使用による応答の予測とMATLAB Function ブロックの使用によるクラス ラベルの予測を参照してください。
使用するアプローチを判断する際は、以下を考慮してください。
Statistics and Machine Learning Toolbox ライブラリ ブロックを使用する場合、固定小数点ツール (Fixed-Point Designer)を使用して浮動小数点モデルを固定小数点に変換できます。
MATLAB Function ブロックを関数
predictと共に使用する場合は、可変サイズの配列に対するサポートを有効にしなければなりません。MATLAB Function ブロックを使用する場合、予測の前処理や後処理のために、同じ MATLAB Function ブロック内で MATLAB 関数を使用することができます。
拡張機能
使用上の注意および制限:
saveLearnerForCoder、loadLearnerForCoderおよびcodegen(MATLAB Coder) を使用して、関数predictのコードを生成します。saveLearnerForCoderを使用して、学習済みモデルを保存します。loadLearnerForCoderを使用して保存済みモデルを読み込んで関数predictを呼び出す、エントリポイント関数を定義します。次に、codegenを使用して、エントリポイント関数のコードを生成します。多重応答回帰のコード生成はサポートされていません。predictの単精度の C/C++ コードを生成するには、loadLearnerForCoder関数を呼び出すときにDataType="single"を指定します。次の表は、
predictの引数に関する注意です。この表に含まれていない引数は、完全にサポートされています。引数 注意と制限 Mdlモデル オブジェクトの使用上の注意および制限については、
CompactRegressionNeuralNetworkオブジェクトのコード生成を参照してください。XXは、単精度または倍精度の行列か、数値変数、カテゴリカル変数、またはその両方を含む table でなければなりません。Xの行数、または観測値の数は可変サイズにすることができますが、Xの列数は固定でなければなりません。Xを table として指定する場合、モデルは table を使用して学習させたものでなければならず、かつ予測のためのエントリポイント関数で次を行う必要があります。データを配列として受け入れる。
データ入力の引数から table を作成し、その table 内で変数名を指定する。
table を
predictに渡す。
この table のワークフローの例については、table のデータを分類するためのコードの生成を参照してください。コード生成における table の使用の詳細については、table のコード生成 (MATLAB Coder)およびコード生成における table の制限事項 (MATLAB Coder)を参照してください。
名前と値の引数 名前と値の引数に含まれる名前はコンパイル時の定数でなければなりません。
ObservationsInの値はコンパイル時の定数でなければなりません。たとえば、生成されたコードでObservationsIn="columns"を使用するには、{coder.Constant("ObservationsIn"),coder.Constant("columns")}をcodegen(MATLAB Coder) の-argsの値に含めます。名前と値の引数
OutputTypeはコード生成でサポートされません。PredictionForMissingValueの値が非数値の場合、コンパイル時の定数でなければなりません。
詳細は、コード生成の紹介を参照してください。
この関数は、GPU 配列を完全にサポートします。詳細は、GPU での MATLAB 関数の実行 (Parallel Computing Toolbox)を参照してください。
バージョン履歴
R2021a で導入fitrnet 関数を使用して、複数の応答変数をもつニューラル ネットワーク回帰モデルを作成できます。応答変数の数にかかわらず、関数は RegressionNeuralNetwork オブジェクトを返します。predict オブジェクト関数を使用して新しいデータに対する応答を予測できます。
predict を呼び出すときに、名前と値の引数 OutputType を使用して、予測応答値を行列または table として返すかどうかを指定できます。
predict は GPU 配列を完全にサポートします。
R2023b 以降で損失を予測または計算する際、一部の回帰モデルでは、予測子に欠損値がある観測値について予測した応答値を指定できます。名前と値の引数 PredictionForMissingValue を指定して、予測値として数値スカラー、学習セットの中央値、または学習セットの平均値を使用します。損失を計算するときに、予測子に欠損値がある観測値を省略するように指定することもできます。
次の表は、名前と値の引数 PredictionForMissingValue をサポートするオブジェクト関数の一覧です。既定では、これらの関数は、予測子に欠損値がある観測値について予測した応答値として、学習セットの中央値を使用します。
| モデル タイプ | モデル オブジェクト | オブジェクト関数 |
|---|---|---|
| ガウス過程回帰 (GPR) モデル | RegressionGP, CompactRegressionGP | loss, predict, resubLoss, resubPredict |
RegressionPartitionedGP | kfoldLoss, kfoldPredict | |
| ガウス カーネル回帰モデル | RegressionKernel | loss, predict |
RegressionPartitionedKernel | kfoldLoss, kfoldPredict | |
| 線形回帰モデル | RegressionLinear | loss, predict |
RegressionPartitionedLinear | kfoldLoss, kfoldPredict | |
| ニューラル ネットワーク回帰モデル | RegressionNeuralNetwork, CompactRegressionNeuralNetwork | loss, predict, resubLoss, resubPredict |
RegressionPartitionedNeuralNetwork | kfoldLoss, kfoldPredict | |
| サポート ベクター マシン (SVM) 回帰モデル | RegressionSVM, CompactRegressionSVM | loss, predict, resubLoss, resubPredict |
RegressionPartitionedSVM | kfoldLoss, kfoldPredict |
以前のリリースでは、上記の回帰モデル関数 loss および predict は、予測子に欠損値がある観測値について予測した応答値として NaN を使用していました。予測子に欠損値がある観測値は、予測と損失の再代入 ("resub") と交差検証 ("kfold") の計算で省略されていました。
MATLAB Command
You clicked a link that corresponds to this MATLAB command:
Run the command by entering it in the MATLAB Command Window. Web browsers do not support MATLAB commands.
Web サイトの選択
Web サイトを選択すると、翻訳されたコンテンツにアクセスし、地域のイベントやサービスを確認できます。現在の位置情報に基づき、次のサイトの選択を推奨します:
また、以下のリストから Web サイトを選択することもできます。
最適なサイトパフォーマンスの取得方法
中国のサイト (中国語または英語) を選択することで、最適なサイトパフォーマンスが得られます。その他の国の MathWorks のサイトは、お客様の地域からのアクセスが最適化されていません。
南北アメリカ
- América Latina (Español)
- Canada (English)
- United States (English)
ヨーロッパ
- Belgium (English)
- Denmark (English)
- Deutschland (Deutsch)
- España (Español)
- Finland (English)
- France (Français)
- Ireland (English)
- Italia (Italiano)
- Luxembourg (English)
- Netherlands (English)
- Norway (English)
- Österreich (Deutsch)
- Portugal (English)
- Sweden (English)
- Switzerland
- United Kingdom (English)