loss
クラス: RegressionLinear
線形回帰モデルの回帰損失
説明
L = loss(Mdl,Tbl,ResponseVarName)Tbl 内の予測子データと Tbl.ResponseVarName 内の真の応答に対する MSE を返します。
L = loss(___,Name,Value)
入力引数
線形回帰モデル。RegressionLinear モデル オブジェクトとして指定します。RegressionLinear モデル オブジェクトは、fitrlinear を使用して作成できます。
予測子データ。n 行 p 列の完全な行列またはスパース行列を指定します。この X の配置は、各行が個々の観測値に、各列が個々の予測子変数に対応することを示しています。
メモ
観測値が列に対応するように予測子行列を配置して 'ObservationsIn','columns' を指定すると、計算時間が大幅に短縮される可能性があります。
Y の長さと X の観測値数は同じでなければなりません。
データ型: single | double
モデルを学習させるために使用する標本データ。table として指定します。Tbl の各行は 1 つの観測値に、各列は 1 つの予測子変数に対応します。必要に応じて、応答変数用および観測値の重み用の追加列を Tbl に含めることができます。Tbl には、Mdl を学習させるために使用したすべての予測子が含まれていなければなりません。文字ベクトルの cell 配列ではない cell 配列と複数列の変数は使用できません。
Mdl を学習させるために使用した応答変数が Tbl に含まれている場合、ResponseVarName または Y を指定する必要はありません。
テーブルに格納されている標本データを使用して Mdl の学習を行った場合、loss の入力データもテーブルに格納されていなければなりません。
応答変数の名前。Tbl 内の変数の名前で指定します。応答変数は、数値ベクトルでなければなりません。
ResponseVarName を指定する場合は、文字ベクトルまたは string スカラーとして指定しなければなりません。たとえば、応答変数が Tbl.Y として格納されている場合、ResponseVarName として 'Y' を指定します。それ以外の場合、Tbl の列は Tbl.Y を含めてすべて予測子として扱われます。
データ型: char | string
名前と値の引数
オプションの引数のペアを Name1=Value1,...,NameN=ValueN として指定します。ここで、Name は引数名で、Value は対応する値です。名前と値の引数は他の引数の後に指定しなければなりませんが、ペアの順序は重要ではありません。
R2021a より前では、名前と値をそれぞれコンマを使って区切り、Name を引用符で囲みます。
損失関数。'LossFun' と組み込み損失関数名または関数ハンドルから構成されるコンマ区切りのペアとして指定します。
次の表は、使用可能な損失関数の一覧です。対応する値を使用していずれかを指定します。また、この表では です。
β は p 個の係数のベクトルです。
x は p 個の予測子変数による観測値です。
b はスカラー バイアスです。
値 説明 'epsiloninsensitive'イプシロン不感応損失: 'mse'MSE: 'epsiloninsensitive'は SVM 学習器のみに適しています。関数ハンドル表記を使用して独自の関数を指定します。
n を
Xの観測値数とします。使用する関数のシグネチャは次のようになっていなければなりません。ここでlossvalue =lossfun(Y,Yhat,W)出力引数
lossvalueはスカラーです。関数名 (
lossfun) を選択します。Yは観測された応答の n 次元ベクトルです。lossには入力引数YでYを渡します。Yhatは予測された応答の n 次元ベクトルです。これはpredictの出力と同様です。Wは、観測値の重みの n 行 1 列の数値ベクトルです。
'LossFun',@を使用して独自の関数を指定します。lossfun
データ型: char | string | function_handle
R2023b 以降
予測子に欠損値がある観測値に使用する予測した応答値。"median"、"mean"、"omitted"、または数値スカラーとして指定します。
| 値 | 説明 |
|---|---|
"median" | loss は、予測子に欠損値がある観測値について予測した応答値として、学習データ内の観測された応答値の中央値を使用します。 |
"mean" | loss は、予測子に欠損値がある観測値について予測した応答値として、学習データ内の観測された応答値の平均値を使用します。 |
"omitted" | loss は、予測子に欠損値がある観測値を損失の計算から除外します。 |
| 数値スカラー | loss は、予測子に欠損値がある観測値について予測した応答値として、この値を使用します。 |
観測された応答値または観測値の重みが観測値にない場合、その観測値は loss による損失の計算に使用されません。
例: PredictionForMissingValue="omitted"
データ型: single | double | char | string
予測子データにおける観測値の次元。'rows' または 'columns' として指定します。
メモ
観測値が列に対応するように予測子行列を配置して 'ObservationsIn','columns' を指定すると、計算時間が大幅に短縮される可能性があります。table の予測子データに対して 'ObservationsIn','columns' を指定することはできません。
データ型: char | string
観測値の重み。'Weights' と数値ベクトル、または Tbl に含まれる変数の名前から構成されるコンマ区切りのペアとして指定します。
Weightsとして数値ベクトルを指定する場合、WeightsのサイズはXまたはTbl内の観測値の個数と等しくなければなりません。WeightsとしてTbl内の変数名を指定する場合、名前は文字ベクトルまたは string スカラーでなければなりません。たとえば、重みがTbl.Wとして格納されている場合、Weightsとして'W'を指定します。それ以外の場合、Tblの列はTbl.Wを含めてすべて予測子として扱われます。
重みを指定した場合、loss は加重回帰損失を計算し、合計が 1 になるように Weights を正規化します。
データ型: double | single
出力引数
メモ
Mdl.FittedLoss が 'mse' の場合、目的関数の損失項は MSE の半分になります。既定の設定では、loss は MSE を返します。したがって、loss を使用して再代入 (学習) 誤差をチェックした場合、fitrlinear によって返される最適化結果と MSE が異なります。
例
次のモデルにより、10000 個の観測値をシミュレートします。
は、10% の要素が非ゼロ標準正規である 10000 行 1000 列のスパース行列です。
e は、平均が 0、標準偏差が 0.3 のランダムな正規誤差です。
rng(1) % For reproducibility
n = 1e4;
d = 1e3;
nz = 0.1;
X = sprandn(n,d,nz);
Y = X(:,100) + 2*X(:,200) + 0.3*randn(n,1);線形回帰モデルに学習をさせます。観測値の 30% をホールドアウト標本として予約します。
CVMdl = fitrlinear(X,Y,'Holdout',0.3);
Mdl = CVMdl.Trained{1}Mdl =
RegressionLinear
ResponseName: 'Y'
ResponseTransform: 'none'
Beta: [1000×1 double]
Bias: -0.0066
Lambda: 1.4286e-04
Learner: 'svm'
Properties, Methods
CVMdl は RegressionPartitionedLinear モデルです。これには Trained プロパティが含まれています。これは 1 行 1 列の cell 配列で、学習セットにより学習させた RegressionLinear モデルが格納されています。
学習データとテスト データを分割の定義から抽出します。
trainIdx = training(CVMdl.Partition); testIdx = test(CVMdl.Partition);
学習標本とテスト標本の MSE を推定します。
mseTrain = loss(Mdl,X(trainIdx,:),Y(trainIdx))
mseTrain = 0.1496
mseTest = loss(Mdl,X(testIdx,:),Y(testIdx))
mseTest = 0.1798
Mdl 内の正則化強度は 1 つなので、mseTrain と mseTest は数値スカラーになります。
次のモデルにより、10000 個の観測値をシミュレートします。
は、10% の要素が非ゼロ標準正規である 10000 行 1000 列のスパース行列です。
e は、平均が 0、標準偏差が 0.3 のランダムな正規誤差です。
rng(1) % For reproducibility n = 1e4; d = 1e3; nz = 0.1; X = sprandn(n,d,nz); Y = X(:,100) + 2*X(:,200) + 0.3*randn(n,1); X = X'; % Put observations in columns for faster training
線形回帰モデルに学習をさせます。観測値の 30% をホールドアウト標本として予約します。
CVMdl = fitrlinear(X,Y,'Holdout',0.3,'ObservationsIn','columns'); Mdl = CVMdl.Trained{1}
Mdl =
RegressionLinear
ResponseName: 'Y'
ResponseTransform: 'none'
Beta: [1000×1 double]
Bias: -0.0066
Lambda: 1.4286e-04
Learner: 'svm'
Properties, Methods
CVMdl は RegressionPartitionedLinear モデルです。これには Trained プロパティが含まれています。これは 1 行 1 列の cell 配列で、学習セットにより学習させた RegressionLinear モデルが格納されています。
学習データとテスト データを分割の定義から抽出します。
trainIdx = training(CVMdl.Partition); testIdx = test(CVMdl.Partition);
次の Huber 損失 ( = 1) を評価する無名関数を作成します。
ここで
は観測値 j の残差です。カスタム損失関数は特定の形式で記述しなければなりません。カスタム損失関数の記述に関するルールについては、名前と値のペアの引数 'LossFun' を参照してください。
huberloss = @(Y,Yhat,W)sum(W.*((0.5*(abs(Y-Yhat)<=1).*(Y-Yhat).^2) + ...
((abs(Y-Yhat)>1).*abs(Y-Yhat)-0.5)))/sum(W);Huber 損失関数を使用して、学習セットおよびテスト セットの回帰損失を推定します。
eTrain = loss(Mdl,X(:,trainIdx),Y(trainIdx),'LossFun',huberloss,... 'ObservationsIn','columns')
eTrain = -0.4186
eTest = loss(Mdl,X(:,testIdx),Y(testIdx),'LossFun',huberloss,... 'ObservationsIn','columns')
eTest = -0.4010
次のモデルにより、10000 個の観測値をシミュレートします。
は、10% の要素が非ゼロ標準正規である 10000 行 1000 列のスパース行列です。
e は、平均が 0、標準偏差が 0.3 のランダムな正規誤差です。
rng(1) % For reproducibility
n = 1e4;
d = 1e3;
nz = 0.1;
X = sprandn(n,d,nz);
Y = X(:,100) + 2*X(:,200) + 0.3*randn(n,1);~ の範囲で対数間隔で配置された 15 個の正則化強度を作成します。
Lambda = logspace(-4,-1,15);
データの 30% をテスト用にホールドアウトします。テスト標本のインデックスを識別します。
cvp = cvpartition(numel(Y),'Holdout',0.30);
idxTest = test(cvp);Lambda 内の強度による LASSO ペナルティを使用して、線形回帰モデルに学習をさせます。正則化強度、目的関数を最適化するための SpaRSA の使用、およびデータ分割を指定します。実行速度を向上させるため、予測子データを転置し、観測値が列単位であることを指定します。
X = X'; CVMdl = fitrlinear(X,Y,'ObservationsIn','columns','Lambda',Lambda,... 'Solver','sparsa','Regularization','lasso','CVPartition',cvp); Mdl1 = CVMdl.Trained{1}; numel(Mdl1.Lambda)
ans = 15
Mdl1 は RegressionLinear モデルです。Lambda は正則化強度の 15 次元ベクトルなので、Mdl1 はそれぞれが正則化強度に対応する 15 個の学習済みモデルであると考えることができます。
各正則化モデルについてテスト標本の平均二乗誤差を推定します。
mse = loss(Mdl1,X(:,idxTest),Y(idxTest),'ObservationsIn','columns');
Lambda の値が大きくなると、予測子変数がスパースになります。これは回帰モデルの品質として優れています。データ セット全体と、以前に使用した、データ分割指定以外のすべてのオプションを使用して、モデルに再学習をさせます。モデルごとに非ゼロの係数を特定します。
Mdl = fitrlinear(X,Y,'ObservationsIn','columns','Lambda',Lambda,... 'Solver','sparsa','Regularization','lasso'); numNZCoeff = sum(Mdl.Beta~=0);
同じ図に、各正則化強度についての MSE と非ゼロ係数の頻度をプロットします。すべての変数を対数スケールでプロットします。
figure; [h,hL1,hL2] = plotyy(log10(Lambda),log10(mse),... log10(Lambda),log10(numNZCoeff)); hL1.Marker = 'o'; hL2.Marker = 'o'; ylabel(h(1),'log_{10} MSE') ylabel(h(2),'log_{10} nonzero-coefficient frequency') xlabel('log_{10} Lambda') hold off
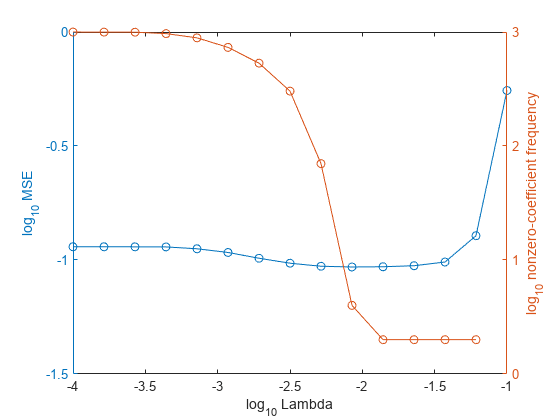
最小分類誤差と予測子変数のスパース性のバランスがとれている Lambda のインデックス (Lambda(11) など) を選択します。
idx = 11; MdlFinal = selectModels(Mdl,idx);
MdlFinal は、正則化強度として Lambda(11) を使用する学習済みの RegressionLinear モデル オブジェクトです。
拡張機能
この関数は、GPU 配列を完全にサポートします。詳細は、GPU での MATLAB 関数の実行 (Parallel Computing Toolbox)を参照してください。
バージョン履歴
R2016a で導入loss は GPU 配列を完全にサポートします。
R2023b 以降で損失を予測または計算する際、一部の回帰モデルでは、予測子に欠損値がある観測値について予測した応答値を指定できます。名前と値の引数 PredictionForMissingValue を指定して、予測値として数値スカラー、学習セットの中央値、または学習セットの平均値を使用します。損失を計算するときに、予測子に欠損値がある観測値を省略するように指定することもできます。
次の表は、名前と値の引数 PredictionForMissingValue をサポートするオブジェクト関数の一覧です。既定では、これらの関数は、予測子に欠損値がある観測値について予測した応答値として、学習セットの中央値を使用します。
| モデル タイプ | モデル オブジェクト | オブジェクト関数 |
|---|---|---|
| ガウス過程回帰 (GPR) モデル | RegressionGP, CompactRegressionGP | loss, predict, resubLoss, resubPredict |
RegressionPartitionedGP | kfoldLoss, kfoldPredict | |
| ガウス カーネル回帰モデル | RegressionKernel | loss, predict |
RegressionPartitionedKernel | kfoldLoss, kfoldPredict | |
| 線形回帰モデル | RegressionLinear | loss, predict |
RegressionPartitionedLinear | kfoldLoss, kfoldPredict | |
| ニューラル ネットワーク回帰モデル | RegressionNeuralNetwork, CompactRegressionNeuralNetwork | loss, predict, resubLoss, resubPredict |
RegressionPartitionedNeuralNetwork | kfoldLoss, kfoldPredict | |
| サポート ベクター マシン (SVM) 回帰モデル | RegressionSVM, CompactRegressionSVM | loss, predict, resubLoss, resubPredict |
RegressionPartitionedSVM | kfoldLoss, kfoldPredict |
以前のリリースでは、上記の回帰モデル関数 loss および predict は、予測子に欠損値がある観測値について予測した応答値として NaN を使用していました。予測子に欠損値がある観測値は、予測と損失の再代入 ("resub") と交差検証 ("kfold") の計算で省略されていました。
関数 loss で加重平均回帰損失を計算する際に、予測が NaN の観測値が省略されなくなりました。そのため、予測子データ X または Tbl 内の予測子変数に欠損値がある場合に loss で NaN が返されることがあります。ほとんどの場合、テスト セットの観測値に予測子の欠損がなければ、関数 loss で NaN が返されることはありません。
この変更により、fitrauto を使用する場合の回帰モデルの自動選択が改善されます。この変更の前は、NaN 以外の予測子が少ないモデルが選択される (新しいデータの応答の予測に最適であると予測される) ことがありました。
コードの loss で NaN が返される場合、このような結果にならないようにコードを更新できます。rmmissing または fillmissing を使用して、欠損値を削除するか置き換えます。
次の表に、オブジェクト関数 loss で NaN が返される可能性がある回帰モデルを示します。詳細については、それぞれの関数 loss の「互換性の考慮事項」を参照してください。
| モデル タイプ | 完全またはコンパクトなモデル オブジェクト | オブジェクト関数 loss |
|---|---|---|
| ガウス過程回帰 (GPR) モデル | RegressionGP, CompactRegressionGP | loss |
| ガウス カーネル回帰モデル | RegressionKernel | loss |
| 線形回帰モデル | RegressionLinear | loss |
| ニューラル ネットワーク回帰モデル | RegressionNeuralNetwork, CompactRegressionNeuralNetwork | loss |
| サポート ベクター マシン (SVM) 回帰モデル | RegressionSVM, CompactRegressionSVM | loss |
MATLAB Command
You clicked a link that corresponds to this MATLAB command:
Run the command by entering it in the MATLAB Command Window. Web browsers do not support MATLAB commands.
Web サイトの選択
Web サイトを選択すると、翻訳されたコンテンツにアクセスし、地域のイベントやサービスを確認できます。現在の位置情報に基づき、次のサイトの選択を推奨します:
また、以下のリストから Web サイトを選択することもできます。
最適なサイトパフォーマンスの取得方法
中国のサイト (中国語または英語) を選択することで、最適なサイトパフォーマンスが得られます。その他の国の MathWorks のサイトは、お客様の地域からのアクセスが最適化されていません。
南北アメリカ
- América Latina (Español)
- Canada (English)
- United States (English)
ヨーロッパ
- Belgium (English)
- Denmark (English)
- Deutschland (Deutsch)
- España (Español)
- Finland (English)
- France (Français)
- Ireland (English)
- Italia (Italiano)
- Luxembourg (English)
- Netherlands (English)
- Norway (English)
- Österreich (Deutsch)
- Portugal (English)
- Sweden (English)
- Switzerland
- United Kingdom (English)