loss
アンサンブル分類モデルの分類損失
説明
L = loss(___,Name=Value)
メモ
予測子データ X または tbl 内の予測子変数に欠損値がある場合、関数 loss で NaN が返されることがあります。詳細については、欠損値がある予測子データに対して loss で NaN が返されることがあるを参照してください。
例
分類誤差の推定
フィッシャーのアヤメのデータ セットを読み込みます。
load fisheririsAdaBoostM2 を使用して、100 本の決定木によるアンサンブル分類に学習をさせます。弱学習器として木の切り株を指定します。
t = templateTree(MaxNumSplits=1);
ens = fitcensemble(meas,species,Method="AdaBoostM2",Learners=t);学習観測値を使用してモデルの分類誤差を推定します。
L = loss(ens,meas,species)
L = 0.0333
または、ens がコンパクトでない場合は、ens を resubLoss に渡すことにより学習標本の分類誤差を推定できます。
ブースティング木のアンサンブルの性能評価
ブースティング木のアンサンブルを作成し、各予測子の重要度を検査します。テスト データを使用して、アンサンブルの分類精度を評価します。
不整脈データ セットを読み込みます。データのクラス表現を判別します。
load arrhythmia
Y = categorical(Y);
tabulate(Y) Value Count Percent
1 245 54.20%
2 44 9.73%
3 15 3.32%
4 15 3.32%
5 13 2.88%
6 25 5.53%
7 3 0.66%
8 2 0.44%
9 9 1.99%
10 50 11.06%
14 4 0.88%
15 5 1.11%
16 22 4.87%
データ セットには 16 個のクラスが含まれていますが、すべてのクラスは表現されていません (たとえば、クラス 13)。ほとんどの観測値は不整脈がないものとして分類されています (クラス 1)。このデータ セットは非常に離散的であり、クラスが不均衡です。
不整脈があるすべての観測値 (クラス 2 ~ 15) を 1 つのクラスに結合します。不整脈の状況が不明である観測値 (クラス 16) をデータ セットから削除します。
idx = (Y ~= "16"); Y = Y(idx); X = X(idx,:); Y(Y ~= "1") = "WithArrhythmia"; Y(Y == "1") = "NoArrhythmia"; Y = removecats(Y);
データを学習セットとテスト セットに均等に分割します。
rng("default") % For reproducibility cvp = cvpartition(Y,"Holdout",0.5); idxTrain = training(cvp); idxTest = test(cvp);
cvp は、学習セットとテスト セットを指定する交差検証分割オブジェクトです。
AdaBoostM1 を使用して 100 本のブースティング分類木のアンサンブルに学習をさせます。弱学習器として木の切り株を使用するように指定します。また、欠損値がデータ セットに含まれているので、代理分岐を使用するように指定します。
t = templateTree("MaxNumSplits",1,"Surrogate","on"); numTrees = 100; mdl = fitcensemble(X(idxTrain,:),Y(idxTrain),"Method","AdaBoostM1", ... "NumLearningCycles",numTrees,"Learners",t);
mdl は学習させた ClassificationEnsemble モデルです。
各予測子について重要度を調べます。
predImportance = predictorImportance(mdl); bar(predImportance) title("Predictor Importance") xlabel("Predictor") ylabel("Importance Measure")
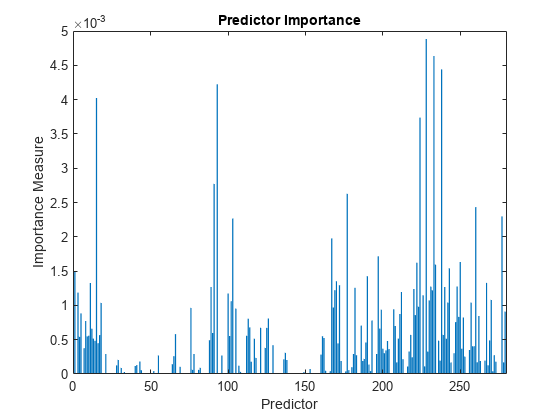
重要度が上位 10 番目までの予測子を識別します。
[~,idxSort] = sort(predImportance,"descend");
idx10 = idxSort(1:10)idx10 = 1×10
228 233 238 93 15 224 91 177 260 277
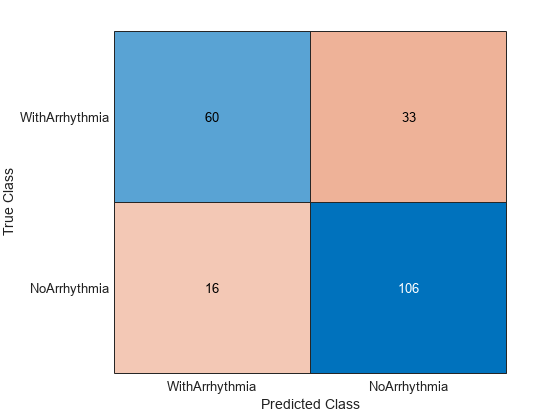
テスト セットの観測値を分類します。混同行列を使用して結果を表示します。青色の値は正しい分類を示し、赤色の値は誤分類された観測値を示します。
predictedValues = predict(mdl,X(idxTest,:)); confusionchart(Y(idxTest),predictedValues)
テスト セットでモデルの精度を計算します。
error = loss(mdl,X(idxTest,:),Y(idxTest), ... "LossFun","classiferror"); accuracy = 1 - error
accuracy = 0.7731
accuracy で、正しく分類された観測値の比率が推定されます。
入力引数
ens — アンサンブル分類モデル
ClassificationEnsemble モデル オブジェクト | CompactClassificationEnsemble モデル オブジェクト
完全なアンサンブル分類モデル。fitcensemble で学習させた ClassificationEnsemble モデル オブジェクト、または compact で作成した CompactClassificationEnsemble モデル オブジェクトとして指定します。
tbl — 標本データ
table
標本データ。テーブルとして指定します。tbl の各行は 1 つの観測値に、各列は 1 つの予測子変数に対応します。tbl には、モデルを学習させるために使用したすべての予測子が含まれていなければなりません。文字ベクトルの cell 配列ではない cell 配列と複数列の変数は使用できません。
table に格納されている標本データを使用して ens に学習させた場合、loss の入力データも table に含まれていなければなりません。
データ型: table
ResponseVarName — 応答変数名
name of variable in tbl
tbl応答変数の名前。tbl 内の変数の名前で指定します。ens を学習させるために使用した応答変数が tbl に含まれている場合、ResponseVarName を指定する必要はありません。
ResponseVarName を指定する場合は、文字ベクトルまたは string スカラーとして指定しなければなりません。たとえば、応答変数 Y が tbl.Y として格納されている場合、"Y" として指定します。それ以外の場合、tbl の列は Y を含めてすべて予測子として扱われます。
応答変数は、categorical 配列、文字配列、string 配列、logical ベクトル、数値ベクトル、または文字ベクトルの cell 配列でなければなりません。応答変数が文字配列の場合、各要素は配列の 1 つの行に対応しなければなりません。
データ型: char | string
Y — クラス ラベル
categorical 配列 | 文字配列 | string 配列 | logical ベクトル | 数値ベクトル | 文字ベクトルの cell 配列
クラス ラベル。categorical 配列、文字配列、string 配列、logical ベクトル、数値ベクトル、または文字ベクトルの cell 配列として指定します。Y のデータ型は tbl または X と同じでなければなりません。(string 配列は文字ベクトルの cell 配列として扱われます)。
Y の型は ens の学習に使用した分類と同じでなければならず、要素数は tbl または X の行数と等しくなければなりません。
データ型: categorical | char | string | logical | single | double | cell
名前と値の引数
オプションの引数のペアを Name1=Value1,...,NameN=ValueN として指定します。ここで Name は引数名、Value は対応する値です。名前と値の引数は他の引数の後ろにする必要がありますが、ペアの順序は関係ありません。
R2021a より前では、名前と値をそれぞれコンマを使って区切り、Name を引用符で囲みます。
例: loss(ens,X,LossFun="exponential",UseParallel=true) は、指数の損失関数を使用し、並列に実行するように指定します。
Learners — 弱学習器のインデックス
[1:ens.NumTrained] (既定値) | 正の整数のベクトル
loss で使用するアンサンブル内の弱学習器のインデックス。範囲 [1:ens.NumTrained] の正の整数のベクトルとして指定します。既定では、すべての学習器が使用されます。
例: Learners=[1 2 4]
データ型: single | double
LossFun — 損失関数
"classiferror" (既定値) | "binodeviance" | "classifcost" | "classiferror" | "exponential" | "hinge" | "logit" | "mincost" | "quadratic" | 関数ハンドル
損失関数。組み込みの損失関数名または関数ハンドルを指定します。
次の表に、組み込みの損失関数の値を示します。
| 値 | 説明 |
|---|---|
"binodeviance" | 二項分布からの逸脱度 |
"classifcost" | 観測誤分類コスト |
"classiferror" | 10 進数の誤分類率 |
"exponential" | 指数損失 |
"hinge" | ヒンジ損失 |
"logit" | ロジスティック損失 |
"mincost" | 最小予測誤分類コスト (事後確率である分類スコアの場合) |
"quadratic" | 二次損失 |
"mincost"は、事後確率である分類スコアに適しています。既定の設定では、バギング アンサンブルと部分空間アンサンブルは事後確率を返します (
ens.Methodが"Bag"または"Subspace")。アンサンブル法が
"AdaBoostM1"、"AdaBoostM2"、"GentleBoost"、または"LogitBoost"の場合に事後確率を分類スコアとして使用するには、次のように入力してダブルロジットのスコア変換を指定する必要があります。ens.ScoreTransform = "doublelogit";他のすべてのアンサンブル法の場合、事後確率は分類スコアとしてサポートされていません。
関数ハンドル表記を使用して独自の関数を指定できます。n は X 内の観測値の個数、K は異なるクラスの個数 (numel(ens.ClassNames)、ens は入力モデル) であるとします。使用する関数のシグネチャは次のようになっていなければなりません。
lossvalue = lossfun(C,S,W,Cost)出力引数
lossvalueはスカラーです。関数名 (
lossfun) を指定します。Cはn行K列の logical 行列であり、対応する観測値が属するクラスを各行が示します。列の順序はens.ClassNamesのクラスの順序に対応します。各行について観測値
pがクラスqに属する場合はC(p,q) = 1を設定することにより、Cを作成します。行pの他のすべての要素を0に設定します。Sは、分類スコアのn行K列の行列です。列の順序はens.ClassNamesのクラスの順序に対応します。Sは分類スコアの行列で、predictの出力と同様です。Wは、観測値の重みのn行 1 列の数値ベクトルです。Wを渡す場合、重みは合計が1になるように正規化されます。Costは、誤分類コストの、K 行K列の数値行列です。たとえば、Cost = ones(K) - eye(K)は、正しい分類のコストとして0を、誤分類のコストとして1を指定します。
損失関数の詳細については、分類損失を参照してください。
例: LossFun="binodeviance"
例: LossFun=@Lossfun
データ型: char | string | function_handle
Mode — 出力の集約レベル
"ensemble" (既定値) | "individual" | "cumulative"
出力の集約レベル。"ensemble"、"individual"、または "cumulative" として指定します。
| 値 | 説明 |
|---|---|
"ensemble" | 出力は、アンサンブル全体に対する損失を表すスカラー値になります。 |
"individual" | 出力は、学習させた学習器ごとに 1 つの要素をもつベクトルになります。 |
"cumulative" | 出力は、学習器の入力リストから学習器 1:J を使用して要素 J が取得されたベクトルになります。 |
例: Mode="individual"
データ型: char | string
UseParallel — 並列実行のフラグ
false または 0 (既定値) | true または 1
並列実行のフラグ。数値または logical の 1 (true) または 0 (false) として指定します。UseParallel=true を指定した場合、関数 loss は parfor を使用して for ループの反復を実行します。Parallel Computing Toolbox™ がある場合、ループが並列に実行されます。
例: UseParallel=true
データ型: logical
Weights — 観測値の重み
ones(size(X,1),1) (既定値) | 数値ベクトル | tbl 内の変数の名前
観測値の重み。数値ベクトルまたは tbl 内の変数の名前を指定します。重みを指定すると、loss は各クラスの観測値の重みの合計がそのクラスの事前確率になるように重みを正規化します。
数値ベクトルで Weights を指定する場合、Weights のサイズは X または tbl 内の観測値の数と等しくなければなりません。合計が各クラスの事前確率の値と等しくなるように Weights が正規化されます。
tbl 内の変数名を Weights として指定する場合、文字ベクトルまたは string スカラーとして指定しなければなりません。たとえば、重みが tbl.W として格納されている場合、Weights を "W" として指定します。それ以外の場合、tbl の列は tbl.W を含めすべて予測子として扱われます。
例: Weights="W"
データ型: single | double | char | string
出力引数
L — 分類損失
数値スカラー | 数値列ベクトル
分類損失。数値スカラーまたは数値列ベクトルとして返されます。
Modeが"ensemble"の場合、Lはアンサンブル全体に対する損失を表すスカラー値です。Modeが"individual"の場合、Lは学習させた学習器ごとに 1 つの要素をもつベクトルです。Modeが"cumulative"の場合、Lは学習器の入力リストから学習器1:Jを使用して要素Jが取得されたベクトルです。
関数 loss は、損失を計算するときに、ens の Prior プロパティに格納されている、学習に使用されたクラス確率に対して、ResponseVarName または Y のクラス確率を正規化します。
詳細
分類損失
"分類損失" 関数は分類モデルの予測誤差を評価します。複数のモデルで同じタイプの損失を比較した場合、損失が低い方が予測モデルとして優れていることになります。
以下のシナリオを考えます。
L は加重平均分類損失です。
n は標本サイズです。
バイナリ分類は以下です。
yj は観測されたクラス ラベルです。陰性クラスを示す -1 または陽性クラスを示す 1 (あるいは、
ClassNamesプロパティの最初のクラスを示す -1 または 2 番目のクラスを示す 1) を使用して符号化されます。f(Xj) は予測子データ X の観測値 (行) j に対する陽性クラスの分類スコアです。
mj = yjf(Xj) は、yj に対応するクラスに観測値 j を分類する分類スコアです。正の値の mj は正しい分類を示しており、平均損失に対する寄与は大きくありません。負の値の mj は正しくない分類を示しており、平均損失に大きく寄与します。
マルチクラス分類 (つまり、K ≥ 3) をサポートするアルゴリズムの場合、次のようになります。
yj* は、K - 1 個の 0 と、観測された真のクラス yj に対応する位置の 1 から構成されるベクトルです。たとえば、2 番目の観測値の真のクラスが 3 番目のクラスであり K = 4 の場合、y2* = [
0 0 1 0]′ になります。クラスの順序は入力モデルのClassNamesプロパティ内の順序に対応します。f(Xj) は予測子データ X の観測値 j に対するクラス スコアのベクトルで、長さは K です。スコアの順序は入力モデルの
ClassNamesプロパティ内のクラスの順序に対応します。mj = yj*′f(Xj).したがって mj は、観測された真のクラスについてモデルが予測するスカラー分類スコアです。
観測値 j の重みは wj です。観測値の重みは、その合計が
Priorプロパティに格納された対応するクラスの事前確率になるように正規化されます。そのため、次のようになります。
この状況では、名前と値の引数 LossFun を使用して指定できる、サポートされる損失関数は次の表のようになります。
| 損失関数 | LossFun の値 | 式 |
|---|---|---|
| 二項分布からの逸脱度 | "binodeviance" | |
| 観測誤分類コスト | "classifcost" | ここで、 はスコアが最大のクラスに対応するクラス ラベル、 は真のクラスが yj である場合に観測値をクラス に分類するユーザー指定のコストです。 |
| 10 進数の誤分類率 | "classiferror" | ここで、I{·} はインジケーター関数です。 |
| クロスエントロピー損失 | "crossentropy" |
加重クロスエントロピー損失は次となります。 ここで重み は、合計が 1 ではなく n になるように正規化されます。 |
| 指数損失 | "exponential" | |
| ヒンジ損失 | "hinge" | |
| ロジット損失 | "logit" | |
| 最小予測誤分類コスト | "mincost" |
重み付きの最小予測分類コストは、次の手順を観測値 j = 1、...、n について使用することにより計算されます。
最小予測誤分類コスト損失の加重平均は次となります。 |
| 二次損失 | "quadratic" |
既定のコスト行列 (正しい分類の場合の要素値は 0、誤った分類の場合の要素値は 1) を使用する場合、"classifcost"、"classiferror"、および "mincost" の損失の値は同じです。既定以外のコスト行列をもつモデルでは、ほとんどの場合は "classifcost" の損失と "mincost" の損失が等価になります。これらの損失が異なる値になる可能性があるのは、最大の事後確率をもつクラスへの予測と最小の予測コストをもつクラスへの予測が異なる場合です。"mincost" は分類スコアが事後確率の場合にしか適さないことに注意してください。
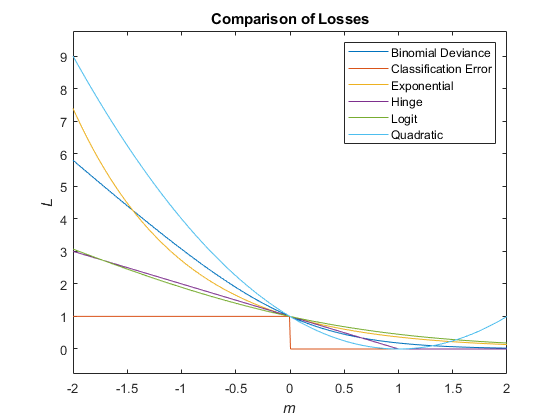
次の図では、1 つの観測値のスコア m に対する損失関数 ("classifcost"、"crossentropy"、および "mincost" を除く) を比較しています。いくつかの関数は、点 (0,1) を通過するように正規化されています。
拡張機能
tall 配列
メモリの許容量を超えるような多数の行を含む配列を計算します。
自動並列サポート
Parallel Computing Toolbox™ を使用して自動的に並列計算を実行することで、コードを高速化します。
並列実行するには、この関数を呼び出すときに名前と値の引数 UseParallel を true に設定します。
並列計算の全般的な情報については、自動並列サポートを使用した MATLAB 関数の実行 (Parallel Computing Toolbox)を参照してください。
名前と値の引数 UseParallel は、tall 配列、GPU 配列、コード生成では使用できません。
GPU 配列
Parallel Computing Toolbox™ を使用してグラフィックス処理装置 (GPU) 上で実行することにより、コードを高速化します。
使用上の注意事項および制限事項:
関数
lossでは代理分岐をもつ決定木学習器を使用して学習させたアンサンブルはサポートしていません。
詳細は、GPU での MATLAB 関数の実行 (Parallel Computing Toolbox)を参照してください。
バージョン履歴
R2011a で導入R2022a: 既定以外のコスト行列をもつモデルに対して loss で異なる値が返される
入力モデル オブジェクトに学習させるときに既定以外のコスト行列を指定すると、関数 loss で以前のリリースとは異なる値が返されます。
関数 loss は、Prior プロパティに格納された事前確率を使用して入力データの観測値の重みを正規化します。さらに、名前と値の引数 LossFun を "classifcost" または "mincost" として指定した場合、関数は Cost プロパティに格納されたコスト行列を使用します。Prior プロパティと Cost プロパティの値を関数で使用する方法については変更されていません。ただし、既定以外のコスト行列をもつモデルについて入力モデル オブジェクトに格納されるプロパティの値が変更されたため、関数から異なる値が返されることがあります。
プロパティの値の変更に関する詳細については、Cost property stores the user-specified cost matrixを参照してください。
ソフトウェアでコスト行列、事前確率、および観測値の重みを以前のリリースと同じように扱う場合は、誤分類コスト行列に応じた事前確率と観測値の重みの調整の説明に従って、既定以外のコスト行列の事前確率と観測値の重みを調整します。その後、分類モデルに学習させるときに、調整後の事前確率と観測値の重みを名前と値の引数 Prior と Weights を使用して指定し、既定のコスト行列を使用します。
R2022a: 欠損値がある予測子データに対して loss で NaN が返されることがある
関数 loss で加重平均分類損失を計算する際に、スコアが NaN の観測値が省略されなくなりました。そのため、予測子データ X または tbl 内の予測子変数に欠損値がある場合に loss で NaN が返されることがあります。ほとんどの場合、テスト セットの観測値に予測子の欠損がなければ、関数 loss で NaN が返されることはありません。
この変更により、fitcauto を使用する場合の分類モデルの自動選択が改善されます。この変更の前は、NaN 以外の予測子が少ないモデルが選択される (新しいデータの分類に最適であると予測される) ことがありました。
コードの loss で NaN が返される場合、このような結果にならないようにコードを更新できます。rmmissing または fillmissing を使用して、欠損値を削除するか置き換えます。
次の表に、関数 loss で NaN が返される可能性がある分類モデルを示します。詳細については、それぞれのオブジェクト関数 loss の「互換性の考慮事項」を参照してください。
| モデル タイプ | 完全またはコンパクトなモデル オブジェクト | オブジェクト関数 loss |
|---|---|---|
| 判別分析分類モデル | ClassificationDiscriminant, CompactClassificationDiscriminant | loss |
| 分類用のアンサンブル学習器 | ClassificationEnsemble, CompactClassificationEnsemble | loss |
| ガウス カーネル分類モデル | ClassificationKernel | loss |
| k 最近傍分類モデル | ClassificationKNN | loss |
| 線形分類モデル | ClassificationLinear | loss |
| ニューラル ネットワーク分類モデル | ClassificationNeuralNetwork, CompactClassificationNeuralNetwork | loss |
| サポート ベクター マシン (SVM) 分類モデル | loss |
MATLAB コマンド
次の MATLAB コマンドに対応するリンクがクリックされました。
コマンドを MATLAB コマンド ウィンドウに入力して実行してください。Web ブラウザーは MATLAB コマンドをサポートしていません。

Select a Web Site
Choose a web site to get translated content where available and see local events and offers. Based on your location, we recommend that you select: .
You can also select a web site from the following list:
How to Get Best Site Performance
Select the China site (in Chinese or English) for best site performance. Other MathWorks country sites are not optimized for visits from your location.
Americas
- América Latina (Español)
- Canada (English)
- United States (English)
Europe
- Belgium (English)
- Denmark (English)
- Deutschland (Deutsch)
- España (Español)
- Finland (English)
- France (Français)
- Ireland (English)
- Italia (Italiano)
- Luxembourg (English)
- Netherlands (English)
- Norway (English)
- Österreich (Deutsch)
- Portugal (English)
- Sweden (English)
- Switzerland
- United Kingdom (English)