異方性があるボクセル間隔の 3 次元イメージ ボリュームのスライスの調査
この例では、異方性 3 次元 MRI ボリュームのスライスを表示する方法を説明します。異方性イメージ ボリュームでは、"ボクセル" (ボリューム ピクセル) の間隔が空間次元ごとに異なります。たとえば、1×1×1 のボクセルをワールド座標内の 1×1×2 mm の領域にマッピングした場合、ボクセル間隔には異方性があります。既定では、Image Processing Toolbox™ の関数は、ボクセル単位でイメージを表示し、ワールド座標内で間隔が一様であると仮定しています。そのため、異方性ボリュームは歪んで表示されます。
この例では、各次元に沿ったスライスを歪ませることなく表示するための 2 つの方法を適用します。
表示設定の適用 — 表示関数で名前と値の引数を設定し、基になるイメージを変更することなく表示をカスタマイズします。
イメージ データの変換 — 基になるイメージ データに対してリサンプリングと幾何学的変換を適用してからイメージを表示します。
Medical Imaging Toolbox™ では、Image Processing Toolbox™ の機能が拡張されており、ボクセル、ワールド座標、および解剖学的座標軸の間の空間参照が自動的に管理されます。詳細については、Choose Approach for Medical Image Visualization (Medical Imaging Toolbox)を参照してください。
MRI データの読み込み
数値配列 D およびグレースケール カラーマップ map を含む MRI データ セットを読み込みます。数値配列には、MRI のイメージ データが含まれています。ボクセル間隔には異方性があり、ワールド座標におけるトランスバース スライスは、コロナル面とサジタル面の座標軸に沿ったスライスよりも 2.5 倍厚くなっています。カラーマップを使用することで、十分高いコントラストでボリュームを表示できます。関数 squeeze を使用して、大きさが 1 の次元を削除します。
load mri
DTransverse = squeeze(D);表示設定の適用
このセクションでは、表示関数の名前と値の引数を使用して、歪ませることなく MRI ボリュームを表示します。この名前と値の引数は、表示の縦横比のみを更新し、基になるイメージ データは更新しません。表示のみの調整は、内挿による生データの強度値の変更を行わない場合に役立ちます。この方法には、ボリュームを表示するたびに名前と値の引数を設定しなければならないという制約があります。また、montage などの一部の関数では、異方性ボリュームをスケーリングするための引数が用意されていません。
直交スライスの表示
orthosliceViewerオブジェクトを使用して、直交スライス平面を表示します。名前と値の引数 ScaleFactors を指定して、3 番目の次元にスケール係数 2.5 を適用します。各スライス ビューには、ボリューム内を移動するための十字形が表示されます。スライスを移動するには、カーソルが花形 ![]() に変わるまでいずれかの十字座標軸にカーソルを置き、クリックして新しい場所までドラッグします。他のスライスの表示は自動的に更新されます。
に変わるまでいずれかの十字座標軸にカーソルを置き、クリックして新しい場所までドラッグします。他のスライスの表示は自動的に更新されます。
figure orthosliceViewer(DTransverse,ScaleFactors=[1 1 2.5]);

スライスのスタックの表示
sliceViewerオブジェクトを使用して、スライスから成るスクロール可能なスタックを表示します。既定では、sliceViewer は 3 番目の次元に沿ってスライスを表示します。この例では、最初の 2 つの次元のボクセル間隔が等しいため、2 次元スライスは歪まずに表示されます。
sliceViewer(DTransverse,Parent=figure);
title("Transverse Slices")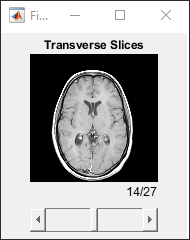
名前と値の引数 SliceDirection と ScaleFactors を使用して、それぞれスライスの方向の変更とスケール係数の適用を行います。スライスの方向を "X" として指定し、3 番目の次元のスケール係数を 2.5 として、サジタル面のスライスのスタックを表示します。
sliceViewer(DTransverse,SliceDirection="X",ScaleFactors=[1 1 2.5],Parent=figure); title("Sagittal Slices")

スライスの 3 次元オブジェクトとしての表示
関数volshowを SlicePlanes レンダリング スタイルと共に使用して、スライスを 3 次元で表示します。異方性ボクセルを歪ませずに表示するには、3 番目の次元のボリュームを 2.5 倍にスケーリングするaffinetform3dオブジェクトとして名前と値の引数 Transformation を指定します。軸に沿ってスライス間を移動するには、スライスをクリックしてドラッグします。目的の向きにボリュームを回転させるには、Figure ウィンドウ内の何もない場所をクリックしてドラッグします。特定のスライス面にビューをスナップさせるには、ビューアーの左下隅にある軸インジケーターで X、Y、または Z のラベルをクリックします。
sx = 1;
sy = 1;
sz = 2.5;
A = [sx 0 0 0; 0 sy 0 0; 0 0 sz 0; 0 0 0 1];
tform = affinetform3d(A);
vol = volshow(DTransverse,renderingStyle="SlicePlanes",Transformation=tform);
シネマティック レンダリング スタイルを使用して、高度なライティングが適用された 3 次元オブジェクトとしてボリュームを表示することもできます。
vol = volshow(DTransverse,renderingStyle="CinematicRendering",Transformation=tform);
ボリューム ビューアーを使用したスライスと 3 次元ボリュームの調査
ボリューム ビューアーアプリを使用して、イメージ ボリュームを対話形式で調査します。関数 volumeViewer を使用してアプリを起動します。異方性ボクセルを正しく表示するため、名前と値の引数 ScaleFactors を指定します。
volumeViewer(DTransverse,ScaleFactors=[1 1 2.5])
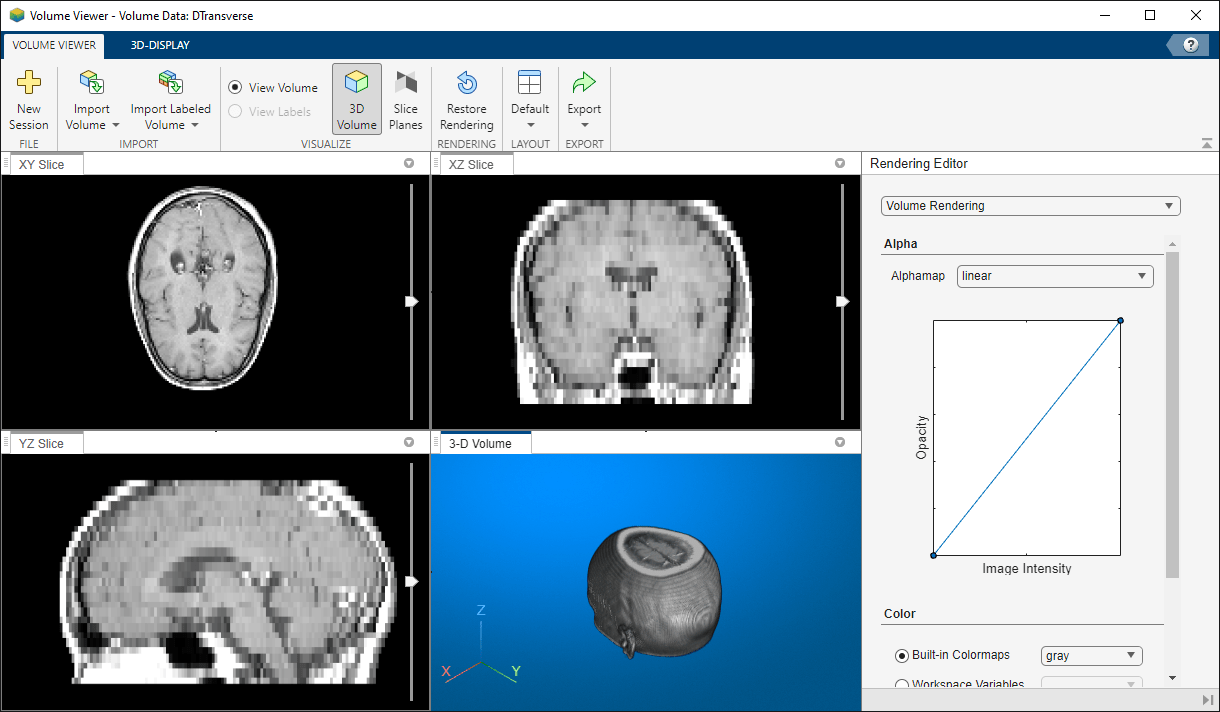
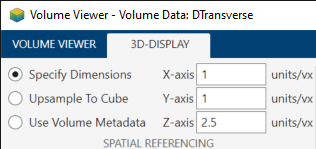
このアプリでは、スケール係数を指定することもできます。アプリのツールストリップの [3-D 表示] タブで、[空間参照] に各軸のスケール係数の値を入力します。値を入力すると、値の左側にあるオプションが自動的に [次元の指定] に変わります。
イメージ データの変換
このセクションでは、リサンプリングと幾何学的変換を使用してイメージ ボリュームを変更してから、そのイメージ ボリュームを表示します。リサンプリングを実行すると、内挿によって異方性ボクセルのグリッドが等方性グリッドに変換され、歪みをスケーリングすることなくイメージが表示されます。また、ボリュームの回転やその他の幾何学的変換を適用することで、目的の表示方法に合わせた向きでボリュームを表示させることができます。この方法には、ボリュームを表示するたびに名前と値の引数を指定する必要がないという利点があります。また、この方法では、montage のように、名前と値の引数を使用して既定のスケーリングやスライス方向を変更できない関数を使用することができます。
ボリュームのリサンプリングによる等方性ボクセル間隔への変換
関数imresize3を使用して元のボリュームをリサンプリングし、等方性ボクセルのグリッドに変換します。スケール係数 2.5 を使用して、等方性ボリュームの目標スライス数を計算します。
numSlices = round(2.5*size(DTransverse,3)); DTransverseIsotropic = imresize3(DTransverse,[128 128 numSlices]);
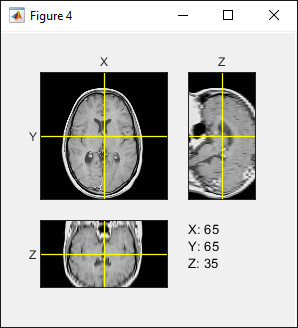
スケール係数を適用することなく、リサンプリング後のボリュームを表示関数で正確に表示することができます。名前と値の引数 ScaleFactors を使用せずに、リサンプリング後のボリュームを orthoSliceViewer で表示します。
figure orthosliceViewer(DTransverseIsotropic);
向きが指定されたスライスのモンタージュの表示
関数 montage は、3 番目の次元に沿ってイメージ ボリュームをスライスのスタックとして表示します。このセクションでは、3 つのすべての次元に沿って各解剖学的平面で要求される向きでスライスが表示されるように、イメージ データを変更する方法を説明します。
リサンプリング後のトランスバース ボリュームのスライスを表示します。十分高いコントラストでイメージを表示するため、カラーマップ map を適用します。
figure montage(DTransverseIsotropic,map)

title("Transverse Slices")ボリュームをサジタル面のスライスのスタックとして表示するため、DTransverseIsotropic の次元を並べ替えます。
DSagittal = permute(DTransverseIsotropic,[1 3 2]);
サジタル面のスライスで要求される向きに合わせてボリュームを回転させます。このとき、頭頂部がイメージの上部を向くようにし、前頭部がイメージの左側を向くようにします。
DSagittalRotated = imrotate3(DSagittal,90,[0 0 1],"cubic");サジタル面のスライスをモンタージュとして表示します。カラーマップを含め、表示範囲を設定します。名前と値の引数 Indices を使用して 20 ~ 100 番目のスライスを表示し、空のスライスを除外します。
figure
montage(DSagittalRotated,map,Indices=20:100)
title("Sagittal Slices")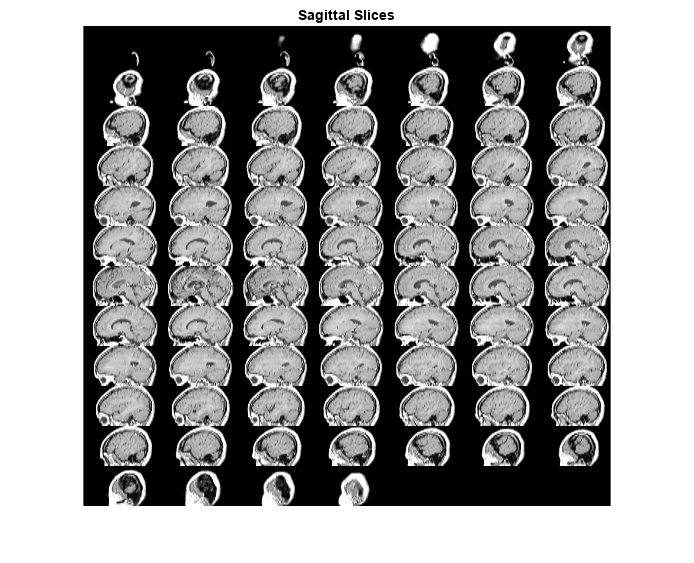
ボリュームをコロナル面のスライスのスタックとして表示するため、DTransverseIsotropic の次元を並べ替えます。
DCoronal = permute(DTransverseIsotropic,[2 3 1]);
コロナル面のスライスで要求される向きに合わせてボリュームを回転させます。このとき、頭頂部がイメージの上部を向くようにし、左側頭部がイメージの左側を向くようにします。
DCoronalRotated = imrotate3(DCoronal,90,[0 0 1],"cubic");コロナル面のスライスをモンタージュとして表示します。カラーマップを含め、表示範囲を設定します。名前と値の引数 Indices を使用して 17 ~ 127 番目のスライスを表示し、空のスライスを除外します。
figure
montage(DCoronalRotated,map,Indices=17:127)
title("Coronal Slices")
参考
ボリューム ビューアー | 医用画像ラベラー (Medical Imaging Toolbox) | sliceViewer | orthosliceViewer | volshow | imresize3 | imrotate3