edge
説明
e = edge(Mdl,Tbl,ResponseVarName)Tbl 内の予測子データと table 変数 ResponseVarName のクラス ラベルを使用して、学習済みのニューラル ネットワーク分類器 Mdl の分類エッジを返します。
e は、分類マージンの平均を表すスカラー値として返されます。
e = edge(___,Name,Value)
メモ
予測子データ X または Tbl 内の予測子変数に欠損値がある場合、関数 edge で NaN が返されることがあります。詳細については、欠損値がある予測子データに対して edge で NaN が返されることがあるを参照してください。
例
ニューラル ネットワーク分類器のテスト セットの分類エッジを計算します。
patients データ セットを読み込みます。データ セットから table を作成します。各行が 1 人の患者に対応し、各列が診断の変数に対応します。変数 Smoker を応答変数として使用し、残りの変数を予測子として使用します。
load patients
tbl = table(Diastolic,Systolic,Gender,Height,Weight,Age,Smoker);層化ホールドアウト分割を使用して、データを学習セット tblTrain とテスト セット tblTest に分割します。観測値の約 30% がテスト データ セット用に予約され、残りの観測値が学習データ セットに使用されます。
rng("default") % For reproducibility of the partition c = cvpartition(tbl.Smoker,"Holdout",0.30); trainingIndices = training(c); testIndices = test(c); tblTrain = tbl(trainingIndices,:); tblTest = tbl(testIndices,:);
学習セットを使用してニューラル ネットワーク分類器に学習させます。tblTrain の列 Smoker を応答変数として指定します。数値予測子を標準化するための指定を行います。
Mdl = fitcnet(tblTrain,"Smoker", ... "Standardize",true);
テスト セットの分類エッジを計算します。
e = edge(Mdl,tblTest,"Smoker")e = 0.8657
分類マージンの平均が 1 に近く、モデルの性能が全体的に高いことを示しています。
テスト セットの分類マージン、エッジ、誤差、予測を比較することにより、特徴選択を実行します。すべての予測子を使用して学習させたモデルのテスト セット メトリクスを予測子のサブセットのみを使用して学習させたモデルのテスト セット メトリクスと比較します。
標本ファイル fisheriris.csv を読み込みます。これには、アヤメについてのがく片の長さ、がく片の幅、花弁の長さ、花弁の幅、種の種類などのデータが格納されています。ファイルを table に読み込みます。
fishertable = readtable('fisheriris.csv');層化ホールドアウト分割を使用して、データを学習セット trainTbl とテスト セット testTbl に分割します。観測値の約 30% がテスト データ セット用に予約され、残りの観測値が学習データ セットに使用されます。
rng("default") c = cvpartition(fishertable.Species,"Holdout",0.3); trainTbl = fishertable(training(c),:); testTbl = fishertable(test(c),:);
学習セット内のすべての予測子を使用して 1 つのニューラル ネットワーク分類器に学習させ、PetalWidth を除くすべての予測子を使用してもう 1 つの分類器に学習させます。両方のモデルについて、Species を応答変数として指定し、予測子を標準化します。
allMdl = fitcnet(trainTbl,"Species","Standardize",true); subsetMdl = fitcnet(trainTbl,"Species ~ SepalLength + SepalWidth + PetalLength", ... "Standardize",true);
2 つのモデルのテスト セットの分類マージンを計算します。テスト セットに含まれる観測値は 45 個だけであるため、棒グラフを使用してマージンを表示します。
各観測値の分類マージンは、真のクラスの分類スコアと偽のクラスの最大スコアの差を表します。ニューラル ネットワーク分類器から返される分類スコアは事後確率であるため、マージンの値が 1 に近いほど信頼度が高い分類であることを示し、負のマージンの値は誤分類を示します。
tiledlayout(2,1) % Top axes ax1 = nexttile; allMargins = margin(allMdl,testTbl); bar(ax1,allMargins) xlabel(ax1,"Observation") ylabel(ax1,"Margin") title(ax1,"All Predictors") % Bottom axes ax2 = nexttile; subsetMargins = margin(subsetMdl,testTbl); bar(ax2,subsetMargins) xlabel(ax2,"Observation") ylabel(ax2,"Margin") title(ax2,"Subset of Predictors")

2 つのモデルのテスト セットの分類エッジ (分類マージンの平均) を比較します。
allEdge = edge(allMdl,testTbl)
allEdge = 0.8198
subsetEdge = edge(subsetMdl,testTbl)
subsetEdge = 0.9556
テスト セットの分類マージンと分類エッジからは、予測子のサブセットで学習させたモデルの方がすべての予測子で学習させたモデルよりも性能が優れていると考えられます。
2 つのモデルのテスト セットの分類誤差を比較します。
allError = loss(allMdl,testTbl); allAccuracy = 1-allError
allAccuracy = 0.9111
subsetError = loss(subsetMdl,testTbl); subsetAccuracy = 1-subsetError
subsetAccuracy = 0.9778
この場合も、予測子のサブセットのみを使用して学習させたモデルの方がすべての予測子を使用して学習させたモデルよりも性能が優れていることがわかります。
混同行列を使用してテスト セットの分類結果を可視化します。
allLabels = predict(allMdl,testTbl);
figure
confusionchart(testTbl.Species,allLabels)
title("All Predictors")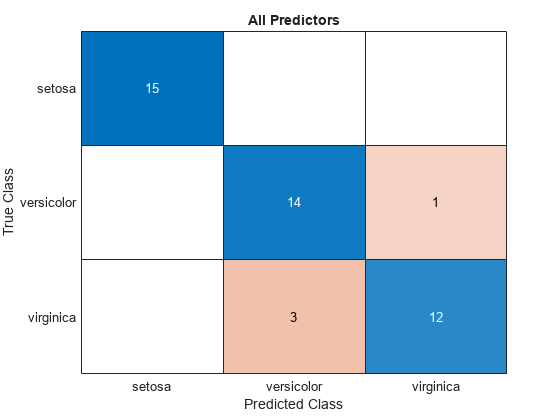
subsetLabels = predict(subsetMdl,testTbl);
figure
confusionchart(testTbl.Species,subsetLabels)
title("Subset of Predictors")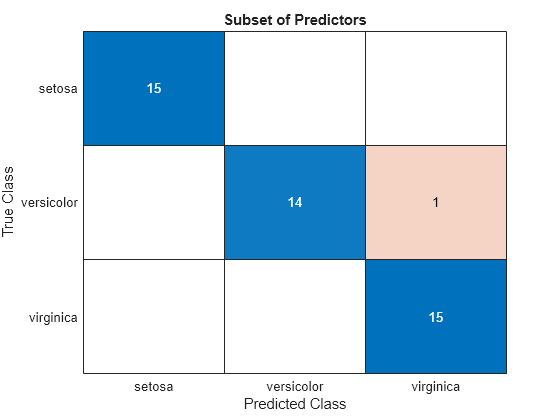
すべての予測子を使用して学習させたモデルには、テスト セットの観測値の誤分類が 4 件あります。予測子のサブセットを使用して学習させたモデルでは、テスト セットの観測値の誤分類は 1 件だけです。
2 つのモデルのテスト セットの性能から、PetalWidth を除くすべての予測子を使用して学習させたモデルを使用することを検討します。
入力引数
学習させたニューラル ネットワーク分類器。fitcnet によって返される ClassificationNeuralNetwork モデル オブジェクト、または compact によって返される CompactClassificationNeuralNetwork モデル オブジェクトとして指定します。
標本データ。table として指定します。Tbl の各行は 1 つの観測値に、各列は 1 つの予測子変数に対応します。オプションとして、応答変数用の追加列を Tbl に含めることができます。Tbl には、Mdl を学習させるために使用したすべての予測子が含まれていなければなりません。文字ベクトルの cell 配列ではない cell 配列と複数列の変数は使用できません。
Mdlを学習させるために使用した応答変数がTblに含まれている場合、ResponseVarNameまたはYを指定する必要はありません。table に格納されている標本データを使用して
Mdlに学習をさせた場合、edgeの入力データも table に含まれていなければなりません。Mdlに学習させるときにfitcnetで'Standardize',trueを設定した場合、予測子データの数値列が対応する平均および標準偏差を使用して標準化されます。
データ型: table
応答変数の名前。Tbl 内の変数の名前で指定します。Mdl を学習させるために使用した応答変数が Tbl に含まれている場合、ResponseVarName を指定する必要はありません。
ResponseVarName を指定する場合は、文字ベクトルまたは string スカラーとして指定しなければなりません。たとえば、応答変数が Tbl.Y として格納されている場合、ResponseVarName として 'Y' を指定します。それ以外の場合、Tbl の列は Tbl.Y を含めてすべて予測子として扱われます。
応答変数は、categorical 配列、文字配列、string 配列、logical ベクトル、数値ベクトル、または文字ベクトルの cell 配列でなければなりません。応答変数が文字配列の場合、各要素は配列の 1 つの行に対応しなければなりません。
データ型: char | string
クラス ラベル。categorical 配列、文字配列、string 配列、logical ベクトル、数値ベクトル、または文字ベクトルの cell 配列を指定します。
データ型: categorical | char | string | logical | single | double | cell
名前と値の引数
オプションの引数のペアを Name1=Value1,...,NameN=ValueN として指定します。ここで、Name は引数名で、Value は対応する値です。名前と値の引数は他の引数の後に指定しなければなりませんが、ペアの順序は重要ではありません。
R2021a より前では、名前と値をそれぞれコンマを使って区切り、Name を引用符で囲みます。
例: edge(Mdl,Tbl,"Response","Weights","W") は、table Tbl 内の変数 Response および W をそれぞれクラス ラベルおよび観測値の重みとして使用するように指定します。
予測子データにおける観測値の次元。'rows' または 'columns' として指定します。
メモ
観測値が列に対応するように予測子行列を配置して 'ObservationsIn','columns' を指定すると、計算時間が大幅に短縮される可能性があります。table の予測子データに対して 'ObservationsIn','columns' を指定することはできません。
データ型: char | string
観測値の重み。非負の数値ベクトルまたは Tbl 内の変数の名前を指定します。ソフトウェアは、X または Tbl の各観測値に、Weights の対応する値で重みを付けます。Weights の長さは、X または Tbl の観測値の数と等しくなければなりません。
入力データを table Tbl として指定した場合、Weights は数値ベクトルが含まれている Tbl 内の変数の名前にすることができます。この場合、Weights には文字ベクトルまたは string スカラーを指定しなければなりません。たとえば、重みベクトル W が Tbl.W として格納されている場合、'W' として指定します。
既定の設定では、Weights は ones(n,1) です。n は X または Tbl の観測値数です。
重みを指定した場合、edge は重み付きの分類エッジを計算し、合計がそれぞれのクラスの事前確率の値になるように重みを正規化します。
データ型: single | double | char | string
詳細
"分類エッジ" は "分類マージン" の平均であり、Weights を指定した場合は "分類マージン" の加重平均になります。
特徴選択を実行する場合などに複数の分類器から選択する方法の 1 つは、エッジが最大になる分類器を選択することです。
バイナリ分類の "分類マージン" は、各観測値における真のクラスの分類スコアと偽のクラスの分類スコアの差です。マルチクラス分類の "分類マージン" は、真のクラスの分類スコアと偽のクラスの最大スコアの差を表します。
各マージンのスケールが同じである場合 (つまり、スコア値が同じスコア変換に基づく場合)、マージンを分類の信頼尺度として使用できます。複数の分類器の中で、マージンが大きい分類器の方が優れています。
拡張機能
この関数は、GPU 配列を完全にサポートします。詳細は、GPU での MATLAB 関数の実行 (Parallel Computing Toolbox)を参照してください。
バージョン履歴
R2021a で導入edge は GPU 配列を完全にサポートします。
関数 edge で分類マージンの加重平均を計算する際に、スコアが NaN の観測値が省略されなくなりました。そのため、予測子データ X または Tbl 内の予測子変数に欠損値がある場合に edge で NaN が返されることがあります。ほとんどの場合、テスト セットの観測値に予測子の欠損がなければ、関数 edge で NaN が返されることはありません。
この変更により、fitcauto を使用する場合の分類モデルの自動選択が改善されます。この変更の前は、NaN 以外の予測子が少ないモデルが選択される (新しいデータの分類に最適であると予測される) ことがありました。
コードの edge で NaN が返される場合、このような結果にならないようにコードを更新できます。rmmissing または fillmissing を使用して、欠損値を削除するか置き換えます。
次の表に、オブジェクト関数 edge で NaN が返される可能性がある分類モデルを示します。詳細については、それぞれの関数 edge の「互換性の考慮事項」を参照してください。
| モデル タイプ | 完全またはコンパクトなモデル オブジェクト | オブジェクト関数 edge |
|---|---|---|
| 判別分析分類モデル | ClassificationDiscriminant, CompactClassificationDiscriminant | edge |
| 分類用のアンサンブル学習器 | ClassificationEnsemble, CompactClassificationEnsemble | edge |
| ガウス カーネル分類モデル | ClassificationKernel | edge |
| k 最近傍分類モデル | ClassificationKNN | edge |
| 線形分類モデル | ClassificationLinear | edge |
| ニューラル ネットワーク分類モデル | ClassificationNeuralNetwork, CompactClassificationNeuralNetwork | edge |
| サポート ベクター マシン (SVM) 分類モデル | edge |
MATLAB Command
You clicked a link that corresponds to this MATLAB command:
Run the command by entering it in the MATLAB Command Window. Web browsers do not support MATLAB commands.
Web サイトの選択
Web サイトを選択すると、翻訳されたコンテンツにアクセスし、地域のイベントやサービスを確認できます。現在の位置情報に基づき、次のサイトの選択を推奨します:
また、以下のリストから Web サイトを選択することもできます。
最適なサイトパフォーマンスの取得方法
中国のサイト (中国語または英語) を選択することで、最適なサイトパフォーマンスが得られます。その他の国の MathWorks のサイトは、お客様の地域からのアクセスが最適化されていません。
南北アメリカ
- América Latina (Español)
- Canada (English)
- United States (English)
ヨーロッパ
- Belgium (English)
- Denmark (English)
- Deutschland (Deutsch)
- España (Español)
- Finland (English)
- France (Français)
- Ireland (English)
- Italia (Italiano)
- Luxembourg (English)
- Netherlands (English)
- Norway (English)
- Österreich (Deutsch)
- Portugal (English)
- Sweden (English)
- Switzerland
- United Kingdom (English)