sam
スペクトル角マッパーを使用したスペクトルの類似度の評価
説明
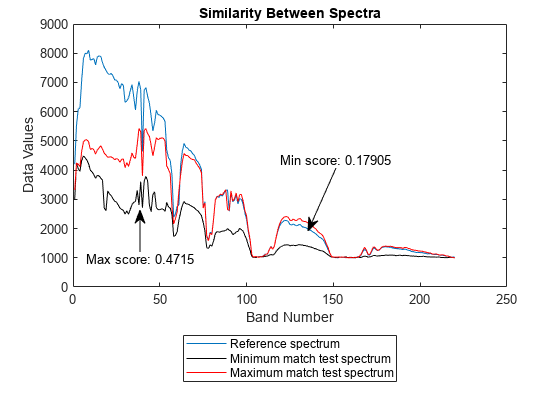
score = sam(inputData,refSpectra)inputData 内の各ピクセルのスペクトルと、指定した基準スペクトル refSpectra の間のスペクトルの類似度を評価します。この構文を使用して、ハイパースペクトル データ キューブ内で異なる領域または物質を特定します。
score = sam(testSpectra,refSpectra)testSpectra と基準スペクトル refSpectra の間のスペクトルの類似度を評価します。この構文を使用して、未知の物質のスペクトル シグネチャを基準スペクトルと比較したり、2 つのスペクトル シグネチャ間のスペクトルのばらつきを計算したりします。
メモ
この関数には、Hyperspectral Imaging Library for Image Processing Toolbox™ が必要です。Hyperspectral Imaging Library for Image Processing Toolbox はアドオン エクスプローラーからインストールできます。アドオンのインストールの詳細については、アドオンの入手と管理を参照してください。
Hyperspectral Imaging Library for Image Processing Toolbox は、MATLAB® Online™ および MATLAB Mobile™ によってサポートされないため、デスクトップの MATLAB が必要です。
例
入力引数
出力引数
制限
この関数は、パフォーマンスが既に最適化されているため、parfor ループをサポートしません。 (R2023a 以降)
詳細
参照
[1] Kruse, F.A., A.B. Lefkoff, J.W. Boardman, K.B. Heidebrecht, A.T. Shapiro, P.J. Barloon, and A.F.H. Goetz. “The Spectral Image Processing System (SIPS)—Interactive Visualization and Analysis of Imaging Spectrometer Data.” Remote Sensing of Environment 44, no. 2–3 (May 1993): 145–63. https://doi.org/10.1016/0034-4257(93)90013-N.
バージョン履歴
R2020a で導入
参考
spectralMatch | readEcostressSig | sid | hypercube | sidsam | jmsam