多重解像度のブロック化されたイメージ内の腫瘍の分類
この例では、Inception-v3 深層ニューラル ネットワークを使用して、メモリに収まらない可能性のある多重解像度のスライド ガラス標本全体のイメージ (WSI) を分類する方法を説明します。
腫瘍分類のための深層学習の手法は、組織を載せたスライド全体を撮像してデジタル化するデジタル病理学に依存しています。結果として得られる WSI は高解像度であり、およそ 200,000 行 100,000 列ピクセルになります。WSI は、イメージの効率的な表示、ナビゲーション、処理を容易にするために、多重解像度フォーマットで頻繁に格納されます。
この例は、ブロックベースの処理を使用して大きな WSI の学習を行うアーキテクチャを概説します。この例では、個々のブロックを normal または tumor に分類する転移学習の手法を使用して、Inception-v3 ベースのネットワークに学習をさせます。
学習データをダウンロードしてネットワークに学習をさせる手順を省略する場合は、この例のネットワークの学習または事前学習済みのネットワークのダウンロードのセクションに進んでください。
学習データの準備
Preprocess Multiresolution Images for Training Classification Networkの手順に従って、学習データと検証データを準備します。この前処理の例では、前処理した学習データストアと検証データストアを trainingAndValidationDatastores.mat という名前のファイルに保存しています。
変数 dataDir の値を、trainingAndValidationDatastores.mat ファイルが存在する場所に設定します。学習データストアと検証データストアを変数 dsTrainLabeled と変数 dsValLabeled に読み込みます。
dataDir = fullfile(tempdir,"Camelyon16"); load(fullfile(dataDir,"trainingAndValidationDatastores.mat"))
転移学習用の Inception-v3 ネットワーク層の設定
この例では、Inception-v3 ネットワークを使用します [2]。このネットワークは、ImageNet データベースの 100 万個を超えるイメージで学習させた畳み込みニューラル ネットワークです [3]。このネットワークは、深さが 48 層であり、イメージを 1,000 個のオブジェクト カテゴリ (キーボード、マウス、鉛筆、多くの動物など) に分類できます。
関数 imagePretrainedNetwork は、事前学習済みの Inception-v3 ネットワークを返します。Inception-v3 には、"Deep Learning Toolbox™ Model for Inception-v3 Network" サポート パッケージが必要です。このサポート パッケージがインストールされていない場合、関数によってダウンロード用リンクが表示されます。
この例の目的は、2 つのクラス (tumor と normal) 間でバイナリ セグメンテーションを実行することです。2 つのクラスを使用して、転移学習用に Inception-v3 ネットワークを準備します。
net = imagePretrainedNetwork("inceptionv3",numClasses=2);学習オプションの指定
平方根平均二乗伝播 (RMSProp) 最適化を使用してネットワークに学習をさせます。関数 trainingOptions (Deep Learning Toolbox) を使用して、RMSProp 用ハイパーパラメーター設定を指定します。
大量の学習データによってネットワークがすぐに収束に到達するため、MaxEpochs を小さい値に減らします。使用可能なメモリに応じて MiniBatchSize を指定します。ミニバッチのサイズを大きくすると学習を高速にすることができますが、サイズを大きくするほどネットワークの汎化性能は低下する可能性があります。正規化統計量を計算するために学習データをすべて読み取るのを避けるため、ResetInputNormalization を false に設定します。
maxEpochs = 1; miniBatchSize = 128; options = trainingOptions("rmsprop", ... MaxEpochs=maxEpochs, ... MiniBatchSize=miniBatchSize, ... Shuffle="every-epoch", ... ValidationFrequency=250, ... InitialLearnRate=1e-4, ... SquaredGradientDecayFactor=0.99, ... ResetInputNormalization=false, ... Verbose=true, ... VerboseFrequency=1, ... Plots="training-progress");
ネットワークの学習または事前学習済みネットワークのダウンロード
既定では、この例は分類ネットワークの事前学習済みバージョンをダウンロードします。この事前学習済みのネットワークを使用することで、学習の完了を待たずに例全体を実行できます。
ネットワークに学習させるには、次のコードで変数 doTraining を true に設定します。関数trainnet (Deep Learning Toolbox)を使用してモデルに学習させます。分類用にクロスエントロピー損失を指定します。既定では、関数 trainnet は利用可能な GPU がある場合にそれを使用します。GPU での学習には、Parallel Computing Toolbox™ ライセンスとサポートされている GPU デバイスが必要です。サポートされているデバイスの詳細については、GPU 計算の要件 (Parallel Computing Toolbox)を参照してください。そうでない場合、関数 trainnet は CPU を使用します。実行環境を指定するには、ExecutionEnvironment 学習オプションを使用します。
doTraining =false; if doTraining checkpointsDir = fullfile(dataDir,"checkpoints"); if ~exist(checkpointsDir,"dir") mkdir(checkpointsDir); end options.CheckpointPath=checkpointsDir; options.ValidationData=dsValLabeled; trainedNet = trainnet(dsTrainLabeled,net,"crossentropy",options); modelDateTime = string(datetime("now",Format="yyyy-MM-dd-HH-mm-ss")); save(dataDir+"trainedCamelyonNet-"+modelDateTime+".mat","trainedNet"); else trainedCamelyonNet_url = "https://www.mathworks.com/supportfiles/" + ... "vision/data/trainedCamelyonNet_v2.mat"; dataDir = fullfile(tempdir,"Camelyon16"); downloadTrainedNetwork(trainedCamelyonNet_url,dataDir); load(fullfile(dataDir,"trainedCamelyonNet_v2.mat")); end
Downloading pretrained network. This can take several minutes to download... Done.
テスト データのダウンロード
Camelyon16 テスト データセットは 130 個の WSI で構成されています。これらのイメージには、正常組織と腫瘍組織の両方が含まれています。各ファイルのサイズは約 2 GB です。
まず、ダウンロードしたデータを保存するフォルダーをマシンに作成します。
testDir = fullfile(dataDir,"testing"); testImageDir = fullfile(testDir,"images"); testAnnotationDir = fullfile(testDir,"lesion_annotations"); if ~exist(testDir,"dir") mkdir(testDir); mkdir(fullfile(testDir,"images")); mkdir(fullfile(testDir,"lesion_annotations")); end
データをダウンロードするには、データ セットの GigaDB Web サイトにアクセスし、テーブルで [ファイル] タブに移動します。[ファイル名] 列の名前をクリックして個々のファイルをダウンロードし、次の手順に従ってそれらのファイルを適切なサブフォルダーに移動します。
ファイル名が
test_で始まるすべての TIF イメージをダウンロードし、testImageDir変数で指定されたフォルダーに保存します。CAMELYON16 のテスト データ セットの
lesion_annotations.zipファイルをダウンロードし、testAnnotationDir変数で指定されたフォルダーに圧縮ファイルを解凍します。この表の [説明] 列の情報を使用して、学習データ セットとテスト データ セットの病変ファイルを区別できます。
テスト データの前処理
テスト イメージを管理する blockedImage オブジェクトの作成
テスト イメージのファイル名を取得します。その後、テスト イメージを管理するblockedImageオブジェクトの配列を作成します。各 blockedImage オブジェクトは、ディスク上の該当するイメージ ファイルを指します。
testFileSet = matlab.io.datastore.FileSet(testImageDir+filesep+"test*");
testImages = blockedImage(testFileSet);補助関数 setSpatialReferencingForCamelyon16 を使用して、すべての学習データに空間参照を設定します。この関数は、この例にサポート ファイルとして添付されています。関数 setSpatialReferencingForCamelyon16 は、TIF ファイル メタデータからの空間参照情報を使用して、各 blockedImage オブジェクトの WorldStart プロパティと WorldEnd プロパティを設定します。
testImages = setSpatialReferencingForCamelyon16(testImages);
組織マスクの作成
WSI データを効率的に処理するために、各テスト イメージに組織マスクを作成します。この処理は、正常部分の学習イメージの前処理に使用したものと同じです。詳細については、Preprocess Multiresolution Images for Training Classification Networkを参照してください。
normalMaskLevel = 8; testDir = fullfile(dataDir,"testing"); testTissueMaskDir = fullfile(testDir,"test_tissue_mask_level"+num2str(normalMaskLevel)); if ~isfolder(testTissueMaskDir) testTissueMasks = apply(testImages, @(bs)im2gray(bs.Data)<150, ... BlockSize=[512 512], ... Level=normalMaskLevel, ... UseParallel=canUseGPU, ... DisplayWaitbar=false, ... OutputLocation=testTissueMaskDir); save(fullfile(testTissueMaskDir,"testTissueMasks.mat"),"testTissueMasks") else % Load previously saved data load(fullfile(testTissueMaskDir,"testTissueMasks.mat"),"testTissueMasks"); end
組織マスクがもつレベルは 1 つのみであり、大きさはメモリ内に十分に収まるように小さくなっています。browseBlockedImages 補助関数を使用して、イメージ ブラウザー アプリで組織マスクを表示します。この補助関数は、この例にサポート ファイルとして添付されています。
browseBlockedImages(testTissueMasks,1);
腫瘍グラウンド トゥルース イメージの前処理
腫瘍マスクの解像度レベルを指定します。
tumorMaskLevel = 8;
補助関数 createMaskForCamelyon16TumorTissue を使用して、グラウンド トゥルース腫瘍イメージごとに腫瘍マスクを作成します。この補助関数は、この例にサポート ファイルとして添付されています。この関数は、イメージごとに以下の操作を行います。
注釈付き XML ファイル内のすべての ROI について、境界座標 (x, y) を読み取ります。
腫瘍組織と正常組織の ROI の境界座標を別々の cell 配列に分離します。
関数
polyToBlockedImageを使用して、境界座標の cell 配列をバイナリ ブロック化イメージに変換します。バイナリ イメージでは、ROI は腫瘍ピクセルを示し、背景は正常組織ピクセルを示します。腫瘍組織 ROI と正常組織 ROI の両方の内側にあるピクセルは、背景として分類されます。
testTumorMaskDir = fullfile(testDir,['test_tumor_mask_level' num2str(tumorMaskLevel)]); if ~isfolder(testTumorMaskDir) testTumorMasks = createMaskForCamelyon16TumorTissue(testImages, ... testAnnotationDir,testTumorMaskDir,tumorMaskLevel); save(fullfile(testTumorMaskDir,"testTumorMasks.mat"),"testTumorMasks") else load(fullfile(testTumorMaskDir,"testTumorMasks.mat"),"testTumorMasks"); end
腫瘍確率のヒートマップの予測
学習済み分類ネットワークを使用して、テスト イメージごとにヒートマップを予測します。ヒートマップから、各ブロックが tumor クラスである確率のスコアが得られます。この例では、テスト イメージごとに以下の操作を実行してヒートマップを作成します。
関数
selectBlockLocationsを使用してブロックを選択します。名前と値の引数InclusionThresholdを0として指定することにより、少なくとも 1 つの組織ピクセルをもつブロックをすべて含めるようにします。関数
applyと、補助関数predictBlockによって定義された処理演算を使用して、ブロックのバッチを処理します。この補助関数は、この例にサポート ファイルとして添付されています。補助関数predictBlockはデータのブロックに対して関数predict(Deep Learning Toolbox)を呼び出し、そのブロックがtumorである確率のスコアを返します。関数
writeを使用して、ヒートマップ データを TIF ファイルに書き込みます。すべてのブロックを処理した後の最終的な出力は、WSI 全体において腫瘍が見つかる確率を示すヒートマップとなります。
numTest = numel(testImages); outputHeatmapsDir = fullfile(testDir,"heatmaps"); networkBlockSize = [299,299,3]; for ind = 1:numTest % Check if TIF file already exists [~,id] = fileparts(testImages(ind).Source); outFile = fullfile(outputHeatmapsDir,id+".tif"); if ~exist(outFile,"file") bls = selectBlockLocations(testImages(ind),Levels=1, ... BlockSize=networkBlockSize, ... Mask=testTissueMasks(ind),InclusionThreshold=0); % Resulting heat maps are in-memory blockedImage objects bhm = apply(testImages(ind),@(x)predictBlockForCamelyon16(x,trainedNet), ... Level=1,BlockLocationSet=bls,BatchSize=128, ... PadPartialBlocks=true,DisplayWaitBar=false); % Write results to a TIF file write(bhm,outFile,BlockSize=[512 512]); end end
Elapsed time is 0.156674 seconds.
書き込まれたすべてのヒートマップを収集して blockedImage オブジェクトの配列とします。
heatMapFileSet = matlab.io.datastore.FileSet(outputHeatmapsDir,FileExtensions=".tif");
bheatMapImages = blockedImage(heatMapFileSet);ヒートマップの可視化
表示するテスト イメージを選択します。補助関数 showCamelyon16TumorAnnotations を使用して、Figure の左側に、グラウンド トゥルース境界座標をフリーハンド ROI として表示します。この補助関数は、この例にサポート ファイルとして添付されています。正常領域 (緑色の境界で示される) は、腫瘍領域 (赤色の境界で示される) の内側に発生することもあります。
idx = 27;
figure
tiledlayout(1,2)
nexttile
hBim1 = showCamelyon16TumorAnnotations(testImages(idx),testAnnotationDir);
title("Ground Truth")Figure の右側に、テスト イメージのヒートマップを表示します。
nexttile
hBim2 = bigimageshow(bheatMapImages(idx),Interpolation="nearest");
colormap(jet)座標軸をリンクさせ、対象領域を拡大します。
linkaxes([hBim1.Parent,hBim2.Parent])
xlim([53982, 65269])
ylim([122475, 133762])
title("Predicted Heatmap")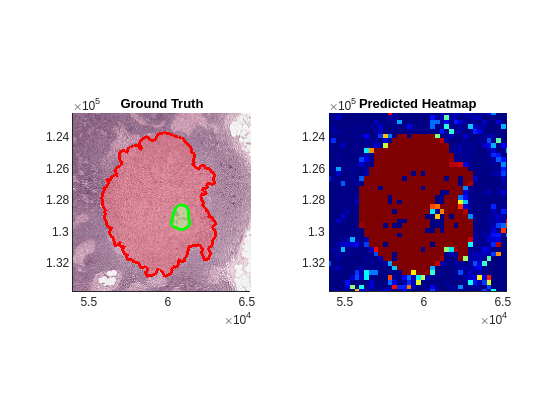
AUC-ROC 曲線によるしきい値の特定
テスト データ セットをテスト データ セットと検証データ セットに分割します。
rng("default") valPercent = 0.3; datapartition = cvpartition(numTest,"Holdout",valPercent); idxTest = training(datapartition); idxVal = test(datapartition);
検証には、最も粗い解像度のヒートマップとグラウンド トゥルース マスクを使用します。ヒートマップのサイズに合わせてグラウンド トゥルース マスクのサイズ変更と調整を行います。検証データ セット内の各イメージの各ピクセルに対するグラウンド トゥルース分類を格納する labels という名前のベクトルを作成します。腫瘍マスクからグラウンド トゥルース分類を取得できます。同様に、検証データ セット内の各イメージの各ピクセルに対するヒートマップ スコアを格納する scores という名前のベクトルを作成します。
labels = []; scores = []; for ind = find(idxVal)' heatMap = gather(bheatMapImages(ind)); tissueMask = gather(testTissueMasks(ind)); tissueMask = imresize(tissueMask,size(heatMap),"nearest"); tissueMask = tissueMask | heatMap>0; tumorMask = gather(testTumorMasks(ind)); tumorMask = imresize(tumorMask,size(heatMap),"nearest"); heatMap = heatMap.*tissueMask; tumorMask = tumorMask.*tissueMask; labels = [labels; reshape(tumorMask,[],1)]; scores = [scores; reshape(heatMap,[],1)]; end
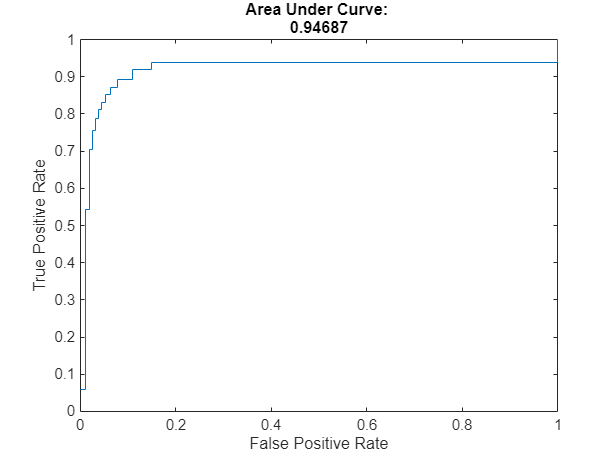
rocmetrics (Deep Learning Toolbox)関数を使用して、さまざまなしきい値で検証データ セットの ROC 曲線値を計算します。曲線下面積 (AUC) メトリクスを計算します。このメトリクスは、範囲 [0, 1] の値を返します。ここで、1 は完璧なモデル パフォーマンスを示します。このデータセットの AUC は 1 に近くなります。
roc = rocmetrics(labels,scores,true,AdditionalMetrics="PositivePredictiveValue");roc =
rocmetrics with properties:
Metrics: [910020×5 table]
AUC: 0.9462
Properties, Methods
roc.AUC
ans = single
0.9462
F1 スコアが最大となる最適なしきい値を選択します。
precisionValues = roc.Metrics.PositivePredictiveValue; recallValues = roc.Metrics.TruePositiveRate; F1score = 2.*precisionValues.*recallValues./(precisionValues+recallValues); [~,threshIdx] = max(F1score); thresh = roc.Metrics.Threshold(threshIdx)
thresh = single
0.6942
ROC 曲線と最適な動作点をプロットします。
figure plot(roc) hold on scatter(roc.Metrics.FalsePositiveRate(threshIdx), ... roc.Metrics.TruePositiveRate(threshIdx), ... "filled",DisplayName="true Optimal Operating Point") hold off
特定のしきい値でのテスト イメージの分類
各テスト イメージ内のブロックを分類し、関数applyと、補助関数 computeBlockConfusionMatrixForCamelyon16 によって定義された処理演算を使用して、混同行列を計算します。この補助関数は、この例にサポート ファイルとして添付されています。
補助関数 computeBlockConfusionMatrixForCamelyon16 は、各ヒートマップに対して以下の操作を行います。
ヒートマップのサイズに合わせてグラウンド トゥルース マスクのサイズ変更と調整を行います。
しきい値をヒートマップに適用します。
最も細かい解像度レベルで、すべてのブロックについて混同行列を計算します。この混同行列からは、真陽性 (TP)、偽陽性 (FP)、真陰性 (TN)、偽陰性 (FN) の各分類予測の数が得られます。
TP、FP、TN、FN のブロックの合計数を、ブロック化されたイメージ内の構造体として保存します。ブロック化されたイメージは、ブロック化されたイメージの配列
bcmatrix内の要素として返されます。ブロック化されたイメージ内の分類予測の数値ラベル付きイメージを保存します。0、1、2、3 の各値は、それぞれ TN、FP、FN、TP の結果に相当します。ブロック化されたイメージは、ブロック化されたイメージの配列
bcmatrixImage内の要素として返されます。
for ind = find(idxTest)' [bcmatrix(ind),bcmatrixImage{ind}] = apply(bheatMapImages(ind), ... @(bs,tumorMask,tissueMask)computeBlockConfusionMatrixForCamelyon16(bs, ... tumorMask,tissueMask,thresh), ... ExtraImages=[testTumorMasks(ind),testTissueMasks(ind)]); end
すべてのテスト イメージに対するグローバル混同行列を計算します。
bcmatrixEval = bcmatrix(idxTest);
cmArray = arrayfun(@(c)gather(c),bcmatrixEval);
cm = [sum([cmArray.tp]),sum([cmArray.fn]);
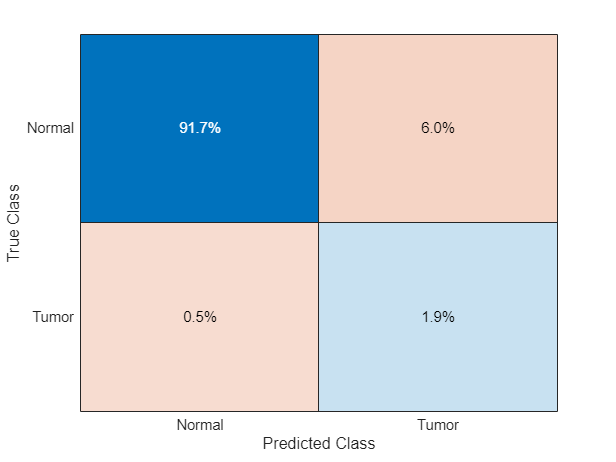
sum([cmArray.fp]),sum([cmArray.tn])];正規化されたグローバル混同行列の混同チャートを表示します。WSI イメージの大部分のブロックは正常組織であり、結果として真陰性予測のパーセンテージが高くなっています。偽陰性の予測が非常に少ないことを確認します。これにより、腫瘍の存在を必ず見つけられるようにします。
figure confusionchart(cm,["Tumor","Normal"],Normalization="row-normalized")
分類結果の可視化
グラウンド トゥルース ROI 境界座標と分類結果を比較します。Figure の左側に、グラウンド トゥルース境界座標をフリーハンド ROI として表示します。Figure の右側に、テスト イメージを表示し、混同行列に基づいて各ブロックの上に色を重ね合わせます。真陽性を赤、偽陽性をシアン、偽陰性を黄、真陰性を色なしで表示します。
偽陰性と偽陽性が、腫瘍領域の端の周辺に現れています。これは、ネットワークが部分的クラスでブロックの分類を行うことに困難があったことを示しています。
idx = 27; figure tiledlayout(1,2) nexttile hBim1 = showCamelyon16TumorAnnotations(testImages(idx),testAnnotationDir); title("Ground Truth") nexttile hBim2 = bigimageshow(testImages(idx)); cmColormap = [0 0 0; 0 1 1; 1 1 0; 1 0 0]; showlabels(hBim2,bcmatrixImage{idx}, ... Colormap=cmColormap,AlphaData=bcmatrixImage{idx}) title("Classified Blocks") linkaxes([hBim1.Parent,hBim2.Parent]) xlim([56000 63000]) ylim([125000 132600])
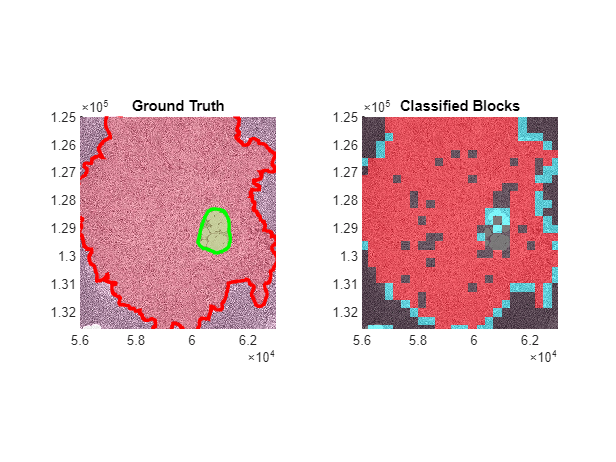
メモ: 腫瘍周辺の分類誤差を減らすためには、均質なブロックが少なくなるようにネットワークを維持します。学習データセットの Tumor ブロックを前処理するときに、名前と値の引数 InclusionThreshold の値を減らします。
参考文献
[1] Ehteshami Bejnordi, Babak, Mitko Veta, Paul Johannes van Diest, Bram van Ginneken, Nico Karssemeijer, Geert Litjens, Jeroen A. W. M. van der Laak, et al.“Diagnostic Assessment of Deep Learning Algorithms for Detection of Lymph Node Metastases in Women With Breast Cancer.”JAMA 318, no. 22 (December 12, 2017):2199–2210. https://doi.org/10.1001/jama.2017.14585.
[2] Szegedy, Christian, Vincent Vanhoucke, Sergey Ioffe, Jonathon Shlens, and Zbigniew Wojna.“Rethinking the Inception Architecture for Computer Vision.”Preprint, submitted December 2, 2015. https://arxiv.org/abs/1512.00567v3.
[3] ImageNet. https://www.image-net.org.
参考
blockedImageDatastore | blockedImage | blockLocationSet | selectBlockLocations | bigimageshow | dlnetwork (Deep Learning Toolbox) | trainingOptions (Deep Learning Toolbox) | trainnet (Deep Learning Toolbox) | rocmetrics (Deep Learning Toolbox)