ROI およびマスクからのラベル付きのブロック化されたイメージの作成
この例では、一連の ROI からラベル付きのブロック化されたイメージを作成する方法を説明します。
この例では、2 つの方法を使用してラベル付きデータの取得と表示を行います。1 番目の方法では、腫瘍組織と正常組織の領域の境界の座標を格納する多角形 ROI オブジェクトを使用します。関数 polyToBlockedImage は、ROI 座標をラベル付きのブロック化されたイメージに変換します。2 番目の方法では、マスクを使用して、イメージを組織と背景に分けるバイナリ セグメンテーションを示します。この例では、ROI とマスク イメージの情報を組み合わせ、腫瘍組織、正常組織、および背景領域に対応する数値ピクセル ラベルをもつ 1 つのブロック化されたイメージを作成します。
CAMELYON16 データ セットの腫瘍組織が含まれるリンパ節の学習イメージを変更したバージョンを使用して、ブロック化されたイメージを作成します。変更したイメージには、3 つの粗い解像度レベルが含まれています。空間参照は、縦横比が一定に維持され、各レベルで特徴がレジストレーションされるように調整されています。
bim = blockedImage("tumor_091R.tif");ラベル データの読み込み
CAMELYON16 データ セットは、最も細かい解像度レベルに対して、手動で注釈が付けられた領域境界を指定する一連の座標として、腫瘍領域と正常領域のラベルを提供します。正常領域と腫瘍領域の両方の境界内にピクセルが存在する場合、それらのピクセルの正しいラベルは正常組織です。
ブロック化されたイメージのラベル データを読み込みます。この例では、CAMELYON16 データ セットの tumor_091.tif イメージのラベルを変更したバージョンを使用します。元のラベルは XML 形式で格納されています。変更されたラベルは、リサンプリングされて MAT ファイルとして保存されています。
roiPoints = load("labelledROIs.mat")roiPoints = struct with fields:
nonCancerRegions: {[46×2 double]}
cancerRegions: {6×1 cell}
ROI オブジェクトとしてラベルを表現
腫瘍の境界と正常組織の境界の座標を格納する多角形 ROI オブジェクトを作成します。
tumorPolys = cellfun(@(position) images.roi.Polygon(Position=position, ... Visible="on",Color="r"),roiPoints.cancerRegions); normalPolys = cellfun(@(position) images.roi.Polygon(Position=position, ... Visible="on",Color="g"),roiPoints.nonCancerRegions);
ラベルが付けられた ROI の元のデータ上での表示
多角形 ROI を重ね合わせたイメージを表示します。ROI の座標系はイメージの座標系と同じであるため、表示されるイメージの解像度レベルを変更しても、ROI は正確に描画されます。
h = bigimageshow(bim);
set(tumorPolys,Parent=gca);
set(normalPolys,Parent=gca);
title("Resolution Level: " + num2str(h.ResolutionLevel));
1 つの ROI を拡大します。腫瘍領域の境界は赤で示され、その内側にある緑で示された正常組織を囲んでいます。
xlim([3940 4290])
ylim([2680 3010])
title("Resolution Level: " + num2str(h.ResolutionLevel));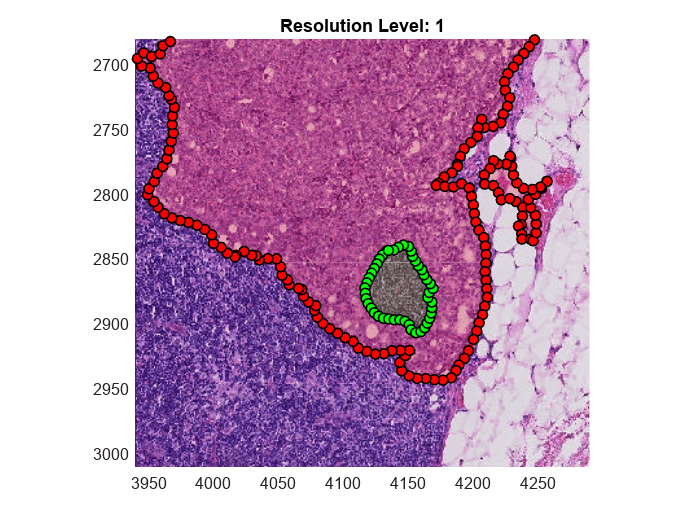
ROI データのラベル付きのブロック化されたイメージの作成
関数 polyToBlockedImage を使用して、ROI 座標データからラベル付きのブロック化されたイメージを作成します。関数 polyToBlockedImage には、ROI 座標、ROI ラベル、出力であるブロック化されたイメージのサイズを入力する必要があります。
正常領域と腫瘍領域の "xy" 座標データを取得し、それらを組み合わせて 1 つの cell 配列 roiPositions を作成します。
normalRegions = roiPoints.nonCancerRegions; tumorRegions = roiPoints.cancerRegions; roiPositions = [normalRegions; tumorRegions];
正常領域と腫瘍領域の数を調べます。また、正常組織にラベル 1 を割り当て、腫瘍組織にラベル 2 を割り当てます。roiPositions で指定されたのと同じ順序でラベルを割り当てます。格納に必要なメモリ量を減らすため、uint8 値としてラベルを指定します。
numNormalRegions = numel(normalRegions); numTumorRegions = numel(tumorRegions); roiLabelIDs = [ones(numNormalRegions,1,"uint8"); 2*ones(numTumorRegions,1,"uint8")];
新しいブロック化されたイメージに対し、目的の解像度レベルを選択します。この選択は効率と精度のトレードオフとなります。粗い解像度レベルを使用すると、処理時間と格納サイズが減少します。細かい解像度レベルを使用すると、マスクに保持される詳細レベルが増加します。多角形などの通常の ROI の場合は、粗い解像度を使用できます。小さいフリーハンド ROI の場合は、細かい解像度レベルが適している可能性があります。この例では中間の解像度レベルを使用します。
maskLevel = 2;
元のイメージ bim とサイズが一致するように、新しいブロック化されたイメージの目的の解像度レベルにおけるイメージ サイズを指定します。
imageSize = bim.Size(maskLevel,1:2);
ラベル付きのブロック化されたイメージを作成します。目的の解像度レベルにおいて、元のブロック化されたイメージ bim の空間参照を維持します。既定では、いずれの ROI にも含まれないピクセルには数値ラベル 0 が割り当てられます。
bROILabels = polyToBlockedImage(roiPositions,roiLabelIDs,imageSize, ... BlockSize=bim.BlockSize(maskLevel,1:2), ... WorldStart=bim.WorldStart(maskLevel,1:2),WorldEnd=bim.WorldEnd(maskLevel,1:2));
ROI ラベルと元データのオーバーレイの表示
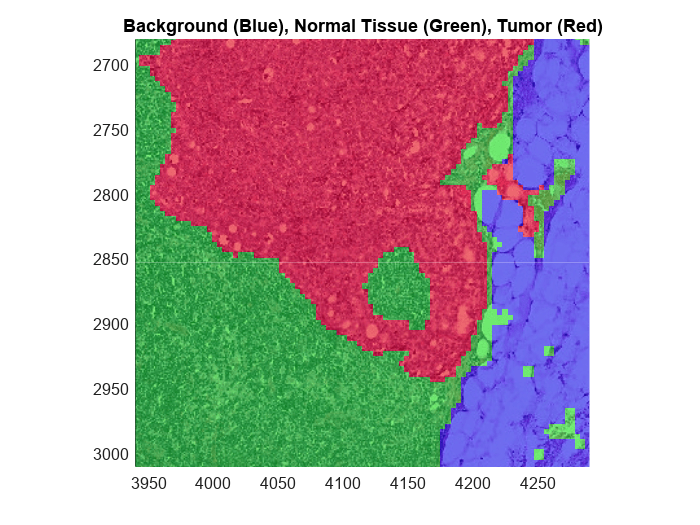
ラベル付きのブロック化されたイメージを元のイメージに重ね合わせて表示します。腫瘍領域は赤で示され、腫瘍組織に完全に囲まれている正常組織の領域は緑で示されています。背景、および背景と接している正常組織が青で示されています。これは、背景と接している正常領域が背景として間違って分類されていることを示しています。
hbim = bigimageshow(bim); showlabels(hbim,bROILabels,Colormap=[0 0 1; 0 1 0; 1 0 0]);
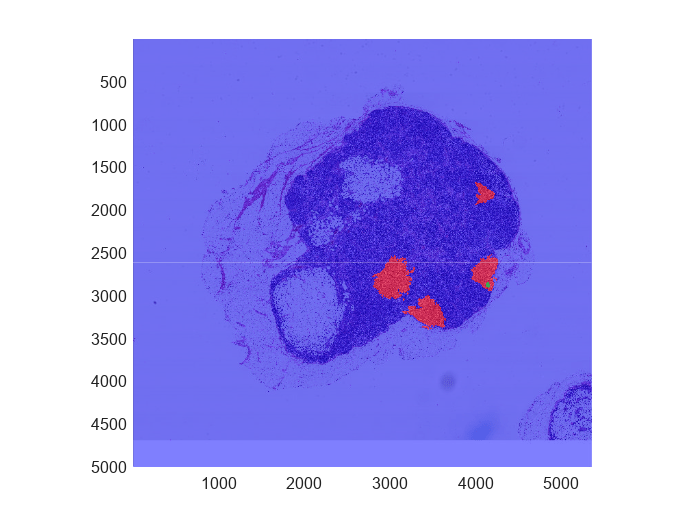
緑で表示されている正常組織の領域を拡大します。ROI の境界が十分細かく表されていることを目視で確認します。
xlim([3940 4290]) ylim([2680 3010])

マスクを使用した組織と背景の分離
イメージのセグメンテーションを使用して、背景と正常組織を正しく区別するマスクを作成します。しきい値処理を行うには、基となるイメージ データをメモリに読み取る必要があり、背景と前景の領域が十分大きくなければならないため、最も粗い解像度レベルが適しています。マスクは、グレースケール値が 130 未満のピクセルの場合に 1 (true) になります。関数 bwmorph を使用してモルフォロジー クロージングを実行し、マスク内にある小さな穴を塗りつぶします。
btissueMask = apply(bim,@(bs)bwmorph(im2gray(bs.Data)<130,"close"),Level=3);
bigimageshow(btissueMask);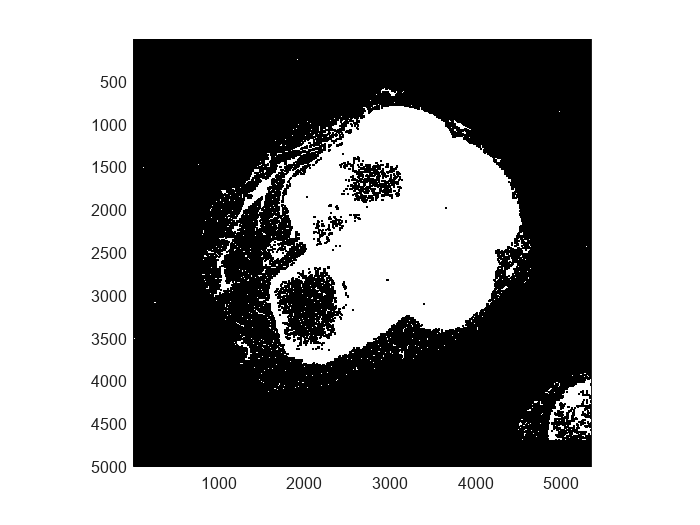
単一のラベル付きのブロック化されたイメージの作成
組織のマスクと ROI のラベル データを組み合わせ、最終的なラベル付きのブロック化されたイメージを作成します。ROI のイメージは組織のマスクよりも細かい解像度レベルで作成されていますが、両者は同じイメージから派生しており、同じワールド範囲をもっているため、両者をまとめて処理することができます。この例の最後で定義されている補助関数 combineLabels を使用します。
bLabels = apply(bROILabels,@combineLabels,ExtraImages=btissueMask);
ラベル付きのブロック化されたイメージを元のイメージに重ね合わせて表示します。3 つのラベル (正常、腫瘍、背景) は、それぞれ緑、赤、青で表示されます。
hbim = bigimageshow(bim);
showlabels(hbim,bLabels,Colormap=[0 0 1; 0 1 0; 1 0 0]);
title("Background (Blue), Normal Tissue (Green), Tumor (Red)")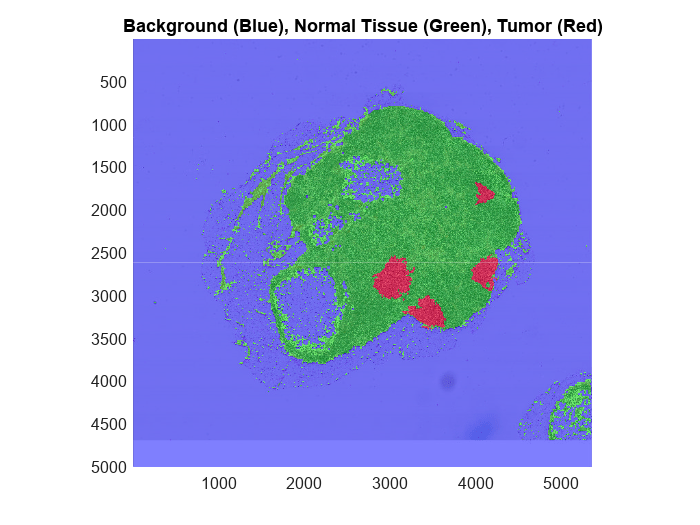
関心領域を拡大して詳しく調べます。
xlim([3940 4290]) ylim([2680 3010])
サポート関数
補助関数 combineLabels は、ROI ベースのラベル イメージとマスク ベースのラベル イメージを組み合わせて 1 つのラベル イメージにします。
function blabel = combineLabels(bs,btissueMask) % The tissue mask block is smaller in size btissueMask = imresize(btissueMask,size(bs.Data)); % Convert to labels, 0 is background, 1 is foreground. blabel = uint8(btissueMask); % Label tumor regions with 2 blabel(bs.Data==2) = 2; end
参考
blockedImage | bigimageshow | blockedImageDatastore