dicomreadVolume
DICOM イメージのセットからの 4 次元ボリュームの作成
構文
説明
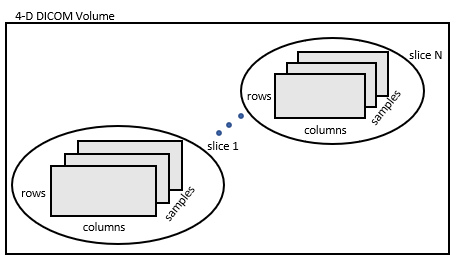
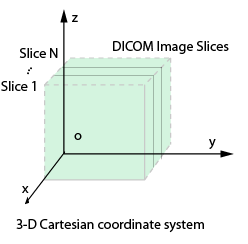
V = dicomreadVolume(sourceTable)sourceTable にリストされている入力ファイルから 4 次元 DICOM ボリュームを作成します。この table には DICOM ボリュームのメタデータを指定する 1 行のみが含まれていなければなりません。
V = dicomreadVolume(sourceTable,rowname)rowname にリストされている入力ファイルから 4 次元 DICOM ボリュームを作成します。sourceTable が複数の行を含む場合、この構文を使用します。