深層学習を使用した脳腫瘍の 3 次元セグメンテーション
この例では、3 次元医用画像から脳腫瘍のセマンティック セグメンテーションを実行する方法を説明します。
セマンティック セグメンテーションでは、イメージの各ピクセルまたは 3 次元ボリュームのボクセルにクラスでラベル付けします。この例では、3 次元 U-Net 深層学習ネットワークを使用して、磁気共鳴法 (MRI) スキャンで脳腫瘍のバイナリ セマンティック セグメンテーションを実行します。U-Net は、セマンティック セグメンテーションの分野で一般的になった高速かつ高効率でシンプルなネットワークです [1]。
医用画像のセグメンテーションの課題の 1 つとして、3 次元ボリュームの格納と処理に必要なメモリの量があります。GPU リソースの制約があるため、全入力ボリュームでネットワークに学習させてセグメンテーションを実行することは現実的ではありません。この例では、学習用およびセグメンテーション用にイメージを小さなパッチ (ブロック) に分割することで問題を解決します。
医用画像のセグメンテーションのもう 1 つの課題として、データにクラスの不均衡があり、従来のクロス エントロピー損失の使用時に学習の妨げになることがあります。この例では、重み付きマルチクラス Dice 損失関数 [4] を使用して、問題を解決します。クラスへの重み付けは、Dice スコアに対する大きな領域の影響を無効にするのに役立ち、より小さな領域をセグメント化する方法をネットワークが学習しやすくします。
この例では、事前学習済みの 3 次元 U-Net アーキテクチャを使用して脳腫瘍のセグメンテーションを実行する方法、および一連のテスト イメージを使用してネットワーク性能を評価する方法を説明します。オプションとして、BraTS データ セット [2] を使用して 3 次元 U-Net に学習させることもできます。
事前学習済みの 3 次元 U-Net の読み込み
事前学習済みの 3 次元 U-Net を trainedNet という変数にダウンロードします。事前学習済みのネットワークを使用することで、学習の完了を待たずに例を実行することができます。
dataDir = fullfile(tempdir,"BraTS"); if ~exist(dataDir,"dir") mkdir(dataDir); end trained3DUnetURL = "https://www.mathworks.com/supportfiles/"+ ... "vision/data/brainTumorSegmentation3DUnet_v2.zip"; downloadTrainedNetwork(trained3DUnetURL,dataDir); load(fullfile(dataDir,"brainTumorSegmentation3DUnet_v2.mat"));
セマンティック セグメンテーションの実行
事前学習済みのネットワークを使用して、テスト MRI ボリュームの腫瘍ラベルを予測します。
サンプル BraTS データの読み込み
この例では、BraTS データ セット [2] を使用します。データ セット全体では、750 人の患者の脳腫瘍のラベル付き MRI スキャンが格納されています。事前学習済みのネットワークを試すには、MathWorks® Web サイトから 5 つの MRI スキャンのサブセットとそれらに対応するラベルをダウンロードします。この例にサポート ファイルとして添付されている downloadBraTSSampleTestData 補助関数 [3] を使用して、サンプル データをダウンロードします。
downloadBraTSSampleTestData(dataDir);
MRI ボリュームの 1 つをピクセル ラベルのグラウンド トゥルースと共に読み込みます。各ボリュームは 4 次元配列であり、最初の 3 次元はイメージ データの高さ、幅、奥行きに対応し、4 番目の次元の各ページには各 MRI モダリティが格納されます。
testDir = fullfile(dataDir,"sampleBraTSTestSetValid"); data = load(fullfile(testDir,"imagesTest","BraTS446.mat")); labels = load(fullfile(testDir,"labelsTest","BraTS446.mat")); volTest = data.cropVol; volTestLabels = labels.cropLabel;
ブロック化イメージ内の脳腫瘍のセグメント化
大量のボリュームを効率的に処理するために、この例では、各 MRI スキャンをblockedImage (Image Processing Toolbox)オブジェクト内の一連の 3 次元ブロックとして処理します。ネットワークは、関数apply (Image Processing Toolbox)を使用して各ブロックのラベルを予測し、ブロックを結合して、セグメント化された完全なボリュームに戻します。
ダウンロードしたサンプル ボリューム用の blockedImage オブジェクトを作成します。
bim = blockedImage(volTest);
関数 apply は、blockedImage 内の各ブロックに対してカスタム関数を実行します。各ブロックに適用する関数として semanticsegBlock を定義します。classNames 内のクラスのラベルを予測するために、関数 semanticsegBlock は、関数semanticseg (Computer Vision Toolbox)を使用して事前学習済みネットワークを適用します。
classNames = ["background","tumor"]; semanticsegBlock = @(bstruct)semanticseg(bstruct.Data,trainedNet,Classes=classNames);
関数 apply を呼び出す際、ブロック サイズやブロック間の境界線のサイズなどのオプションを指定できます。ネットワーク出力サイズに合わせてブロック サイズを指定します。ネットワーク入力と同じサイズのサンプル イメージを事前学習済みネットワークに渡して、ネットワーク出力サイズを取得します。
networkInputSize = trainedNet.Layers(1).InputSize;
sampleImage = rand(networkInputSize,"single");
sampleOutput = predict(trainedNet,sampleImage);
networkOutputSize = size(sampleOutput);
blockSize = [networkOutputSize(1:3) networkInputSize(end)];オーバーラップするブロックを作成するには、非ゼロの境界サイズを指定します。この例では、ブロックと境界を合わせたサイズがネットワークの入力サイズと等しくなるように境界サイズを指定します。
borderSize = (networkInputSize(1:3) - blockSize(1:3))/2;
関数 semanticsegBlock をテスト イメージのすべてのブロックに適用します。部分ブロックのパディングを true として指定します。ボリューム データに複数のモダリティが含まれているため、既定のパディング手法である "replicate" が適しています。メモリ リソースが限られているマシンでメモリ不足エラーが発生するのを防ぐため、バッチ サイズを 1 として指定します。ただし、マシンに十分なメモリがある場合は、ブロック サイズを増やすことで処理速度を上げることができます。
batchSize = 1; results = apply(bim, ... semanticsegBlock, ... BlockSize=blockSize, ... BorderSize=borderSize, ... PadPartialBlocks=true, ... BatchSize=batchSize); predictedLabels = gather(results);
グラウンド トゥルース ラベルおよび予測されたラベルの中心スライスが奥行方向に沿って示されたモンタージュを表示します。
zID = size(volTest,3)/2;
zSliceGT = labeloverlay(volTest(:,:,zID),volTestLabels(:,:,zID));
zSlicePred = labeloverlay(volTest(:,:,zID),predictedLabels(:,:,zID));
figure
montage({zSliceGT,zSlicePred},Size=[1 2],BorderSize=5)
title("Labeled Ground Truth (Left) vs. Network Prediction (Right)")
次のアニメーションは、1 つのテスト ボリュームのグラウンド トゥルースと予測されたラベルの横方向のスライスを並べて表示したものです。左側はラベル付きのグラウンド トゥルース、右側はネットワーク予測です。

学習用データの準備
BraTS データ セット全体のダウンロード
学習を実行するには、BraTS データ セット全体をダウンロードしなければなりません。データ セットの合計サイズは約 7 GB で、750 人の患者のデータが格納されています。学習データ セットのダウンロードやネットワークの学習を行わない場合は、この例のネットワーク性能の評価のセクションに進んでください。
BraTS データをダウンロードするには、Medical Segmentation Decathlon の Web サイトに移動し、[Download Data] リンクをクリックします。Task01_BrainTumour.tar ファイル [3] をダウンロードします。変数 imageDir で指定されたディレクトリに TAR ファイルを解凍します。正常に解凍されると、imageDir には imagesTr、imagesTs、および labelsTr という 3 つのサブディレクトリをもつ Task01_BrainTumour という名前のディレクトリが含まれます。
サンプル データ セットと同様に、各スキャンは 4 次元配列であり、最初の 3 次元が 3 次元イメージの高さ、幅、奥行きに対応し、4 番目の次元の各ページが各モダリティに対応します。このデータ セットは、ボクセル ラベルを含む 484 個の学習ボリュームと 266 個のテスト ボリュームに分割されています。テスト ボリュームにはラベルがないため、この例ではテスト データを使用しません。代わりに、この例では 484 個の学習ボリュームを 3 つの個別のセットに分割して、学習、検証、およびテストに使用します。
データの前処理
より効率的に 3 次元 U-Net ネットワークに学習させるには、補助関数 preprocessBraTSDataset を使用して MRI データを前処理します。この関数は、この例にサポート ファイルとして添付されています。この補助関数は以下の操作を実行します。
脳と腫瘍を含む領域に合わせてデータをトリミングします。トリミングすると、各 MRI ボリュームの最も重要な部分とそれに対応するラベルを維持しながら、データのサイズが小さくなります。
平均を減算し、トリミングされた脳の領域の標準偏差で除算することにより、各ボリュームの各モダリティを個別に正規化します。
484 個の学習ボリュームを 400 個の学習セット、29 個の検証セット、および 55 個のテスト セットに分割します。
データの前処理は、完了するのに約 30 分かかることがあります。
sourceDataLoc = fullfile(dataDir,"Task01_BrainTumour"); preprocessDataLoc = fullfile(dataDir,"preprocessedDataset"); if ~isfolder(preprocessDataLoc) preprocessBraTSDataset(preprocessDataLoc,sourceDataLoc); end
学習および検証用のランダム パッチ抽出データストアの作成
3 次元の学習イメージ データを読み取って管理するためのimageDatastoreオブジェクトを作成します。カスタム読み取り関数 matRead を指定して、MAT ファイルからイメージ データを読み取ります。補助関数 matRead は、この例にサポート ファイルとして添付されています。
volLocTrain = fullfile(preprocessDataLoc,"imagesTr"); voldsTrain = imageDatastore(volLocTrain,FileExtensions=".mat",ReadFcn=@matRead);
ラベルを読み取って管理するためのpixelLabelDatastore (Computer Vision Toolbox)オブジェクトを作成します。変数 classNames で定義されているものと同じクラス名を指定します。ピクセル ラベル ID 1 はクラス名 "tumor" にマッピングされ、ピクセル ラベル ID 0 はクラス名 "background" にマッピングされます。
disp(classNames)
"background" "tumor"
pixelLabelID = [0 1]; lblLocTrain = fullfile(preprocessDataLoc,"labelsTr"); pxdsTrain = pixelLabelDatastore(lblLocTrain,classNames,pixelLabelID, ... FileExtensions=".mat",ReadFcn=@matRead);
グラウンド トゥルース イメージおよび対応するピクセル ラベル データからランダム パッチを抽出するrandomPatchExtractionDatastore (Image Processing Toolbox)オブジェクトを作成します。パッチ サイズとして 132 x 132 x 132 ボクセルを指定します。名前と値の引数 "PatchesPerImage" を指定して、学習中にボリュームとラベルの各ペアからランダムに配置された 16 個のパッチを抽出します。ミニバッチ サイズとして 4 を指定します。
patchSize = [132 132 132];
patchPerImage = 16;
miniBatchSize = 4;
patchdsTrain = randomPatchExtractionDatastore(voldsTrain,pxdsTrain,patchSize, ...
PatchesPerImage=patchPerImage);
patchdsTrain.MiniBatchSize = miniBatchSize;同様に、検証データ セットを管理するために、imageDatastore オブジェクト、pixeLabelDatastore オブジェクト、および randomPatchExtractionDatastore オブジェクトを作成します。検証データを使用して、ネットワークが継続的に学習しているか、学習中に適合不足や過適合が発生していないかを評価できます。
volLocVal = fullfile(preprocessDataLoc,"imagesVal"); voldsVal = imageDatastore(volLocVal,FileExtensions=".mat", ... ReadFcn=@matRead); lblLocVal = fullfile(preprocessDataLoc,"labelsVal"); pxdsVal = pixelLabelDatastore(lblLocVal,classNames,pixelLabelID, ... FileExtensions=".mat",ReadFcn=@matRead); patchdsVal = randomPatchExtractionDatastore(voldsVal,pxdsVal,patchSize, ... PatchesPerImage=patchPerImage); patchdsVal.MiniBatchSize = miniBatchSize;
3 次元 U-Net ネットワーク アーキテクチャの定義
この例では、3 次元 U-Net ネットワーク [1] を使用します。U-Net では、最初の一連の畳み込み層に最大プーリング層が点在し、入力イメージの解像度を逐次下げていきます。これらの層に、一連の畳み込み層が続き、その中にアップサンプリング演算処理が点在し、入力イメージの解像度を逐次上げていきます。バッチ正規化層は各 ReLU 層の前に作成されます。U-Net の名前は、このネットワークが文字「U」のように対称の形状で描けることに由来しています。
関数unet3d (Computer Vision Toolbox)を使用して、既定の 3 次元 U-Net ネットワークを作成します。2 クラスのセグメンテーションを指定します。境界アーティファクトを回避するために、有効な畳み込みパディングも指定します。
numChannels = 4; inputPatchSize = [patchSize numChannels]; numClasses = 2; [net,outPatchSize] = unet3d(inputPatchSize, ... numClasses,ConvolutionPadding="valid");
関数transformを、補助関数 augmentAndCrop3dPatch によって指定されたカスタム前処理演算と共に使用して、学習データと検証データを拡張します。この関数は、この例にサポート ファイルとして添付されています。関数 augmentAndCrop3dPatch は以下の操作を実行します。
学習データをランダムに回転および反転させて、学習をさらにロバストにする。この関数では検証データの回転または反転は行われません。
応答パッチをトリミングし、ネットワークの出力サイズを 44 x 44 x 44 ボクセルにする。
dsTrain = transform(patchdsTrain, ... @(patchIn)augmentAndCrop3dPatch(patchIn,outPatchSize,"Training")); dsVal = transform(patchdsVal, ... @(patchIn)augmentAndCrop3dPatch(patchIn,outPatchSize,"Validation"));
データはこの例の学習用データの準備セクションで既に正規化されており、image3dInputLayerでのデータ正規化は不要なため、入力層をデータ正規化が行われない入力層に置き換えます。
inputLayer = image3dInputLayer(inputPatchSize, ... Normalization="none",Name="ImageInputLayer"); net = replaceLayer(net,net.Layers(1).Name,inputLayer);
または、ディープ ネットワーク デザイナー アプリを使用して 3 次元 U-Net ネットワークを変更できます。
deepNetworkDesigner(net)
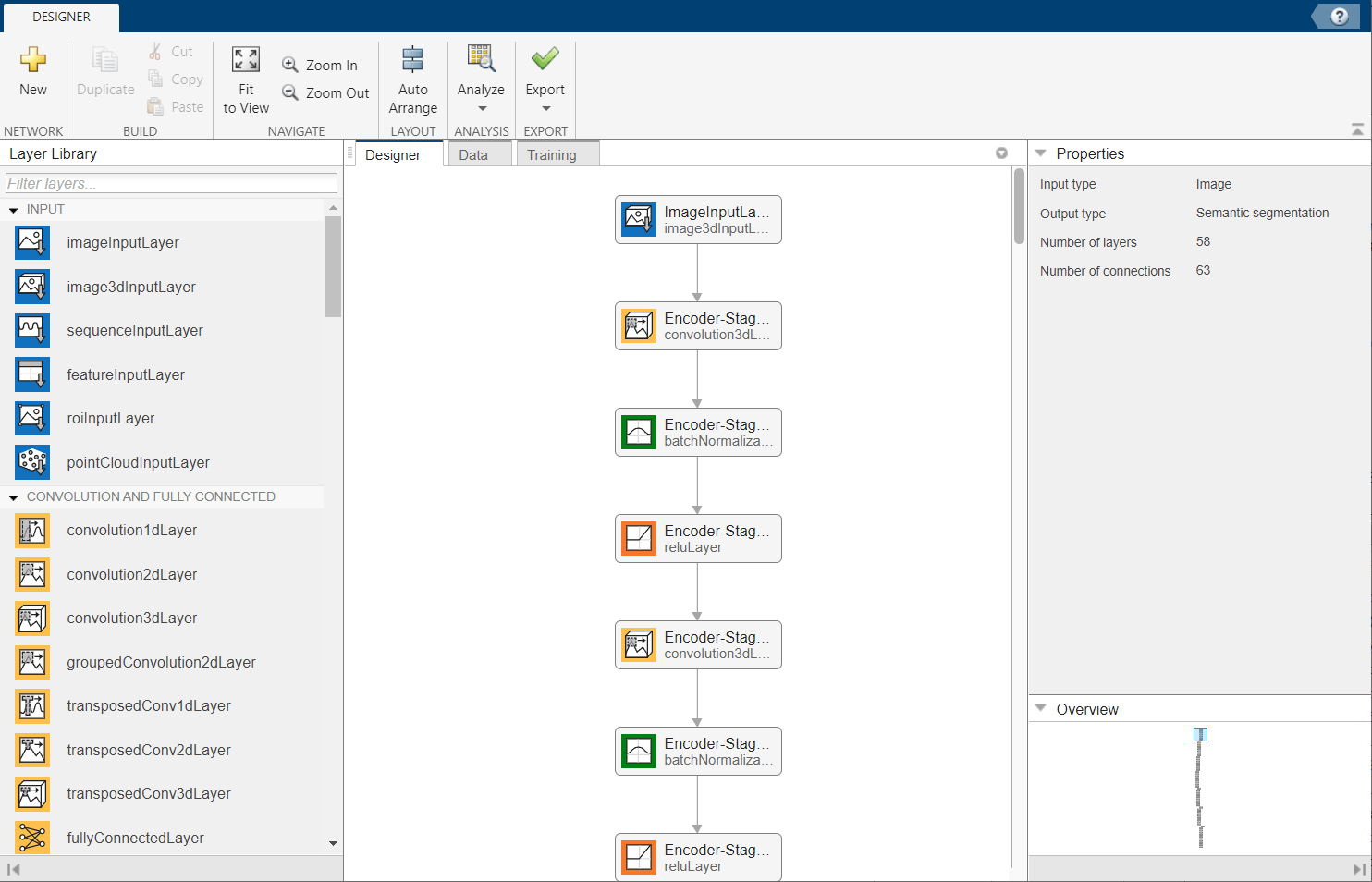
3 次元 U-Net の学習
学習オプションの指定
adam 最適化ソルバーを使用してネットワークに学習させます。関数trainingOptionsを使用してハイパーパラメーター設定を指定します。学習率の初期値は 5e-4 に設定されており、学習が進むにつれて徐々に減少します。GPU メモリに基づいて MiniBatchSize プロパティを試すことができます。GPU メモリを最大限に活用するには、バッチ サイズを大きくすることより入力パッチを大きくすることを優先します。MiniBatchSize の値が小さい場合、バッチ正規化層の効果が小さくなることに注意してください。MiniBatchSize に基づいて初期学習率を微調整します。
options = trainingOptions("adam", ... MaxEpochs=50, ... InitialLearnRate=5e-4, ... LearnRateSchedule="piecewise", ... LearnRateDropPeriod=5, ... LearnRateDropFactor=0.95, ... ValidationData=dsVal, ... ValidationFrequency=400, ... Plots="training-progress", ... Verbose=false, ... MiniBatchSize=miniBatchSize);
損失関数の定義
予測 Y とターゲット T を受け取って一般化 Dice 損失を返すカスタム損失関数 generalizedDiceLoss を定義します。一般化 Dice 類似度係数は、2 つのセグメント化されたイメージ間のオーバーラップを測定します。この係数は Sørensen-Dice 類似度に基づいており、期待される領域の逆サイズでクラスを重み付けすることにより、各クラスの類似度にもたらす貢献を制御します。詳細については、generalizedDice (Computer Vision Toolbox)を参照してください。
function loss = generalizedDiceLoss(Y,T) % Ignore any NaNs introduced to the training data during augmentation T(isnan(T)) = 0; z = generalizedDice(Y,T); % Calculate the mean of the Dice loss across the batch loss = 1 - mean(z,"all"); end
ネットワークの学習
この例では既定で、ダウンロードした事前学習済みの 3 次元 U-Net ネットワークを使用します。この事前学習済みのネットワークを使用することで、学習の完了を待たずにセマンティック セグメンテーションを実行してセグメンテーションの結果を評価できます。
ネットワークに学習させるには、次のコードで変数 doTraining を true に設定します。関数trainnetを使用してネットワークに学習させます。既定では、関数 trainnet は利用可能な GPU がある場合にそれを使用します。GPU での学習には、Parallel Computing Toolbox™ ライセンスとサポートされている GPU デバイスが必要です。サポートされているデバイスの詳細については、GPU 計算の要件 (Parallel Computing Toolbox)を参照してください。そうでない場合、関数 trainnet は CPU を使用します。実行環境を指定するには、ExecutionEnvironment 学習オプションを使用します。
doTraining =false; if doTraining [trainedNet,info] = trainnet(dsTrain,net,@generalizedDiceLoss,options); modelDateTime = string(datetime("now",Format="yyyy-MM-dd-HH-mm-ss")); save("trained3DUNet-"+modelDateTime+".mat","trainedNet"); end
3 次元 U-Net の評価
ネットワークのテストに使用するデータの場所を指定します。既定では、useFullTestSet は false であり、この例では、サンプル BraTS データの読み込みセクションでダウンロードした 5 つのサンプル ボリュームを使用します。useFullTestSet 値を true に変更すると、この例では、データ セット全体からテストに割り当てられたスキャンのうち 55 個が使用されます。
useFullTestSet =false; if useFullTestSet volLocTest = fullfile(preprocessDataLoc,"imagesTest"); lblLocTest = fullfile(preprocessDataLoc,"labelsTest"); else volLocTest = fullfile(testDir,"imagesTest"); lblLocTest = fullfile(testDir,"labelsTest"); end
学習データ セットおよび検証データ セットと同様に、imageDatastore オブジェクトと pixelLabelDatastore オブジェクトをそれぞれ作成して、テスト データ セットのイメージ データとラベルを管理します。
voldsTest = imageDatastore(volLocTest,FileExtensions=".mat", ... ReadFcn=@matRead); pxdsTest = pixelLabelDatastore(lblLocTest,classNames,pixelLabelID, ... FileExtensions=".mat",ReadFcn=@matRead);
各テスト ボリュームについて、ボリュームを blockedImage オブジェクトとして読み取り、関数apply (Image Processing Toolbox)を使用して各ブロックを処理します。関数 apply は、この例の終わりで定義されている補助関数 calculateBlockMetrics によって指定された演算を実行します。関数 calculateBlockMetrics は、各ブロックのセマンティック セグメンテーションを実行し、予測ラベルとグラウンド トゥルース ラベルの混同行列を計算します。
imageIdx = 1; datasetConfMat = table; while hasdata(voldsTest) % Read volume and label data vol = read(voldsTest); volLabels = read(pxdsTest); % Create blockedImage for volume and label data testVolume = blockedImage(vol); testLabels = blockedImage(volLabels{1}); % Calculate block metrics blockConfMatOneImage = apply(testVolume, ... @(block,labeledBlock) ... calculateBlockMetrics(block,labeledBlock,trainedNet,classNames), ... ExtraImages=testLabels, ... PadPartialBlocks=true, ... BlockSize=blockSize, ... BorderSize=borderSize); % Read all the block results of an image and update the image number blockConfMatOneImageDS = blockedImageDatastore(blockConfMatOneImage); blockConfMat = readall(blockConfMatOneImageDS); blockConfMat = struct2table([blockConfMat{:}]); blockConfMat.ImageNumber = imageIdx.*ones(height(blockConfMat),1); datasetConfMat = [datasetConfMat; blockConfMat]; imageIdx = imageIdx + 1; end
関数evaluateSemanticSegmentation (Computer Vision Toolbox)を使用して、セグメンテーションのデータ セット メトリクスとブロック メトリクスを評価します。
[metrics,blockMetrics] = evaluateSemanticSegmentation( ... datasetConfMat,classNames,Metrics="all");
Evaluating semantic segmentation results
----------------------------------------
* Selected metrics: global accuracy, class accuracy, IoU, weighted IoU.
* Processed 5 images.
* Finalizing... Done.
* Data set metrics:
GlobalAccuracy MeanAccuracy MeanIoU WeightedIoU
______________ ____________ _______ ___________
0.99902 0.97292 0.95915 0.99807
各イメージについて計算されたジャッカード スコアを表示します。
metrics.ImageMetrics.MeanIoU
ans = 5×1
0.9676
0.9521
0.9568
0.9537
0.9635
サポート関数
補助関数 calculateBlockMetrics は、ブロックのセマンティック セグメンテーションを実行し、予測ラベルとグラウンド トゥルース ラベルの混同行列を計算します。この関数は、ブロックに関する混同行列とメタデータが格納されたフィールドをもつ構造体を返します。この構造体と関数 evaluateSemanticSegmentation を使用することで、メトリクスを計算してブロック単位で結果を集約できます。
function blockMetrics = calculateBlockMetrics(bstruct,gtBlockLabels,net,classNames) % Segment block predBlockLabels = semanticseg(bstruct.Data,net,Classes=classNames); % Trim away border region from gtBlockLabels blockStart = bstruct.BorderSize + 1; blockEnd = blockStart + bstruct.BlockSize - 1; gtBlockLabels = gtBlockLabels( ... blockStart(1):blockEnd(1), ... blockStart(2):blockEnd(2), ... blockStart(3):blockEnd(3)); % Evaluate segmentation results against ground truth confusionMat = segmentationConfusionMatrix(predBlockLabels,gtBlockLabels); % blockMetrics is a struct with confusion matrices, image number, % and block information blockMetrics.ConfusionMatrix = confusionMat; blockMetrics.ImageNumber = bstruct.ImageNumber; blockInfo.Start = bstruct.Start; blockInfo.End = bstruct.End; blockMetrics.BlockInfo = blockInfo; end
参考文献
[1] Çiçek, Özgün, Ahmed Abdulkadir, Soeren S. Lienkamp, Thomas Brox, and Olaf Ronneberger. “3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation.” In Medical Image Computing and Computer-Assisted Intervention – MICCAI 2016, edited by Sebastien Ourselin, Leo Joskowicz, Mert R. Sabuncu, Gozde Unal, and William Wells, 9901:424–32. Cham: Springer International Publishing, 2016. https://doi.org/10.1007/978-3-319-46723-8_49.
[2] Isensee, Fabian, Philipp Kickingereder, Wolfgang Wick, Martin Bendszus, and Klaus H. Maier-Hein. “Brain Tumor Segmentation and Radiomics Survival Prediction: Contribution to the BRATS 2017 Challenge.” In Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries, edited by Alessandro Crimi, Spyridon Bakas, Hugo Kuijf, Bjoern Menze, and Mauricio Reyes, 10670:287–97. Cham: Springer International Publishing, 2018. https://doi.org/10.1007/978-3-319-75238-9_25.
[3] "Brain Tumours". Medical Segmentation Decathlon. http://medicaldecathlon.com/
BraTS データセットは、CC-BY-SA 4.0 のライセンスに基づき Medical Segmentation Decathlon によって提供されます。一切の保証および表明を行いません。詳細については、ライセンスを参照してください。MathWorks® は、この例のサンプル BraTS データの読み込みセクションにリンクが示されているデータ セットを変更しています。変更されたサンプル データ セットは主に脳と腫瘍を含む領域に合わせてトリミングされており、各チャネルは平均を減算し、トリミングされた脳の領域の標準偏差で除算することにより、個別に正規化されています。
[4] Sudre, Carole H., Wenqi Li, Tom Vercauteren, Sebastien Ourselin, and M. Jorge Cardoso. “Generalised Dice Overlap as a Deep Learning Loss Function for Highly Unbalanced Segmentations.” In Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support, edited by M. Jorge Cardoso, Tal Arbel, Gustavo Carneiro, Tanveer Syeda-Mahmood, João Manuel R.S. Tavares, Mehdi Moradi, Andrew Bradley, et al., 10553:240–48. Cham: Springer International Publishing, 2017. https://doi.org/10.1007/978-3-319-67558-9_28.
[5] Ronneberger, Olaf, Philipp Fischer, and Thomas Brox. “U-Net: Convolutional Networks for Biomedical Image Segmentation.” In Medical Image Computing and Computer-Assisted Intervention – MICCAI 2015, edited by Nassir Navab, Joachim Hornegger, William M. Wells, and Alejandro F. Frangi, 9351:234–41. Cham: Springer International Publishing, 2015. https://doi.org/10.1007/978-3-319-24574-4_28.
参考
unet3d (Computer Vision Toolbox) | trainnet | trainingOptions | blockedImage (Image Processing Toolbox) | apply (Image Processing Toolbox) | randomPatchExtractionDatastore (Image Processing Toolbox) | semanticseg (Computer Vision Toolbox)