スペクトル ライブラリを使用したエンドメンバー物質の同定
この例では、ハイパースペクトル イメージ内に存在するエンドメンバー物質のクラスを同定する方法を示します。エンドメンバーは、1 つの表面物質に属するピクセルの反射率特性を示すピュア スペクトル シグネチャです。既存のエンドメンバー抽出または同定アルゴリズムでは、ハイパースペクトル イメージ内のピュア ピクセルを抽出または同定します。ただし、これらの手法でエンドメンバー スペクトルが属する物質名またはクラスは同定されません。この例では、エンドメンバー シグネチャを抽出してから、スペクトル マッチングを使用してハイパースペクトル イメージ内のエンドメンバー物質のクラスを分類または同定します。
この例には、Hyperspectral Imaging Library for Image Processing Toolbox™ が必要です。Hyperspectral Imaging Library for Image Processing Toolbox は、アドオン エクスプローラーからインストールできます。アドオンのインストールの詳細については、アドオンの取得と管理を参照してください。Hyperspectral Imaging Library for Image Processing Toolbox は、MATLAB® Online™ および MATLAB® Mobile™ でサポートされないため、デスクトップの MATLAB® が必要です。
この例では、エンドメンバー物質の同定のため、1) ECOSTRESS スペクトル ライブラリのスペクトル シグネチャを基準スペクトルとして使用し、2) Jasper Ridge データセットのデータ サンプルをテスト データとして使用します。
ECOSTRESS スペクトル ライブラリからの基準データの読み取り
ECOSTRESS ライブラリ ファイルが格納されているファイルの絶対パスを追加して、ライブラリから読み取るファイルの名前を指定します。
filenames = ["water.seawater.none.liquid.tir.seafoam.jhu.becknic.spectrum.txt",... "water.tapwater.none.liquid.all.tapwater.jhu.becknic.spectrum.txt",... "water.ice.none.solid.all.ice_dat_.jhu.becknic.spectrum.txt",... "vegetation.tree.eucalyptus.maculata.vswir.jpl087.jpl.asd.spectrum.txt",... "soil.utisol.hapludult.none.all.87p707.jhu.becknic.spectrum.txt",... "soil.mollisol.cryoboroll.none.all.85p4663.jhu.becknic.spectrum.txt",... "manmade.road.tar.solid.all.0099uuutar.jhu.becknic.spectrum.txt",... "manmade.concrete.pavingconcrete.solid.all.0092uuu_cnc.jhu.becknic.spectrum.txt"]; lib = readEcostressSig(filenames);
lib データを表示してその値を検査します。このデータは、クラス、サブクラス、波長、および反射率関係の情報を指定する struct 変数です。
lib
lib=1×8 struct array with fields:
Name
Type
Class
SubClass
ParticleSize
Genus
Species
SampleNo
Owner
WavelengthRange
Origin
CollectionDate
Description
Measurement
FirstColumn
SecondColumn
WavelengthUnit
DataUnit
FirstXValue
LastXValue
NumberOfXValues
AdditionalInformation
Wavelength
Reflectance
⋮
ECOSTRESS スペクトル ライブラリから読み取ったスペクトル シグネチャをプロットします。
figure hold on for idx = 1:numel(lib) plot(lib(idx).Wavelength,lib(idx).Reflectance,LineWidth=2); end axis tight box on xlabel("Wavelength (\mum)"); ylabel("Reflectance (%)"); classNames = {lib.Class}; legend(classNames,Location="northeast") title("Reference Spectra from ECOSTRESS Library"); hold off

テスト データの読み取り
関数 hypercube を使用して Jasper Ridge データセットからテスト データを読み取ります。この関数は、データ キューブおよびテスト データから読み取ったメタデータ情報を格納する hypercube オブジェクトを返します。テスト データには、198 個のスペクトル バンドと、399.4 nm ~ 2457 nm の範囲の波長が含まれています。スペクトル分解能は最大 9.9 nm、各バンド イメージの空間分解能は 100 x 100 です。テスト データには、道路、土壌、水、および樹木を含む 4 つの潜在エンドメンバーが格納されています。
hcube = imhypercube("jasperRidge2_R198.hdr");エンドメンバー スペクトルの抽出
テスト データに存在するスペクトルが特徴的なエンドメンバーの総数を計算するには、関数 countEndmembersHFC を使用します。この関数は、Harsanyi–Farrand–Chang (HFC) 法を使用してエンドメンバーの数を求めます。誤検知を防止するために偽警報の確率 (PFA) を低い値に設定します。
numEndmembers = countEndmembersHFC(hcube,PFA=10^-27);
N-FINDR 法を使用してテスト データのエンドメンバーを抽出します。
endMembers = nfindr(hcube,numEndmembers);
hypercube オブジェクト hcube から波長の値を読み取ります。抽出したエンドメンバー シグネチャをプロットします。このテスト データは、4 つのエンドメンバー物質で構成されています。これらの物質のクラス名はスペクトル マッチングで同定できます。
figure plot(hcube.Wavelength,endMembers,LineWidth=2) axis tight xlabel("Wavelength (nm)") ylabel("Data Values") title("Endmembers Extracted using N-FINDR") num = 1:numEndmembers; legendName = "Endmember "+num; legend(legendName)
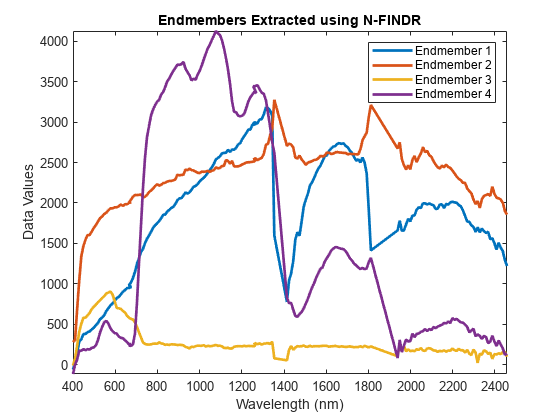
エンドメンバー物質の同定
エンドメンバー物質の名前を同定するには、関数 spectralMatch を使用します。この関数は、ライブラリ ファイルと分類対象のエンドメンバー スペクトルの間のスペクトルの類似度を計算します。マッチング スコアの計算にはスペクトル情報発散 (SID) 法を選択します。通常、SID スコアの値が低いほど、テスト スペクトルと基準スペクトルのマッチングが良好であることを意味します。次に、最適に一致する基準スペクトルのクラスに入るようにテスト スペクトルを分類します。
たとえば、3 番目と 4 番目のエンドメンバー物質のクラスを同定するには、ライブラリ シグネチャとそれぞれのエンドメンバー スペクトルの間でスペクトルの類似度を求めます。最小 SID スコア値のインデックスは、スペクトル ライブラリ内のクラス名を指定します。3 番目のエンドメンバー スペクトルは海水と同定され、4 番目のエンドメンバー スペクトルは樹木と同定されます。
wavelength = hcube.Wavelength; detection = cell(1,1); cnt = 1; queryEndmember = [3 4]; for num = 1:numel(queryEndmember) spectra = endMembers(:,queryEndmember(num)); scoreValues = spectralMatch(lib,spectra,wavelength,Method="sid"); [~, matchIdx] = min(scoreValues); detection{cnt} = lib(matchIdx).Class; disp("Endmember spectrum "+queryEndmember(num)+" is identified as "+detection{cnt}) cnt=cnt+1; end
Endmember spectrum 3 is identified as Sea Water Endmember spectrum 4 is identified as Tree
テスト データ内のエンドメンバー領域のセグメント化
同定結果を視覚的に検査するには、テスト データ内のエンドメンバー物質に固有のイメージ領域の位置を推定してセグメント化します。関数 sid を使用して、ピクセル スペクトルと抽出したエンドメンバー スペクトルの間のピクセル単位のスペクトル類似度を計算します。次に、しきい値処理を実行し、テスト データの対象エンドメンバー領域をセグメント化して、セグメント化されたイメージを生成します。しきい値として 15 を選択し、最適に一致するピクセルを選択します。
可視化のため、colorize 関数を使用してテスト データの RGB バージョンを作成します。RGB イメージの上に、セグメント化された海水と樹木のエンドメンバー領域のオーバーレイを作成します。
threshold = 15; rgbImg = colorize(hcube,Method="rgb",ContrastStretching=true); overlayImg = rgbImg; labelColor = ["Blue","Green"]; datacube = gather(hcube); segmentedImg = cell(size(datacube,1),size(datacube,2),numel(queryEndmember)); for num = 1:numel(queryEndmember) scoreMap = sid(hcube,endMembers(:,queryEndmember(num))); segmentedImg{num} = scoreMap <= threshold; overlayImg = imoverlay(overlayImg,segmentedImg{num},labelColor{num}); end
結果の表示
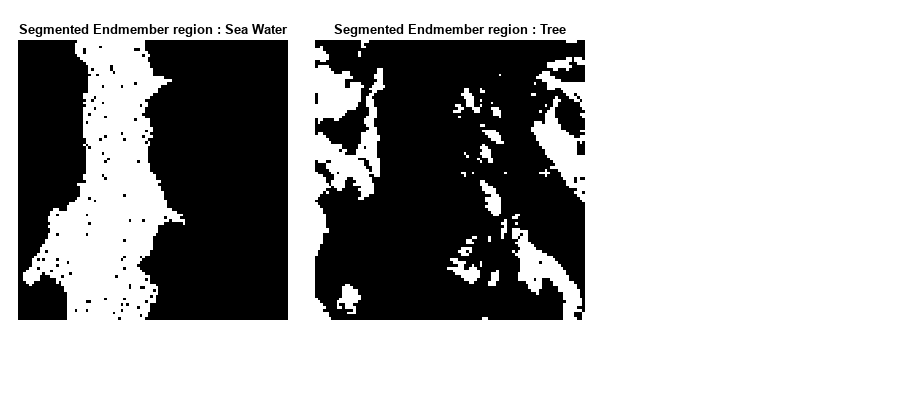
同定結果を視覚的に検査します。海水と樹木のエンドメンバー領域のバイナリ セグメンテーション結果を表示します。
figure(Position=[0 0 900 400]) plotdim = [0.02 0.2 0.3 0.7;0.35 0.2 0.3 0.7]; for num = 1:numel(queryEndmember) subplot(Position=plotdim(num,:)) imagesc(segmentedImg{num}) title("Segmented Endmember region: "+detection{num}); colormap([0 0 0;1 1 1]) axis off end
RGB イメージの上にエンドメンバー領域のオーバーレイを表示します。
figure(Position=[0 0 900 400]) subplot(Position=[0 0.2 0.3 0.7]) imagesc(rgbImg) title("RGB Transformation of Test Data"); axis off subplot(Position=[0.35 0.2 0.3 0.7]) imagesc(overlayImg) title("Segmented Regions Overlay") hold on dim = [0.66 0.6 0.3 0.3]; annotation("textbox",dim,String="Sea Water",Color=[1 1 1],BackgroundColor=[0 0 1],FitBoxToText="on"); dim = [0.66 0.5 0.3 0.3]; annotation("textbox",dim,String="Tree",BackgroundColor=[0 1 0],FitBoxToText="on"); hold off axis off

参考文献
[1] Kruse, F.A., A.B. Lefkoff, J.W. Boardman, K.B. Heidebrecht, A.T. Shapiro, P.J. Barloon, and A.F.H. Goetz. "The Spectral Image Processing System (SIPS)—Interactive Visualization and Analysis of Imaging Spectrometer Data." Remote Sensing of Environment 44, no. 2–3 (May 1993): 145–63. https://doi.org/10.1016/0034-4257(93)90013-N.
[2] ECOSTRESS Spectral Library: https://speclib.jpl.nasa.gov
[3] Meerdink, Susan K., Simon J. Hook, Dar A. Roberts, and Elsa A. Abbott. “The ECOSTRESS Spectral Library Version 1.0.” Remote Sensing of Environment 230 (September 2019): 111196. https://doi.org/10.1016/j.rse.2019.05.015.
[4] Baldridge, A.M., S.J. Hook, C.I. Grove, and G. Rivera. “The ASTER Spectral Library Version 2.0.” Remote Sensing of Environment 113, no. 4 (April 2009): 711–15. https://doi.org/10.1016/j.rse.2008.11.007.
参考
hypercube | colorize | spectralMatch | readEcostressSig | countEndmembersHFC