サポート ベクター マシン分類器を使用したハイパースペクトル イメージの分類
この例では、サポート ベクター マシン (SVM) 分類器を使用してハイパースペクトル イメージの分類を実行する方法を示します。
この例には、Hyperspectral Imaging Library for Image Processing Toolbox™ が必要です。Hyperspectral Imaging Library for Image Processing Toolbox は、アドオン エクスプローラーからインストールできます。アドオンのインストールの詳細については、アドオンの取得と管理を参照してください。Hyperspectral Imaging Library for Image Processing Toolbox は、MATLAB® Online™ および MATLAB® Mobile™ でサポートされないため、デスクトップの MATLAB® が必要です。
ハイパースペクトル イメージは複数のスペクトル バンドにわたって取得され、それぞれが異なる波長における同じシーンを表す数百のバンド イメージで構成されます。この例では、400 ~ 2500 nm の波長範囲の航空機搭載可視/赤外イメージング分光器 (AVIRIS) センサーで取得した Indian Pines データ セットを使用します。このデータ セットには、16 個のクラスと 220 個のバンド イメージが格納されています。各イメージのサイズは 145×145 ピクセルです。
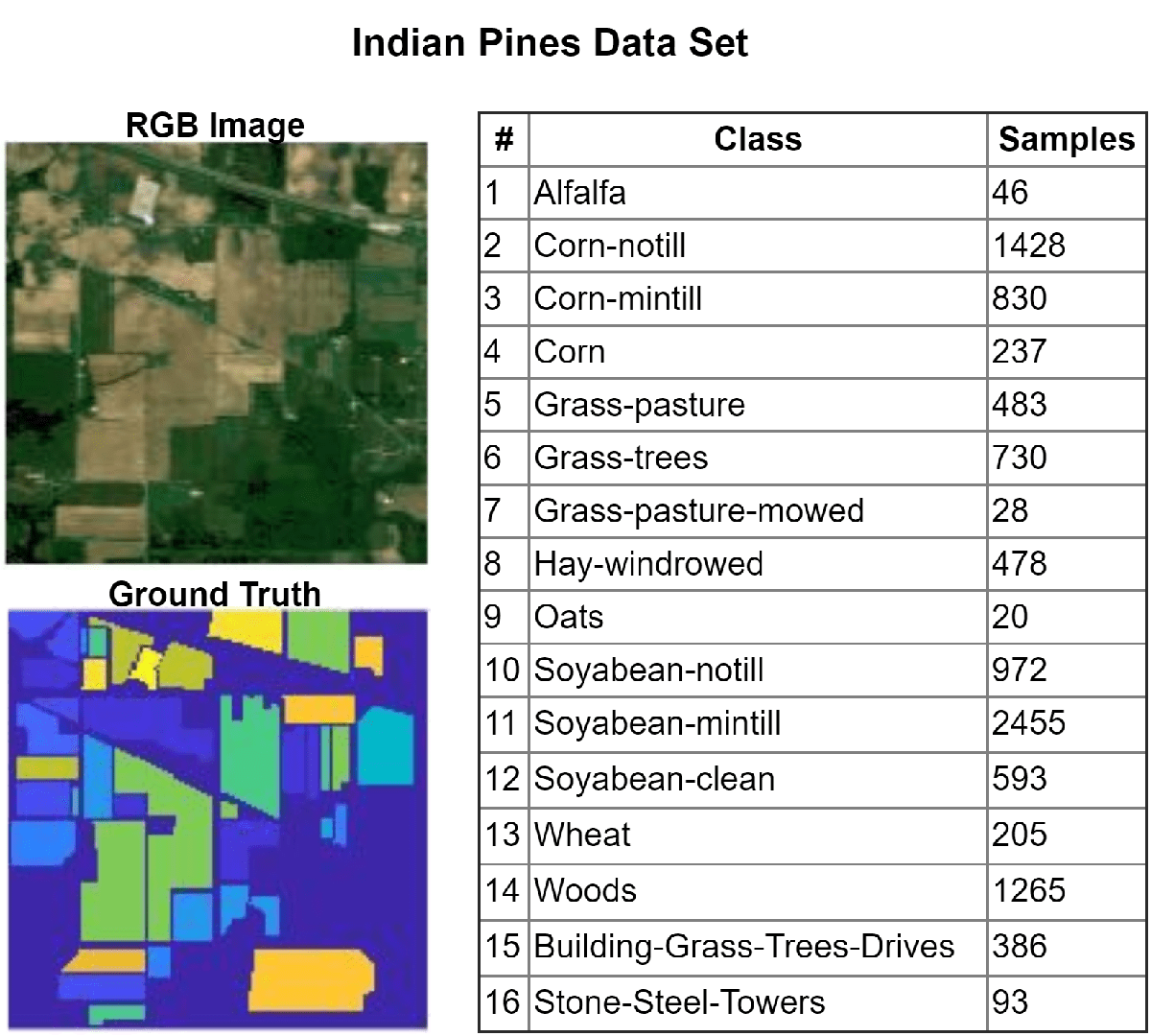
この例では、次を行います。
2 次元ガウス フィルターを使用してハイパースペクトル イメージを前処理します。
SVM 分類器を使用して分類を実行します。
分類精度、分類マップ、混同行列などの分類結果を表示します。
ハイパースペクトル データセットの読み込み
ハイパースペクトル データをワークスペースに読み取ります。
hcube = imhypercube("indian_pines.dat");データ セットのグラウンド トゥルースをワークスペースに読み込みます。
gtLabel = load("indian_pines_gt.mat");
gtLabel = gtLabel.indian_pines_gt;
numClasses = 16;ハイパースペクトル データの前処理
removeBands関数を使用して、データから水の吸収のバンドを削除します。
band = [104:108 150:163 220]; % Water absorption bands
newhcube = removeBands(hcube,BandNumber=band);colorize関数を使用して、入力の超立方体の RGB イメージを推定します。
rgbImg = colorize(newhcube,method="rgb");imgaussfilt関数を使用して、ハイパースペクトル データの各バンド イメージにガウス フィルター () を適用し、グレースケール イメージに変換します。
hsData = gather(newhcube); [M,N,C] = size(hsData); hsDataFiltered = zeros(size(hsData)); for band = 1:C bandImage = hsData(:,:,band); bandImageFiltered = imgaussfilt(bandImage,2); bandImageGray = mat2gray(bandImageFiltered); hsDataFiltered(:,:,band) = uint8(bandImageGray*255); end
分類のためのデータの準備
フィルター処理したハイパースペクトル データを、各ピクセルのフィルター処理済みスペクトル応答を格納する特徴ベクトルのセットに再形成します。
DataVector = reshape(hsDataFiltered,[M*N C]);
グラウンド トゥルース イメージを、クラス ラベルを格納するベクトルに再形成します。
gtVector = gtLabel(:);
クラス ラベルを格納するグラウンド トゥルース ベクトルの位置インデックスを検出します。値が 0 のラベルは、ラベルがなく、クラスを表していないため、破棄します。
gtLocs = find(gtVector~=0); classLabel = gtVector(gtLocs);
cvpartition (Statistics and Machine Learning Toolbox)関数を使用し、指定した学習パーセンテージで学習用とテスト用の位置インデックスを作成します。
per = 0.1; % Training percentage
cv = cvpartition(classLabel,HoldOut=1-per);グラウンド トゥルース位置インデックスを学習用の位置インデックスとテスト用の位置インデックスに分割します。
locTrain = gtLocs(cv.training); locTest = gtLocs(~cv.training);
SVM を使用した分類
fitcecoc (Statistics and Machine Learning Toolbox)関数を使用して SVM 分類器に学習させます。
svmMdl = fitcecoc(DataVector(locTrain,:),gtVector(locTrain,:));
テスト データを使用して SVM 分類器をテストします。
[svmLabelOut,~] = predict(svmMdl,DataVector(locTest,:));
分類結果の表示
分類精度を計算して表示します。
svmAccuracy = sum(svmLabelOut == gtVector(locTest))/numel(locTest);
disp(["Overall Classification Accuracy (%) = ",num2str(svmAccuracy.*100)])"Overall Classification Accuracy (%) = " "95.9996"
SVM 分類マップを作成します。
svmPredLabel = gtLabel; svmPredLabel(locTest) = svmLabelOut;
RGB イメージ、グラウンド トゥルース マップ、SVM 分類マップを表示します。
cmap = parula(numClasses);
figure
montage({rgbImg,gtLabel,svmPredLabel},cmap,Size=[1 3],BorderSize=10)
title("RGB Image | Ground Truth Map | SVM Classification Map")
% Specify the intensity limits for the colorbar
minR = min(min(gtLabel(:)),min(svmPredLabel(:)));
maxR = max(max(gtLabel(:)),max(svmPredLabel(:)));
clim([minR maxR])
colorbar
混同行列を表示します。
fig = figure; confusionchart(gtVector(locTest),svmLabelOut,ColumnSummary="column-normalized") fig_Position = fig.Position; fig_Position(3) = fig_Position(3)*1.5; fig.Position = fig_Position; title("Confusion Matrix: SVM Classification Results")

参照
[1] Patro, Ram Narayan, Subhashree Subudhi, Pradyut Kumar Biswal, and Fabio Dell’acqua. “A Review of Unsupervised Band Selection Techniques: Land Cover Classification for Hyperspectral Earth Observation Data.” IEEE Geoscience and Remote Sensing Magazine 9, no. 3 (September 2021): 72–111. https://doi.org/10.1109/MGRS.2021.3051979.
参考
hypercube | colorize | removeBands | fitcecoc (Statistics and Machine Learning Toolbox) | confusionchart (Statistics and Machine Learning Toolbox) | imgaussfilt