小さいアンサンブルでの LPBoost と TotalBoost
この例では、LPBoost および TotalBoost アルゴリズムを活用する方法を示します。これらのアルゴリズムは、いずれも次のような利点があります。
自動終了するため、含むメンバーの数を考慮する必要がない。
重みが非常に小さいアンサンブルが生成されるため、アンサンブルのメンバーを安全に削除できる。
データの読み込み
ionosphere データ セットを読み込みます。
load ionosphereアンサンブル分類の作成
ionosphere データを分類するためのアンサンブルを、LPBoost、TotalBoost アルゴリズムを使用して作成します。また、比較する場合は AdaBoostM1 アルゴリズムを使用します。アンサンブルに使用するメンバーの数を把握するのは困難です。LPBoost と TotalBoost については、500 を使用します。比較する場合、AdaBoostM1 でも 500 を使用します。
ブースティング法の既定の弱学習器は、MaxNumSplits プロパティが 10 に設定されている決定木です。これらの木は、(最大分割が 1 である) 切り株より良く当てはまる傾向があり、過適合になる場合もあります。したがって、過適合を回避するため、アンサンブルの弱学習器として切り株を使用します。
rng('default') % For reproducibility T = 500; treeStump = templateTree('MaxNumSplits',1); adaStump = fitcensemble(X,Y,'Method','AdaBoostM1','NumLearningCycles',T,'Learners',treeStump); totalStump = fitcensemble(X,Y,'Method','TotalBoost','NumLearningCycles',T,'Learners',treeStump); lpStump = fitcensemble(X,Y,'Method','LPBoost','NumLearningCycles',T,'Learners',treeStump); figure plot(resubLoss(adaStump,'Mode','Cumulative')); hold on plot(resubLoss(totalStump,'Mode','Cumulative'),'r'); plot(resubLoss(lpStump,'Mode','Cumulative'),'g'); hold off xlabel('Number of stumps'); ylabel('Training error'); legend('AdaBoost','TotalBoost','LPBoost','Location','NE');
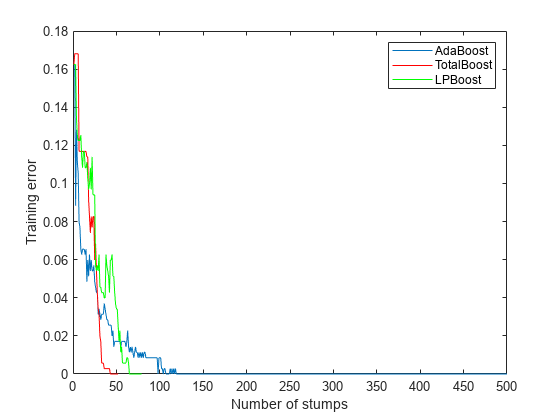
3 つのアルゴリズムからはいずれも、しばらく後に学習データについて正確な予測が得られます。
3 つのアンサンブルすべてのメンバー数を調べます。
[adaStump.NTrained totalStump.NTrained lpStump.NTrained]
ans = 1×3
500 52 68
AdaBoostM1 は 500 件のメンバーすべてを学習しました。他の 2 つのアルゴリズムの学習は早期に停止しています。
アンサンブルの交差検証
アンサンブルを交差検証して、アンサンブルの精度をより詳細に把握します。
cvlp = crossval(lpStump,'KFold',5); cvtotal = crossval(totalStump,'KFold',5); cvada = crossval(adaStump,'KFold',5); figure plot(kfoldLoss(cvada,'Mode','Cumulative')); hold on plot(kfoldLoss(cvtotal,'Mode','Cumulative'),'r'); plot(kfoldLoss(cvlp,'Mode','Cumulative'),'g'); hold off xlabel('Ensemble size'); ylabel('Cross-validated error'); legend('AdaBoost','TotalBoost','LPBoost','Location','NE');
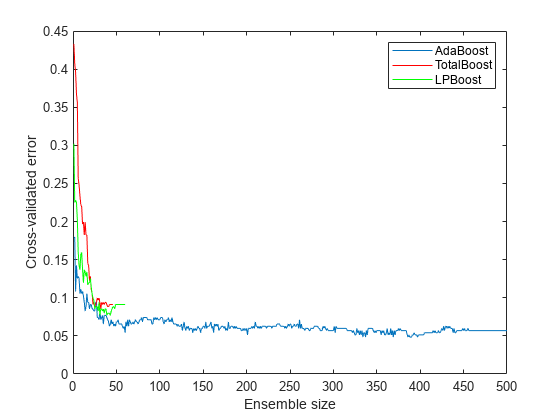
この結果から、アンサンブルのメンバー数が 50 であれば各ブースティング アルゴリズムで損失が 10% 以下になることがわかります。
アンサンブル メンバーの圧縮と削除
アンサンブルのサイズを小さくするには、圧縮してから removeLearners を使用します。ここで問題となるのは「学習器をいくつ削除すればよいか」です。このための測定方法の 1 つとして、交差検証損失曲線があげられます。また、圧縮後に LPBoost と TotalBoost の学習器の重みを調べるという方法もあります。
cada = compact(adaStump); clp = compact(lpStump); ctotal = compact(totalStump); figure subplot(2,1,1) plot(clp.TrainedWeights) title('LPBoost weights') subplot(2,1,2) plot(ctotal.TrainedWeights) title('TotalBoost weights')
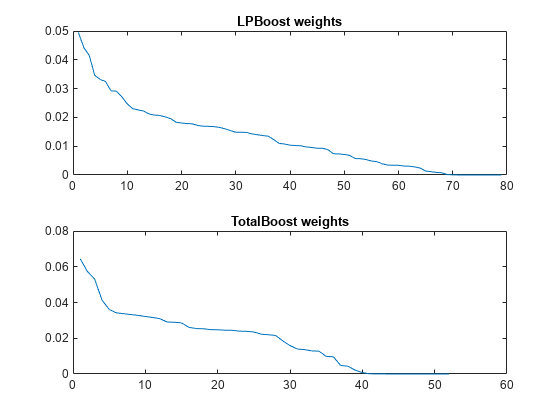
LPBoost と TotalBoost のいずれも、アンサンブルのメンバーの重みが無視できるようになるポイントが明確です。
重要でないアンサンブルのメンバーを削除します。
cada = removeLearners(cada,150:cada.NTrained); clp = removeLearners(clp,60:clp.NTrained); ctotal = removeLearners(ctotal,40:ctotal.NTrained);
これらの学習器を削除しても学習データに対するアンサンブルの精度に影響がないことを確認します。
[loss(cada,X,Y) loss(clp,X,Y) loss(ctotal,X,Y)]
ans = 1×3
0 0 0
圧縮後のアンサンブルのサイズを確認します。
s(1) = whos('cada'); s(2) = whos('clp'); s(3) = whos('ctotal'); s.bytes
ans = 569228
ans = 227374
ans = 151414
圧縮後のアンサンブルのサイズは、各アンサンブルのメンバーの数にほぼ比例します。
参考
fitcensemble | resubLoss | crossval | kfoldLoss | compact | loss | removeLearners