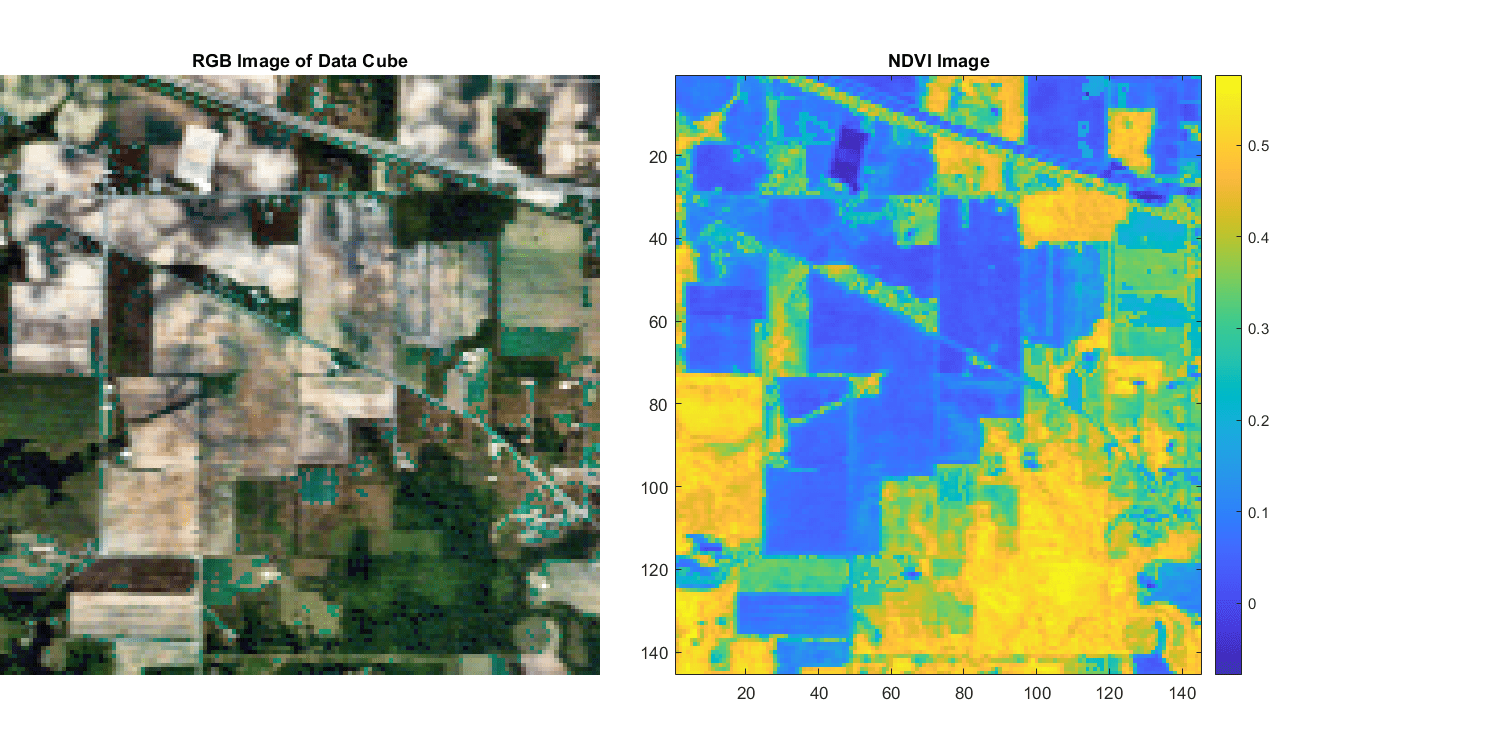
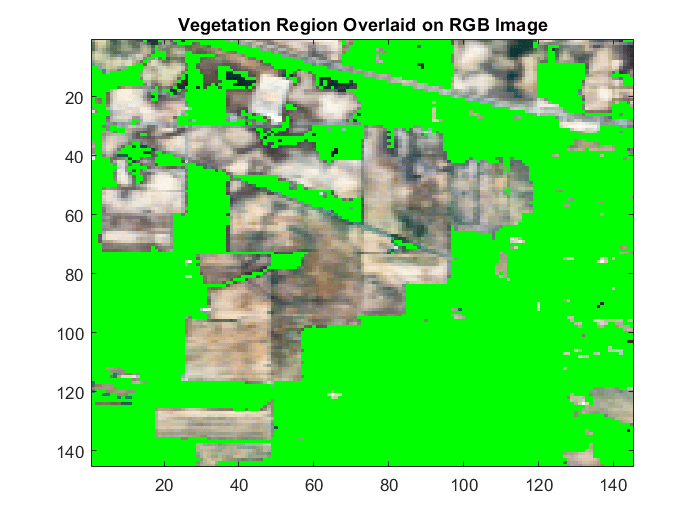
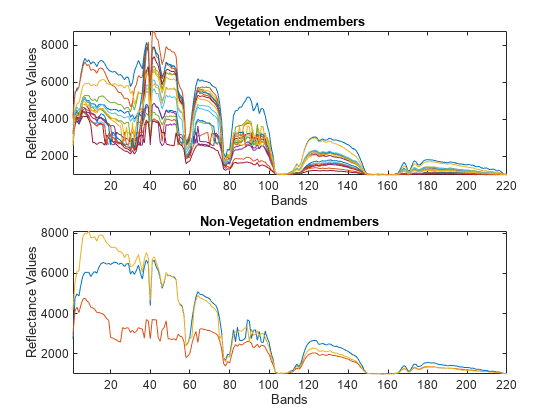
ndvi
正規化植生指標
説明
output = ndvi(spcube,BlockSize=blocksize)
この関数は、入力イメージを個別ブロックに分割して各ブロックを処理し、各ブロックの処理結果を連結して出力行列を形成します。スペクトル イメージは、大きすぎてそのままではシステム メモリに収まらない場合のある多次元データ セットです。そのため、関数 ndvi の実行中にシステムでメモリ不足が発生する場合があります。このような問題に直面した場合は、次の構文を使用してブロック処理を実行します。
たとえば、ndvi(spcube,BlockSize=[50 50]) は、入力イメージを 50 行 50 列のサイズのオーバーラップしないブロックに分割し、各ブロックのピクセルの NDVI 値を計算します。
メモ
この関数には、Hyperspectral Imaging Library for Image Processing Toolbox™ が必要です。Hyperspectral Imaging Library for Image Processing Toolbox はアドオン エクスプローラーからインストールできます。アドオンのインストールの詳細については、アドオンの入手と管理を参照してください。
Hyperspectral Imaging Library for Image Processing Toolbox は、MATLAB® Online™ および MATLAB Mobile™ によってサポートされないため、デスクトップの MATLAB が必要です。
例
入力引数
出力引数
参照
“Change Detection of Vegetation Cover by NDVI Technique on Catchment Area of the Panchet Hill Dam, India.” International Journal of Research in Geography 2, no. 3 (2016). https://doi.org/10.20431/2454-8685.0203002.
[1] Haboudane, D. “Hyperspectral Vegetation Indices and Novel Algorithms for Predicting Green LAI of Crop Canopies: Modeling and Validation in the Context of Precision Agriculture.” Remote Sensing of Environment 90, no. 3 (April 15, 2004): 337–52. https://doi.org/10.1016/j.rse.2003.12.013.
[2] “Change Detection of Vegetation Cover by NDVI Technique on Catchment Area of the Panchet Hill Dam, India.” International Journal of Research in Geography 2, no. 3 (2016). https://doi.org/10.20431/2454-8685.0203002.