植生スペクトルと非植生スペクトルの特定
この例では、以下を行う方法を説明します。
ハイパースペクトル関数用の超立方体として 2 次元スペクトル データを使用する。
関数
ndviを使用して植生スペクトルと非植生スペクトルを区別する。
この例には、Hyperspectral Imaging Library for Image Processing Toolbox™ が必要です。Hyperspectral Imaging Library for Image Processing Toolbox は、アドオン エクスプローラーからインストールできます。アドオンのインストールの詳細については、アドオンの取得と管理を参照してください。Hyperspectral Imaging Library for Image Processing Toolbox は、MATLAB® Online™ および MATLAB® Mobile™ でサポートされないため、デスクトップの MATLAB® が必要です。
2 次元スペクトル データの読み込み
インディアン パイン データセットの 20 個のエンドメンバーを含む 2 次元スペクトル データをワークスペースに読み込みます。
load("indian_pines_endmembers_20.mat")インディアン パイン データセットの各バンドの波長値をワークスペースに読み込みます。
load("indian_pines_wavelength.mat")ハイパースペクトル関数に使用するテスト データの準備
関数 reshape を使用して 2 次元スペクトル データを 3 次元ボリューム データに形状変更します。
[numSpectra,spectralDim] = size(endmembers); dataCube = reshape(endmembers,[numSpectra 1 spectralDim]);
3 次元ボリューム データ dataCube および波長情報 wavelength を関数 hypercube に指定して、大きさが 1 の次元で 3 次元 hypercube オブジェクトを作成します。
hcube = imhypercube(dataCube,wavelength);
NDVI の計算による植生スペクトルと非植生スペクトルの区別
hypercube オブジェクト内のスペクトルごとに NDVI 値を計算します。
ndviVal = ndvi(hcube);
植生スペクトルは一般的に 0 より大きい NDVI 値をもち、非植生スペクトルは一般的に 0 より小さい NDVI 値をもちます。しきい値処理を実行して植生スペクトルと非植生スペクトルを分離します。
index = ndviVal > 0;
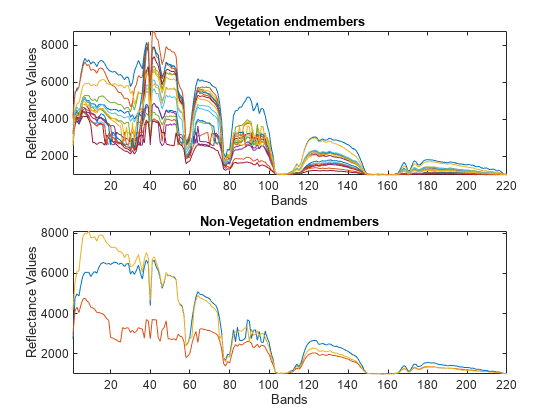
植生エンドメンバーと非植生エンドメンバーをプロットします。
subplot(2,1,1) plot(endmembers(index,:)') title("Vegetation endmembers") xlabel("Bands") ylabel("Reflectance Values") axis tight subplot(2,1,2) plot(endmembers(~index,:)') title("Non-Vegetation endmembers") xlabel("Bands") ylabel("Reflectance Values") axis tight
参考
hypercube | spectralMatch | ndvi