部分最小二乗回帰と主成分回帰
この例では、部分最小二乗回帰 (PLSR) と主成分回帰 (PCR) の適用方法を示し、これら 2 つの手法の有効性について確認します。PLSR と PCR は、どちらも、高相関性または共線性のある多数の予測子変数がある場合に、応答変数をモデル化するために使用する手法です。どちらの手法も、オリジナルの予測子変数の線形結合として、成分と呼ばれる新しい予測子変数を作成しますが、それらの成分の作成方法は異なります。PCR は、応答変数を一切考慮することなく、予測子変数での観測された変動を説明する成分を作成します。一方、PLSR は応答変数を考慮するため、少ない数の成分で応答変数を近似できるモデルにつながります。特定の用途について、これが最終的により倹約的なモデルを意味するかどうかは、コンテキストによります。
データの読み込み
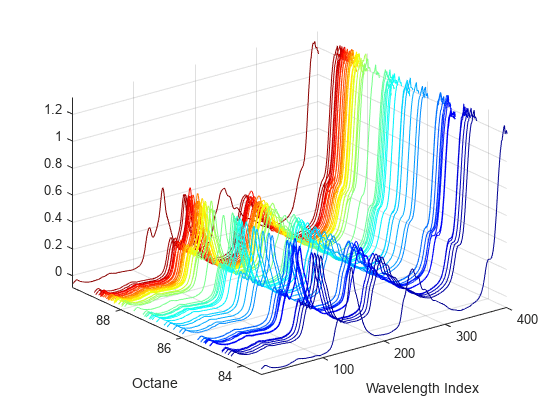
401 波長でのガソリンの 60 標本のスペクトル強度と、オクタン価で構成されるデータ セットを読み込みます。これらのデータは、Kalivas, John H., "Two Data Sets of Near Infrared Spectra," Chemometrics and Intelligent Laboratory Systems, v.37 (1997) pp.255-259 で説明しています。
load spectra whos NIR octane
Name Size Bytes Class Attributes NIR 60x401 192480 double octane 60x1 480 double
[dummy,h] = sort(octane); oldorder = get(gcf,'DefaultAxesColorOrder'); set(gcf,'DefaultAxesColorOrder',jet(60)); plot3(repmat(1:401,60,1)',repmat(octane(h),1,401)',NIR(h,:)'); set(gcf,'DefaultAxesColorOrder',oldorder); xlabel('Wavelength Index'); ylabel('Octane'); axis('tight'); grid on
2 つの成分を使用したデータの近似
10 個の PLS 成分と 1 つの応答を使用して PLSR モデルを当てはめるには、関数 plsregress を使用します。
X = NIR;
y = octane;
[n,p] = size(X);
[Xloadings,Yloadings,Xscores,Yscores,betaPLS10,PLSPctVar] = plsregress(...
X,y,10);データを適切に当てはめるために、成分は 10 個も必要ないかもしれませんが、この近似からの診断を使用すると、成分数の少ないより単純なモデルを選択することができます。たとえば、成分数を簡単に選択する 1 つの方法に、応答変数で記述された分散のパーセントを成分数の関数としてプロットする方法があります。
plot(1:10,cumsum(100*PLSPctVar(2,:)),'-bo'); xlabel('Number of PLS components'); ylabel('Percent Variance Explained in Y');

実際には、成分数の選択は慎重に行うことをお勧めします。たとえば、この例で後述する交差検証は広く使用される方法です。ここでは、2 つの成分を含む PLSR は観測された y の分散のほとんどを説明することが上のプロットで示されています。2 つの成分をもつモデルの近似した応答値を計算します。
[Xloadings,Yloadings,Xscores,Yscores,betaPLS] = plsregress(X,y,2); yfitPLS = [ones(n,1) X]*betaPLS;
次に、2 つの主成分を使用して PCR モデルを当てはめます。最初に、関数 pca を使用して X に対する主成分分析を実行します。この場合 PCR は、それらの 2 成分の応答変数の線形回帰にすぎません。2 つの変数の変動の量が大きく異なる場合は、標準偏差によって各変数を最初に正規化するのが普通ですが、ここではそれは行いません。
[PCALoadings,PCAScores,PCAVar] = pca(X,'Economy',false);
betaPCR = regress(y-mean(y), PCAScores(:,1:2));オリジナルのスペクトル データに関連して、PCR 結果を簡単に解釈できるようにするためには、オリジナルの中心化されていない変数の回帰係数に変換します。
betaPCR = PCALoadings(:,1:2)*betaPCR; betaPCR = [mean(y) - mean(X)*betaPCR; betaPCR]; yfitPCR = [ones(n,1) X]*betaPCR;
PLSR 近似と PCR 近似について、近似した応答と観測した応答をプロットします。
plot(y,yfitPLS,'bo',y,yfitPCR,'r^'); xlabel('Observed Response'); ylabel('Fitted Response'); legend({'PLSR with 2 Components' 'PCR with 2 Components'}, ... 'location','NW');
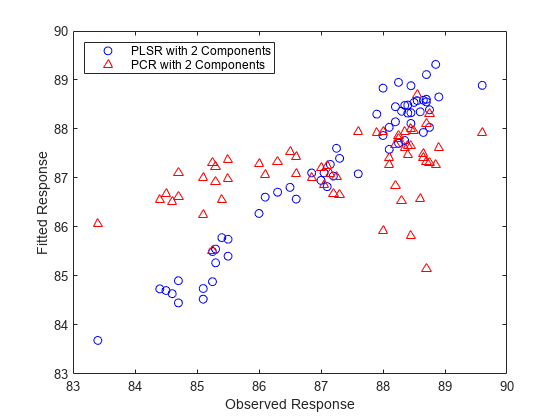
ある意味では、上のプロットでの比較は公平なものではありません。成分数 (2) は、2 成分をもつ PLSR モデルで応答予測が効果的に行われたことを確認してから選択したものです。PCR モデルをそれと同じ数の成分に制限する必要はありません。ただし、同じ数の成分を使った場合、y の近似は PLSR で行った方がはるかに効果的です。実際、上のプロットで近似した値の水平方向の分散を見ると、2 成分をもつ PCR は定数モデルを使用した場合とほとんど変わりません。それは、2 つの回帰からの決定係数によって裏付けられています。
TSS = sum((y-mean(y)).^2); RSS_PLS = sum((y-yfitPLS).^2); rsquaredPLS = 1 - RSS_PLS/TSS
rsquaredPLS = 0.9466
RSS_PCR = sum((y-yfitPCR).^2); rsquaredPCR = 1 - RSS_PCR/TSS
rsquaredPCR = 0.1962
2 つのモデルの予測力を比較するもう 1 つの方法として、両方で 2 つの予測子に対して応答変数をプロットする方法があります。
plot3(Xscores(:,1),Xscores(:,2),y-mean(y),'bo'); legend('PLSR'); grid on; view(-30,30);
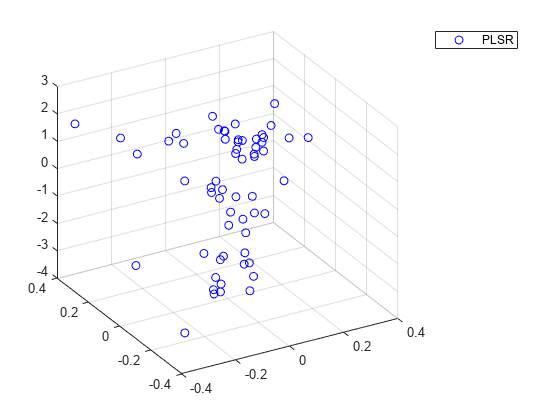
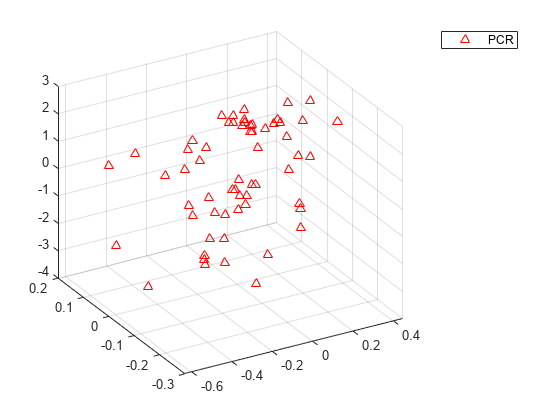
図を対話形式で回転できないので、わかりにくいかもしれませんが、上の PLSR プロットでは、点が平面上に近接して分散しています。一方、下の PCR プロットでは、点群が示されており、線形関係があることはほとんどわかりません。
plot3(PCAScores(:,1),PCAScores(:,2),y-mean(y),'r^'); legend('PCR'); grid on; view(-30,30);
2 つの PLS 成分は、観測された y の予測子としてははるかに優れていますが、次の図を見ると、PCR で使用された最初の 2 つの主成分と比べて、観測された X の分散が少ないことがわかります。
plot(1:10,100*cumsum(PLSPctVar(1,:)),'b-o',1:10, ... 100*cumsum(PCAVar(1:10))/sum(PCAVar(1:10)),'r-^'); xlabel('Number of Principal Components'); ylabel('Percent Variance Explained in X'); legend({'PLSR' 'PCR'},'location','SE');

PCR 曲線が一様に高いという事実は、2 成分の PCR での y の近似が PLSR に劣ることを示しています。PCR では、X を最も効果的に説明するように成分を構成します。その結果、最初の 2 つの成分は、観測された y の近似で重要なデータの情報を無視します。
3 つ以上の成分を使用した近似
PCR に成分が追加されると共に、オリジナル データ y の近似が改善されます。これは、X における重要な予測情報のほとんどが、いつかは主成分に含まれるようになるためです。たとえば、以下の図では、2 つの方法での残差の差は 10 成分を使用する場合のほうが 2 成分を使用する場合よりはるかに少なくなっています。
yfitPLS10 = [ones(n,1) X]*betaPLS10; betaPCR10 = regress(y-mean(y), PCAScores(:,1:10)); betaPCR10 = PCALoadings(:,1:10)*betaPCR10; betaPCR10 = [mean(y) - mean(X)*betaPCR10; betaPCR10]; yfitPCR10 = [ones(n,1) X]*betaPCR10; plot(y,yfitPLS10,'bo',y,yfitPCR10,'r^'); xlabel('Observed Response'); ylabel('Fitted Response'); legend({'PLSR with 10 components' 'PCR with 10 Components'}, ... 'location','NW');
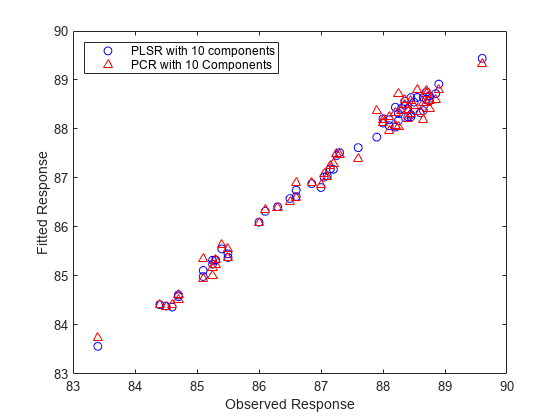
どちらのモデルも y にほぼ正確に当てはまりますが、PLSR の精度の方がわずかに上です。ただし、どちらのモデルについても 10 個の成分は任意で選択した数値です。
交差検証による成分数の選択
成分を複数にすることは、予測子変数での将来の観測からの応答を予測する場合に、予測誤差を減らすために役立ちます。多くの成分を使用すると、現在の観測されたデータの近似の効果は上がりますが、この方法は過適合につながります。現在のデータの近似が正確になりすぎると、他のデータのために一般化し難く、予測誤差について過度に楽観的な推定を提供するモデルが生成されます。
交差検証は、PLSR または PCR で成分を選択する方法として、統計的に優れた方法です。この方法では、モデルの当てはめと、予測誤差の推定に同じデータを再利用しないことで、データの過適合を防いでいます。したがって、予測誤差の推定は楽観的な下方バイアスにはなりません。
plsregress には、交差検証による平均二乗予測誤差 (MSEP) を推定するオプションがあります。この場合は、10 分割交差検証を使用します。
[Xl,Yl,Xs,Ys,beta,pctVar,PLSmsep] = plsregress(X,y,10,'CV',10);PCR の場合、crossval を単純な関数と組み合わせて、PCR の誤差の二乗和を計算することで、MSEP を予測できます。ここでも 10 分割交差検証を使用します。
PCRmsep = sum(crossval(@pcrsse,X,y,'KFold',10),1) / n;PLSR の MSEP 曲線は、2 個または 3 個の成分を使用すると、最大限の効果があることを示しています。一方、PCR で同じ予測精度を実現するには 4 つの成分が必要になります。
plot(0:10,PLSmsep(2,:),'b-o',0:10,PCRmsep,'r-^'); xlabel('Number of components'); ylabel('Estimated Mean Squared Prediction Error'); legend({'PLSR' 'PCR'},'location','NE');
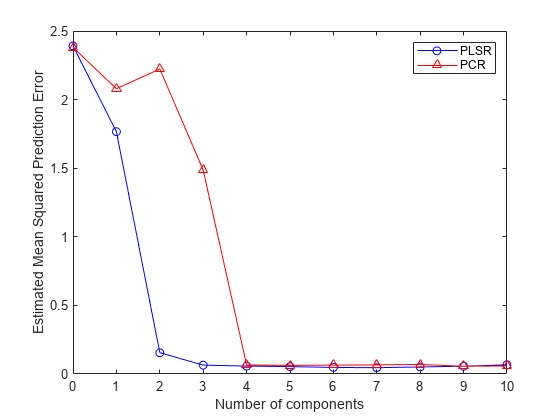
実際、PCR の 2 番目の成分は、モデルの予測誤差を拡大します。これは、その成分に含まれる予測子変数の組み合わせと y との相関が弱いことを示しています。この理由も、PCR では y ではなく X での偏差を説明するように成分を構成するためです。
モデルの倹約性
PCR で 3 成分の PLSR と同じ予測精度を達成するために 4 つの成分が必要だとすると、PLSR モデルの方が倹約的ということでしょうか?その答えはモデルのどの局面を考えているかによって異なります。
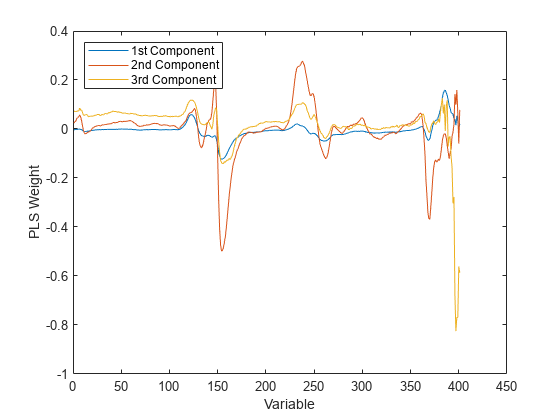
PLS の重みは、PLS 成分を定義するオリジナル変数の線形組み合わせです。つまり、PLSR の各成分がオリジナル変数にどれだけ依存するか、そしてその依存方向を示します。
[Xl,Yl,Xs,Ys,beta,pctVar,mse,stats] = plsregress(X,y,3); plot(1:401,stats.W,'-'); xlabel('Variable'); ylabel('PLS Weight'); legend({'1st Component' '2nd Component' '3rd Component'}, ... 'location','NW');
同様に、PCA 負荷量は、PCR の各成分がオリジナル変数にどれだけ依存するかを示します。
plot(1:401,PCALoadings(:,1:4),'-'); xlabel('Variable'); ylabel('PCA Loading'); legend({'1st Component' '2nd Component' '3rd Component' ... '4th Component'},'location','NW');

PLSR の場合も PCR の場合も、どの変数に最大の重み付けをしているかを調べることで、物理的に意味のある解釈を与えることができる可能性があります。たとえば、これらのスペクトル データでは、ガソリンに含有される化合物に関連して、強度ピークを解釈し、特定の成分についてその重みを観測するために、少数の成分を選択することができます。この観点からは、成分の数が少ないほど解釈が簡単になります。そして、PLSR では応答を正しく予測するために必要な成分の数が少ないので、より倹約的なモデルの生成につながります。
一方、PLSR と PCR の両方で、オリジナルの各予測子変数に対して 1 つの回帰係数、および 1 つの切片が生成されます。その意味では、使用した成分の数にかかわらず、どちらのモデルもすべての予測子に依存するので、どちらかがより倹約的であるとは言えません。具体的には、これらのデータでは、予測のために両方のモデルで 401 のスペクトル強度値が必要です。
しかし、最終的な目標は、オリジナルの変数セットを、1 つのサブセットに縮小し、応答を正確に予測することです。たとえば、PLS の重み、または PCA 負荷量を使用して、各成分への影響度が最も高い変数のみを選択できます。前に示したように、PCR モデル近似の一部の成分は、主に予測子変数の変動の把握のために使用することができ、また応答との相関が弱い変数に対する大きな重みを含むことができます。したがって、PCR は予測には不要な変数を維持する結果になることがあります。
この例で使用するデータについては、正確な予測のために PLSR と PCR で必要とされる成分の数の違いは大きくなく、PLS の重みと PCA の負荷量は同じ変数を選択するようです。他のデータでは必ずしもそのようにはなりません。