RegressionPartitionedGAM
説明
RegressionPartitionedGAM は、交差検証分割で学習を行った一般化加法モデルのセットです。kfold 関数の kfoldPredict、kfoldLoss、kfoldfun を 1 つ以上使用して、交差検証回帰の品質を評価します。
すべての kfold オブジェクト関数では、学習分割 (分割内) 観測値で学習させたモデルを使用して検証分割 (分割外) 観測値に対する応答を予測します。たとえば、データを 5 つに分割して交差検証を行うとします。ほぼ等しいサイズの 5 つのグループに各観測値が無作為に割り当てられます。"学習分割" にはグループのうち 4 つ (データの約 4/5) が含まれ、"検証分割" には他のグループ (データの約 1/5) が含まれます。この場合、交差検証は次のように処理されます。
(
CVMdl.Trained{1}に格納されている) 1 番目のモデルの学習には最後の 4 つのグループの観測値が使用され、1 番目のグループの観測値は検証用に確保されます。(
CVMdl.Trained{2}に格納されている) 2 番目のモデルの学習には、1 番目のグループと最後の 3 つのグループの観測値が使用されます。2 番目のグループの観測値は、検証用に確保されます。3 番目、4 番目および 5 番目のモデルに対しても同様に続けられます。
kfoldPredict を使用して検証する場合、i 番目のモデルを使用してグループ i の観測値について予測が計算されます。つまり、それぞれの観測値に対する応答は、その観測値を使用せずに学習させたモデルによって推定されます。
作成
RegressionPartitionedGAM モデルは 2 つの方法で作成できます。
オブジェクト関数
crossvalを使用して、GAM オブジェクトRegressionGAMから交差検証済みモデルを作成する。関数
fitrgamを使用し、名前と値の引数'CrossVal'、'CVPartition'、'Holdout'、'KFold'、'Leaveout'のいずれかを指定して、交差検証済みモデルを作成する。
プロパティ
GAM のプロパティ
応答変数の標準偏差のモデルを当てはめるかどうかを示すフラグ。false または true を指定します。標準偏差のモデルを当てはめるには、fitrgam の名前と値の引数 'FitStandardDeviation' として true を指定します。
IsStandardDeviationFit が true の場合は、kfoldPredict を使用して、予測子データ X で標準偏差を評価できます。この関数は、X で評価した応答変数の予測区間も返します。
データ型: logical
交差検証のプロパティ
この プロパティ は読み取り専用です。
交差検証済みモデルの名前。'GAM' として指定されます。
この プロパティ は読み取り専用です。
交差検証された分割の数。正の整数で指定します。
データ型: double
この プロパティ は読み取り専用です。
交差検証パラメーター値。オブジェクトを指定します。パラメーター値は、一般化加法モデルの交差検証に使用した名前と値の引数の値に対応します。推定されたパラメーターは ModelParameters に含まれません。
ドット表記を使用して ModelParameters のプロパティにアクセスできます。
この プロパティ は読み取り専用です。
Trained におけるモデルあたりの学習済みの木の数。次のフィールドをもつ構造体として指定します。
| フィールド | 値 |
|---|---|
PredictorTrees | 数値ベクトル — PredictorTrees の要素 i は、Trained 内のモデル i における線形項あたりの木の数を示します。 |
InteractionTrees | 数値ベクトル — InteractionTrees の要素 i は、Trained 内のモデル i における交互作用項あたりの木の数を示します。 |
データ型: struct
この プロパティ は読み取り専用です。
データを交差検証分割に分割する方法を含むデータ分割。cvpartition モデルとして指定します。
この プロパティ は読み取り専用です。
交差検証分割で学習させたコンパクトなモデル。CompactRegressionGAM モデル オブジェクトの cell 配列として指定されます。Trained には k 個のセルがあります。k は分割数です。
データ型: cell
他の回帰のプロパティ
この プロパティ は読み取り専用です。
カテゴリカル予測子のインデックス。正の整数のベクトルとして指定します。CategoricalPredictors には、対応する予測子がカテゴリカルであることを示すインデックス値が格納されます。インデックス値の範囲は 1 ~ p です。p はモデルの学習に使用した予測子の数です。どの予測子もカテゴリカルではない場合、このプロパティは空 ([]) になります。
データ型: double
この プロパティ は読み取り専用です。
X および Y に格納されている学習データ内の観測値の個数。数値スカラーを指定します。
データ型: double
この プロパティ は読み取り専用です。
予測子変数の名前。文字ベクトルの cell 配列を指定します。PredictorNames の要素の順序は、予測子名が学習データに現れる順序に対応します。
データ型: cell
この プロパティ は読み取り専用です。
応答変数名。文字ベクトルを指定します。
データ型: char
応答変換関数。"none" または関数ハンドルを指定します。ResponseTransform は、生の応答値を変換する方法を表します。
MATLAB® 関数やユーザー定義関数の場合は、関数ハンドルを入力します。たとえば、Mdl.ResponseTransform = @function を入力できます。ここで function は、元の応答の数値ベクトルを受け入れ、変換した応答が格納されている同じサイズの数値ベクトルを返します。
データ型: char | string | function_handle
この プロパティ は読み取り専用です。
モデルの学習に使用された観測値の重み。n 行 1 列の数値ベクトルとして指定されます。n は観測値の個数 (NumObservations) です。
W の要素の合計が 1 になるように、名前と値の引数 'Weights' で指定された観測値の重みが正規化されます。
データ型: double
この プロパティ は読み取り専用です。
モデルの交差検証に使用された予測子。数値行列または table として指定されます。
X の各行は 1 つの観測値に対応し、各列は 1 つの変数に対応します。
データ型: single | double | table
この プロパティ は読み取り専用です。
モデルの交差検証に使用された応答。数値ベクトルとして指定されます。
Y の各行は、X の対応する行の観測された応答を表します。
データ型: single | double
オブジェクト関数
kfoldPredict | 交差検証済み回帰モデル内の観測値に対する応答の予測 |
kfoldLoss | 交差検証された分割済みの回帰モデルの損失 |
kfoldfun | 回帰での関数の交差検証 |
例
fitrgam を使用して、交差検証済みの 10 分割 (既定の交差検証オプション) の GAM に学習させます。その後、kfoldPredict を使用し、学習分割観測値に対して学習をさせたモデルを使用して、検証分割観測値の応答を予測します。
carbig データ セットを読み込みます。このデータ セットには、1970 年代と 1980 年代初期に製造された自動車の測定値が格納されています。
load carbig予測子変数 (Acceleration、Displacement、Horsepower、および Weight) と応答変数 (MPG) を格納する table を作成します。
tbl = table(Acceleration,Displacement,Horsepower,Weight,MPG);
既定の交差検証オプションを使用して交差検証済み GAM を作成します。名前と値の引数 'CrossVal' を 'on' として指定します。
rng('default') % For reproducibility CVMdl = fitrgam(tbl,'MPG','CrossVal','on')
CVMdl =
RegressionPartitionedGAM
CrossValidatedModel: 'GAM'
PredictorNames: {'Acceleration' 'Displacement' 'Horsepower' 'Weight'}
ResponseName: 'MPG'
NumObservations: 398
KFold: 10
Partition: [1×1 cvpartition]
NumTrainedPerFold: [1×1 struct]
ResponseTransform: 'none'
IsStandardDeviationFit: 0
Properties, Methods
関数 fitrgam で 10 分割の RegressionPartitionedGAM モデル オブジェクト CVMdl が作成されます。交差検証時は、以下の手順が実行されます。
データを 10 個のセットに無作為に分割する。
各セットについて、そのセットを検証データとして予約し、他の 9 個のセットを使用してモデルに学習させる。
10 個のコンパクトな学習済みモデルを交差検証済みモデル オブジェクト
RegressionPartitionedGAMのTrainedプロパティに 10 行 1 列の cell ベクトルとして格納する。
既定の交差検証の設定は、名前と値の引数 'CVPartition'、'Holdout'、'KFold'、'Leaveout' を使用してオーバーライドできます。
kfoldPredict を使用して tbl の観測値の応答を予測します。それぞれの観測値に対する応答が、その観測値を使用せずに学習させたモデルを使用して予測されます。
yHat = kfoldPredict(CVMdl);
yHat は数値ベクトルです。最初の 5 つの予測応答を表示します。
yHat(1:5)
ans = 5×1
19.4848
15.7203
15.5742
15.3185
17.8223
回帰損失 (平均二乗誤差) を計算します。
L = kfoldLoss(CVMdl)
L = 17.7248
kfoldLoss から 10 個の分割についての平均二乗誤差の平均が返されます。
fitrgam を使用して回帰一般化加法モデル (GAM) に学習させ、crossval とホールドアウト オプションを使用して交差検証済み GAM を作成します。その後、kfoldPredict を使用し、学習分割観測値に対して学習をさせたモデルを使用して、検証分割観測値の応答を予測します。
patients データ セットを読み込みます。
load patients予測子変数 (Age、Diastolic、Smoker、Weight、Gender、SelfAssessedHealthStatus) と応答変数 (Systolic) を格納する table を作成します。
tbl = table(Age,Diastolic,Smoker,Weight,Gender,SelfAssessedHealthStatus,Systolic);
予測子の線形項が格納されている GAM に学習させます。
Mdl = fitrgam(tbl,'Systolic');Mdl は RegressionGAM モデル オブジェクトです。
30% のホールドアウト標本を指定して、モデルを交差検証します。
rng('default') % For reproducibility CVMdl = crossval(Mdl,'Holdout',0.3)
CVMdl =
RegressionPartitionedGAM
CrossValidatedModel: 'GAM'
PredictorNames: {'Age' 'Diastolic' 'Smoker' 'Weight' 'Gender' 'SelfAssessedHealthStatus'}
CategoricalPredictors: [3 5 6]
ResponseName: 'Systolic'
NumObservations: 100
KFold: 1
Partition: [1×1 cvpartition]
NumTrainedPerFold: [1×1 struct]
ResponseTransform: 'none'
IsStandardDeviationFit: 0
Properties, Methods
関数 crossval は、ホールドアウト オプションを使用して RegressionPartitionedGAM モデル オブジェクトの CVMdl を作成します。交差検証時は、以下の手順が実行されます。
30% のデータを無作為に選択して検証データとして確保し、残りのデータを使用してモデルに学習をさせる。
コンパクトな学習済みモデルを交差検証済みモデル オブジェクト
RegressionPartitionedGAMのTrainedプロパティに格納する。
名前と値の引数 'CrossVal'、'CVPartition'、'KFold' または 'Leaveout' を使用すると、別の交差検証の設定を選択できます。
kfoldPredict を使用して検証分割観測値の応答を予測します。この関数は、学習分割観測値に対して学習をさせたモデルを使用して、検証分割観測値の応答を予測します。この関数では、学習分割観測値に NaN が割り当てられます。
yFit = kfoldPredict(CVMdl);
検証分割観測値のインデックスを検出して、観測値のインデックス、観測された応答値および予測された応答値が格納されている table を作成します。table の最初の 8 行を表示します。
idx = find(~isnan(yFit)); t = table(idx,tbl.Systolic(idx),yFit(idx), ... 'VariableNames',{'Obseraction Index','Observed Value','Predicted Value'}); head(t)
Obseraction Index Observed Value Predicted Value
_________________ ______________ _______________
1 124 130.22
6 121 124.38
7 130 125.26
12 115 117.05
20 125 121.82
22 123 116.99
23 114 107
24 128 122.52
検証分割観測値の回帰誤差 (平均二乗誤差) を計算します。
L = kfoldLoss(CVMdl)
L = 43.8715
交差検証済みの 10 分割の一般化加法モデル (GAM) に学習させます。その後、kfoldLoss を使用して交差検証の累積回帰損失 (平均二乗誤差) を計算します。誤差を使用して、予測子 (予測子の線形項) あたりの最適な木の数と交互作用項あたりの最適な木の数を特定します。
代わりに、名前と値の引数OptimizeHyperparametersを使用して fitrgam の名前と値の引数の最適な値を特定することもできます。例については、OptimizeHyperparameters を使用した GAM の最適化を参照してください。
patients データ セットを読み込みます。
load patients予測子変数 (Age、Diastolic、Smoker、Weight、Gender、および SelfAssessedHealthStatus) と応答変数 (Systolic) を格納する table を作成します。
tbl = table(Age,Diastolic,Smoker,Weight,Gender,SelfAssessedHealthStatus,Systolic);
既定の交差検証オプションを使用して交差検証済み GAM を作成します。名前と値の引数 'CrossVal' を 'on' として指定します。また、5 つの交互作用項を含めるように指定します。
rng('default') % For reproducibility CVMdl = fitrgam(tbl,'Systolic','CrossVal','on','Interactions',5);
'Mode' を 'cumulative' として指定すると、関数 kfoldLoss は累積誤差を返します。これは、各分割に同じ数の木を使用して取得したすべての分割の平均誤差です。各分割の木の数を表示します。
CVMdl.NumTrainedPerFold
ans = struct with fields:
PredictorTrees: [300 300 300 300 300 300 300 300 300 300]
InteractionTrees: [76 100 100 100 100 42 100 100 59 100]
kfoldLoss では、最大で 300 個の予測子木と 42 個の交互作用木を使用して累積誤差を計算できます。
10 分割交差検証を行った累積平均二乗誤差をプロットします。'IncludeInteractions' を false として指定して、計算から交互作用項を除外します。
L_noInteractions = kfoldLoss(CVMdl,'Mode','cumulative','IncludeInteractions',false); figure plot(0:min(CVMdl.NumTrainedPerFold.PredictorTrees),L_noInteractions)
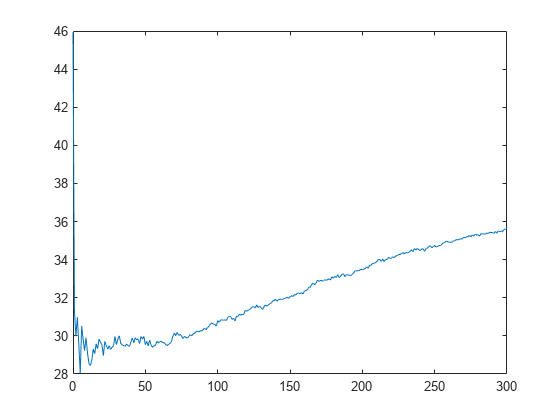
L_noInteractions の最初の要素は、切片 (定数) 項のみを使用して取得したすべての分割の平均誤差です。L_noInteractions の (J+1) 番目の要素は、切片項と各線形項の最初の J 個の予測子木を使用して取得した平均誤差です。累積損失をプロットすると、GAM の予測子木の数が増えるにつれて誤差がどのように変化するかを観察できます。
最小誤差とその最小誤差の達成時に使用された予測子木の数を調べます。
[M,I] = min(L_noInteractions)
M = 28.0506
I = 6
GAM に 5 個の予測子木が含まれるときに誤差が最小になっています。
線形項と交互作用項の両方を使用して累積平均二乗誤差を計算します。
L = kfoldLoss(CVMdl,'Mode','cumulative'); figure plot(0:min(CVMdl.NumTrainedPerFold.InteractionTrees),L)
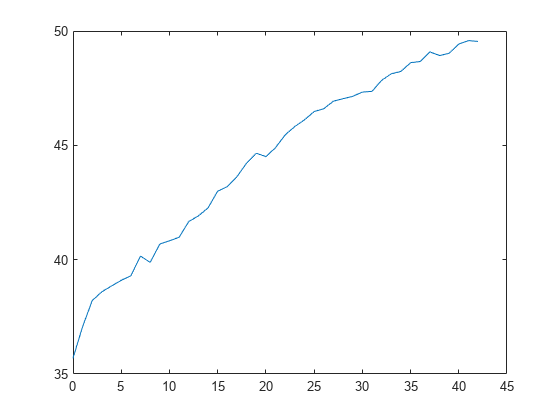
L の最初の要素は、切片 (定数) 項と各線形項のすべての予測子木を使用して取得したすべての分割の平均誤差です。L の (J+1) 番目の要素は、切片項、各線形項のすべての予測子木、および各交互作用項の最初の J 個の交互作用木を使用して取得した平均誤差です。プロットから、交互作用項を追加すると誤差が大きくなることがわかります。
予測子木の数が 5 個のときの誤差で問題がなければ、一変量の GAM にもう一度学習させ、交差検証を使用せずに 'NumTreesPerPredictor',5 と指定して予測モデルを作成できます。
詳細
一般化加法モデル (GAM) は、予測子の一変量および二変量の形状関数の和を使用して応答変数を説明する解釈可能なモデルです。
fitrgam では、各予測子および必要に応じて予測子の各ペアの形状関数としてブースティング木を使用するため、予測子と応答変数の間の非線形関係を取得できます。予測 (応答値) に対する個々の形状関数の寄与が十分に分離されるため、このモデルは解釈が容易です。
標準の GAM では、各予測子の一変量の形状関数を使用します。
y は応答変数で、平均 μ および標準偏差 σ をもつ正規分布に従います。g(μ) は恒等リンク関数、c は切片 (定数) 項です。fi(xi) は i 番目の予測子についての一変量の形状関数で、予測子の線形項のブースティング木 (予測子木) になります。
重要な交互作用項の二変量の形状関数をモデルに追加して、予測子同士の交互作用をモデルに含めることができます。
fij(xixj) は i 番目と j 番目の予測子についての二変量の形状関数で、予測子の交互作用項のブースティング木 (交互作用木) になります。
fitrgam は、重要な交互作用項を F 検定の p 値に基づいて特定します。詳細については、交互作用項の検出を参照してください。
fitrgam の 'FitStandardDeviation' として false (既定値) を指定した場合、fitrgam は平均 μ についてモデルに学習させます。'FitStandardDeviation' として true を指定した場合、fitrgam は標準偏差 σ について追加のモデルに学習させて、GAM オブジェクトの IsStandardDeviationFit プロパティを true に設定します。
バージョン履歴
R2021a で導入
MATLAB Command
You clicked a link that corresponds to this MATLAB command:
Run the command by entering it in the MATLAB Command Window. Web browsers do not support MATLAB commands.
Web サイトの選択
Web サイトを選択すると、翻訳されたコンテンツにアクセスし、地域のイベントやサービスを確認できます。現在の位置情報に基づき、次のサイトの選択を推奨します:
また、以下のリストから Web サイトを選択することもできます。
最適なサイトパフォーマンスの取得方法
中国のサイト (中国語または英語) を選択することで、最適なサイトパフォーマンスが得られます。その他の国の MathWorks のサイトは、お客様の地域からのアクセスが最適化されていません。
南北アメリカ
- América Latina (Español)
- Canada (English)
- United States (English)
ヨーロッパ
- Belgium (English)
- Denmark (English)
- Deutschland (Deutsch)
- España (Español)
- Finland (English)
- France (Français)
- Ireland (English)
- Italia (Italiano)
- Luxembourg (English)
- Netherlands (English)
- Norway (English)
- Österreich (Deutsch)
- Portugal (English)
- Sweden (English)
- Switzerland
- United Kingdom (English)