このページの内容は最新ではありません。最新版の英語を参照するには、ここをクリックします。
matrixProfile
構文
説明
行列プロファイルを返す
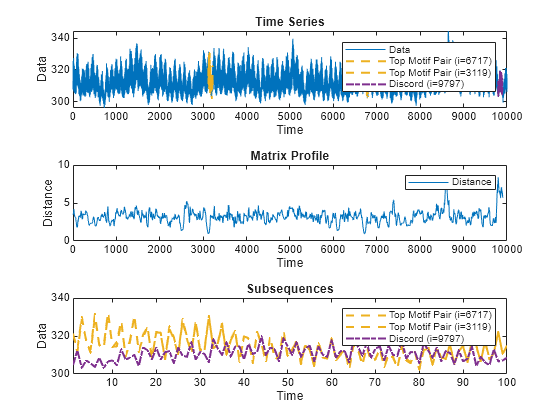
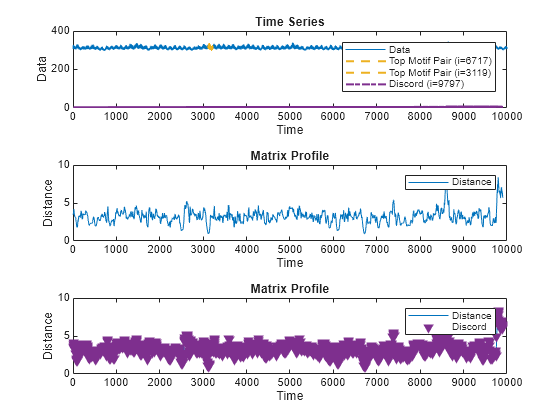
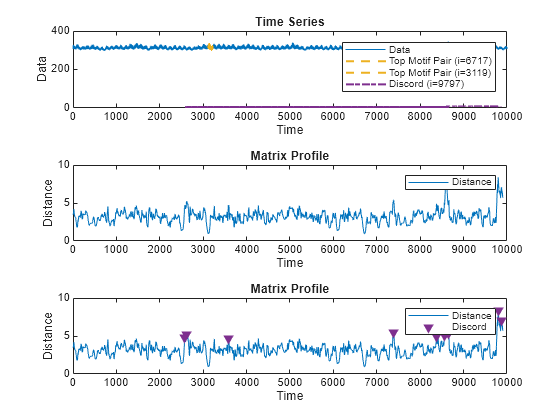
MP = matrixProfile(X,len)X の "行列プロファイル" を返します。行列プロファイルは、X の長さ len の各サブシーケンスとそれに最も近い近傍との間の最小の z 正規化ユークリッド距離のベクトルです。
Xがベクトルの場合、単一チャネルとして扱われます。Xが行列の場合、各列について行列プロファイルが別々に計算されます (多変量解)。
MP の上位の不一致およびモチーフの位置は、関数 findDiscord および findMotif を使用して検出できます。
[___] = matrixProfile(___,Name=Value) は、前述の構文の引数に加えて、1 つ以上の名前と値の引数を使用してオプションを指定します。たとえば、並列処理を使用するには、UseParallel を true に設定します。
行列プロファイルのプロット
matrixProfile(___) は、行列プロファイルの対話型プロットをプロットします。この構文は、前述の任意の入力引数の組み合わせで使用できます。
例
入力引数
名前と値の引数
出力引数
参照
[1] Yeh, Chin-Chia Michael, et al. “Matrix Profile I: All Pairs Similarity Joins for Time Series: A Unifying View That Includes Motifs, Discords and Shapelets.” 2016 IEEE 16th International Conference on Data Mining (ICDM), IEEE, 2016, pp. 1317–22. DOI.org (Crossref), https://doi.org/10.1109/ICDM.2016.0179.
[2] Zhu, Yan, et al. “Matrix Profile II: Exploiting a Novel Algorithm and GPUs to Break the One Hundred Million Barrier for Time Series Motifs and Joins.” 2016 IEEE 16th International Conference on Data Mining (ICDM), IEEE, 2016, pp. 739–48. DOI.org (Crossref), https://doi.org/10.1109/ICDM.2016.0085.