最大存在量分類を使用したハイパースペクトル イメージ解析
この例では、最大存在量分類 (MAC) を実行して、ハイパースペクトル イメージ内のさまざまな領域を識別する方法を示します。存在量マップは、ハイパースペクトル イメージ全体でエンドメンバー分布の特徴を表すものです。イメージ内の各ピクセルは、"ピュア ピクセル" の場合や "ミクセル" の場合があります。各ピクセルについて取得した存在量値のセットは、そのピクセルに存在する各エンドメンバーの割合を表しています。この例では、ピクセルごとの最大存在量値を見つけ、それを関連するエンドメンバー クラスに割り当てることでハイパースペクトル イメージ内のピクセルを分類します。
この例には、Hyperspectral Imaging Library for Image Processing Toolbox™ が必要です。Hyperspectral Imaging Library for Image Processing Toolbox は、アドオン エクスプローラーからインストールできます。アドオンのインストールの詳細については、アドオンの取得と管理を参照してください。Hyperspectral Imaging Library for Image Processing Toolbox は、MATLAB® Online™ および MATLAB® Mobile™ でサポートされないため、デスクトップの MATLAB® が必要です。
この例では、パヴィア大学のデータセットのデータ サンプルをテスト データとして使用します。このテスト データには、次のグラウンド トゥルース クラスを表す 9 つのエンドメンバーが格納されています。アスファルト、牧草地、砂利、樹木、塗装済み金属シート、露出土壌、瀝青、セルフ ブロック レンガ、影。
データの読み込みと可視化
テスト データを含む .mat ファイルをワークスペースに読み込みます。.mat ファイルには、ハイパースペクトル データ キューブを表す配列 paviaU、およびハイパースペクトル データから取得した 9 つのエンドメンバー シグネチャを表す行列 signatures が格納されています。データ キューブには、430 nm ~ 860 nm までの範囲の波長を持つ 103 個のスペクトル バンドがあります。幾何学的分解能は 1.3 メートル、各バンド イメージの空間分解能は 610 x 340 です。
load("paviaU.mat");
img = paviaU;
sig = signatures;波長範囲をスペクトル バンドの数で均等間隔に分けることで、各スペクトル バンドの中心波長を計算します。
wavelengthRange = [430 860]; numBands = 103; wavelength = linspace(wavelengthRange(1),wavelengthRange(2),numBands);
ハイパースペクトル データ キューブと中心波長を使用して hypercube オブジェクトを作成します。次に、ハイパースペクトル データから RGB イメージを推定します。RGB 出力のコントラストを改善するために、ContrastStretching パラメーター値を true に設定します。RGB イメージを可視化します。
hcube = imhypercube(img,wavelength);
rgbImg = colorize(hcube,Method="rgb",ContrastStretching=true);
figure
imshow(rgbImg)
テスト データには、9 つのグラウンド トゥルース クラスのエンドメンバー シグネチャが格納されています。sig の各列には、1 つのグラウンド トゥルース クラスのエンドメンバー シグネチャが格納されています。各エンドメンバーのクラス名と sig の対応する列を一覧表示する table を作成します。
num = 1:size(sig,2); endmemberCol = num2str(num'); classNames = ["Asphalt";"Meadows";"Gravel";"Trees";"Painted metal sheets";"Bare soil";... "Bitumen";"Self blocking bricks";"Shadows"]; table(endmemberCol,classNames,VariableName=["Column of sig";"Endmember Class Name"])
ans=9×2 table
Column of sig Endmember Class Name
_____________ ______________________
1 "Asphalt"
2 "Meadows"
3 "Gravel"
4 "Trees"
5 "Painted metal sheets"
6 "Bare soil"
7 "Bitumen"
8 "Self blocking bricks"
9 "Shadows"
エンドメンバーのシグネチャをプロットします。
figure plot(sig) xlabel("Band Number") ylabel("Data Values") ylim([400 2700]) title("Endmember Signatures") legend(classNames,Location="NorthWest")

存在量マップの推定
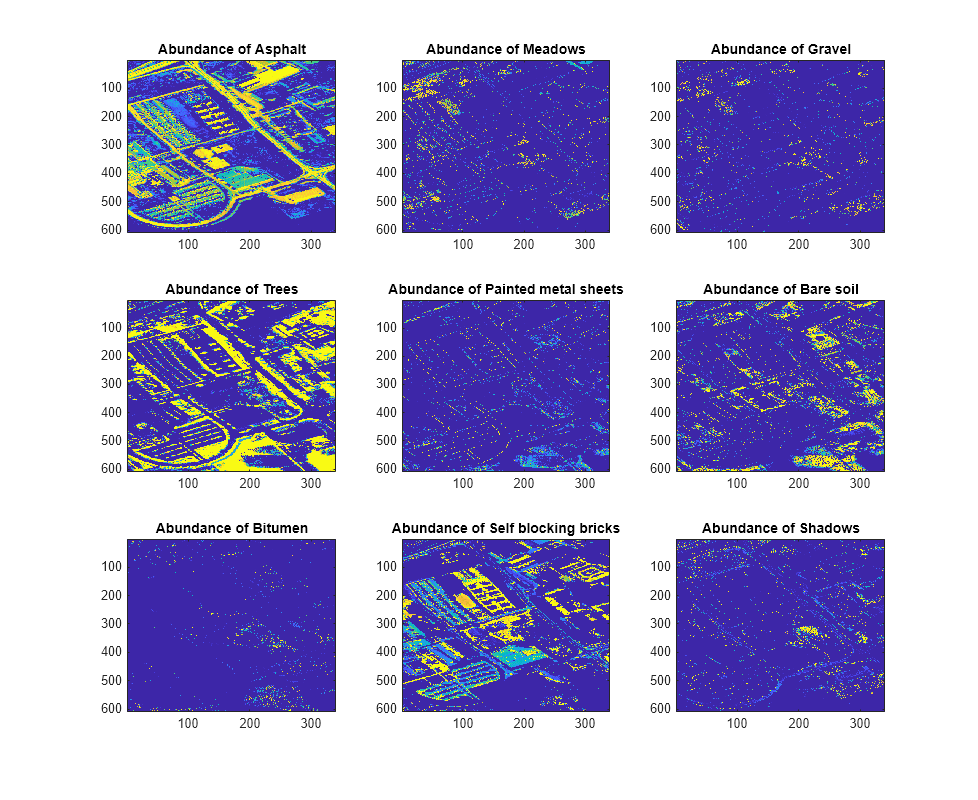
関数 estimateAbundanceLS を使用してエンドメンバーの存在量マップを作成し、完全制約付き最小二乗 (FCLS) 法を選択します。関数は、空間次元を入力データとして使用し、3 次元配列として存在量マップを出力します。各チャネルは、シグネチャの対応する列からのエンドメンバーの存在量マップです。この例では、入力データの空間次元は 610 × 340 で、エンドメンバーの数は 9 です。したがって、出力存在量マップのサイズは 610 x 340 x 9 となります。
abundanceMap = estimateAbundanceLS(hcube,sig,Method="fcls");存在量マップを表示します。
fig = figure(Position=[0 0 1100 900]); n = ceil(sqrt(size(abundanceMap,3))); for cnt = 1:size(abundanceMap,3) subplot(n,n,cnt) imagesc(abundanceMap(:,:,cnt)) title("Abundance of "+classNames{cnt}) hold on end hold off
最大存在量分類の実行
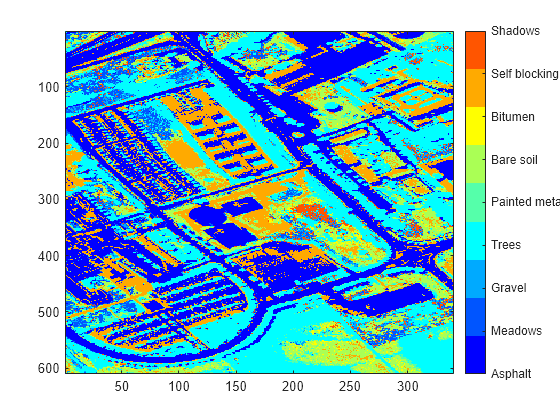
各ピクセルの最大存在量値のチャネル数を見つけます。各ピクセルについて返されるチャネル数は、そのピクセルの最大存在量値に関連するエンドメンバー シグネチャを含む sig 内の列に対応しています。最大存在量値で分類したピクセルの色分けイメージを表示します。
[~,matchIdx] = max(abundanceMap,[],3); figure imagesc(matchIdx) colormap(jet(numel(classNames))) colorbar(TickLabels=classNames)
分類された領域をセグメント化し、ハイパースペクトル データ キューブから推定された RGB イメージにそれぞれを重ね合わせます。
segmentImg = zeros(size(matchIdx)); overlayImg = zeros(size(abundanceMap,1),size(abundanceMap,2),3,size(abundanceMap,3)); for i = 1:size(abundanceMap,3) segmentImg(matchIdx==i) = 1; overlayImg(:,:,:,i) = imoverlay(rgbImg,segmentImg); segmentImg = zeros(size(matchIdx)); end
クラス名とともに、分類および重ね合わせたハイパースペクトル イメージの領域を表示します。イメージから、アスファルト、樹木、露出土壌、セルフ ブロック レンガの領域が正確に分類されていることが分かります。
figure(Position=[0 0 1100 900]); n = ceil(sqrt(size(abundanceMap,3))); for cnt = 1:size(abundanceMap,3) subplot(n,n,cnt); imagesc(uint8(overlayImg(:,:,:,cnt))); title("Regions Classified as "+classNames{cnt}) hold on end hold off

参考
estimateAbundanceLS | hypercube | colorize