細胞セグメンテーション用の事前学習済み Cellpose モデルの選択
この例では、事前学習済みの Cellpose モデルを使用して、顕微鏡イメージから細胞をセグメント化する方法を示します。
Cellpose ライブラリ [1]、[2] では、顕微鏡イメージ内の細胞セグメンテーションのための事前学習済みモデルをいくつか提供しています。各モデルとその学習データの詳細については、Cellpose ライブラリ ドキュメンテーションのモデルのページを参照してください。この例では、3 つの事前学習済みの Cellpose モデルを使用してイメージをセグメント化し、結果を視覚的に比較します。このアプローチを使用すると、独自のイメージに対して最適な事前学習済みモデルを見つけることができます。事前学習済みのモデルがイメージ データ セットに対して適切に機能しない場合は、転移学習を使用してカスタム Cellpose モデルに学習させることができます。学習の詳細については、Train Custom Cellpose Model (Medical Imaging Toolbox)の例を参照してください。
この例では、Medical Imaging Toolbox™ Interface for Cellpose Library が必要です。アドオン エクスプローラーから Medical Imaging Toolbox Interface for Cellpose Library をインストールできます。アドオンのインストールの詳細については、アドオンの取得と管理を参照してください。Medical Imaging Toolbox Interface for Cellpose Library には、Deep Learning Toolbox™ と Computer Vision Toolbox™ が必要です。
イメージの読み込み
イメージを読み取って表示します。この例では、メモリに収まるほど小さい単一チャネルのグレースケール イメージを使用します。独自のイメージを扱うときは、次のヒントを考慮してください。
Cellpose モデルは、入力として 2 次元強度イメージを必要とする。RGB またはマルチチャネル イメージがある場合は、
im2gray関数を使用してグレースケールに変換するか、imsplit関数を使用して特定のカラー チャネルを抽出できます。一般的に、細胞境界の輪郭が最も明瞭に表れるチャネルを選択します。イメージに核染色用の別のチャネルが含まれている場合は、segmentCells2D関数の名前と値の引数AuxiliaryChannelを使用して、セグメンテーション時に核イメージを指定することを検討してください。セグメント化するイメージが多数ある場合は、代表的なイメージまたはイメージのサブセットを選択する。完全なデータ セット内でラベル付けしたいタイプと形状の細胞が含まれているイメージを使用します。
スライド全体の顕微鏡イメージなどの大きなイメージを扱う場合は、代表的な関心領域を抽出する。完全なイメージ内で、ラベル付けしたい細胞タイプが含まれる領域を選択します。
blockedImageオブジェクトとgetRegionオブジェクト関数を使用して大きな顕微鏡イメージから領域を抽出する例については、部分イメージまたは低解像度を使用したブロック化されたイメージの効率的な処理 (Image Processing Toolbox)を参照してください。
img = imread("AT3_1m4_01.tif");
imshow(img)
細胞の平均直径の測定
正確な結果を確実に得るには、細胞のおおよその直径をピクセル単位で指定します。この例では、直径が 56 ピクセルであると想定しています。
averageCellDiameter = 56;
または、イメージ ビューアー (Image Processing Toolbox)アプリでおおよその直径を測定することもできます。次のコマンドを入力してアプリを開きます。
imageViewer(img)
アプリのツールストリップの [ビューアー] タブで、[距離の測定] をクリックします。メイン表示ペインで、測定する細胞の境界上の 1 点をクリックし、細胞を横切る線をドラッグして描きます。ボタンを放すと、アプリはその線にピクセル単位での距離測定値のラベルを付けます。線の端点または中央部分をドラッグして、端点の位置を変更したり、線全体を移動したりできます。次のイメージは、アプリ内での複数の距離測定の例を示しています。

Cellpose モデルを使用したイメージのセグメント化
このセクションでは、3 つの事前学習済み Cellpose ライブラリ モデル (cyto、cyto2、および nuclei) の結果を比較します。
Cellpose モデルの構成
各オブジェクトの Model プロパティを指定し、各モデルに対してcellpose (Medical Imaging Toolbox)オブジェクトを作成します。特定のモデルのオブジェクトを初めて作成すると、MATLAB® によって Cellpose ライブラリからモデルがダウンロードされ、ローカル コピーが保存されます。既定では、MATLAB はuserpath関数によって返されるフォルダー内の cellposeModels という名前のフォルダーにモデルを保存します。オブジェクトの作成時に ModelFolder プロパティを指定することで、保存場所を変更できます。
cpCyto = cellpose(Model="cyto"); cpCyto2 = cellpose(Model="cyto2"); cpNuclei = cellpose(Model="nuclei");
細胞セグメンテーション ラベルの予測
segmentCells2D (Medical Imaging Toolbox)オブジェクト関数を使用し、3 つのモデルのそれぞれを使用してイメージをセグメント化します。
まず、細胞しきい値とフロー誤差しきい値の値を指定します。細胞しきい値は通常 -6 ~ 6 の範囲で、フロー誤差しきい値は通常 0.1 ~ 3 の範囲です。この例では、より多くの検出を優先するようにこれらの値を設定します。
cellThreshold = -6; flowThreshold = 3;
各モデルについて、ラベルの予測を行います。segmentCells2D オブジェクト関数は 2 次元ラベル イメージを返します。このイメージでは、モデルによって検出された各細胞が異なる数値ピクセル値をもちます。
labels1 = segmentCells2D(cpCyto,img, ... ImageCellDiameter=averageCellDiameter, ... CellThreshold=cellThreshold, ... FlowErrorThreshold=flowThreshold); labels2 = segmentCells2D(cpCyto2,img, ... ImageCellDiameter=averageCellDiameter, ... CellThreshold=cellThreshold, ... FlowErrorThreshold=flowThreshold); labels3 = segmentCells2D(cpNuclei,img, ... ImageCellDiameter=averageCellDiameter, ... CellThreshold=cellThreshold, ... FlowErrorThreshold=flowThreshold);
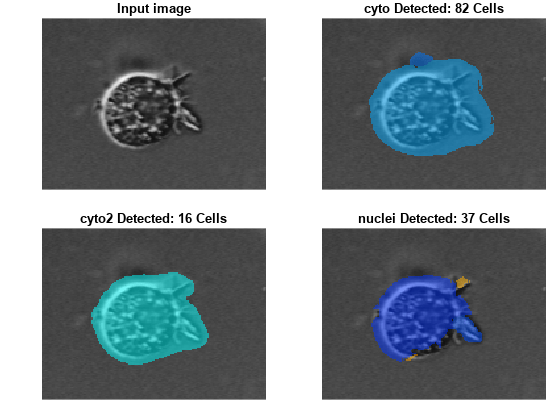
結果の比較
入力イメージと各モデルの結果を可視化します。各イメージのタイトルには、モデル名と検出された細胞の数が含まれます。
figure tiledlayout(2,2,TileSpacing="tight",Padding="tight") nexttile h1 = imshow(img); title("Input image") nexttile h2 = imshow(labeloverlay(img,labels1)); nl = numel(unique(labels1(:))) - 1; title(cpCyto.Model + " Detected: " + nl + " Cells") nexttile h3 = imshow(labeloverlay(img,labels2)); nl = numel(unique(labels2(:))) - 1; title(cpCyto2.Model + " Detected: " + nl + " Cells") nexttile h4 = imshow(labeloverlay(img,labels3)); nl = numel(unique(labels3(:))) - 1; title(cpNuclei.Model + " Detected: " + nl + " Cells") linkaxes([h1.Parent h2.Parent h3.Parent h4.Parent])

各イメージをズームインし、1 つの細胞について、各モデルの結果を比較します。一般的には、実際の細胞 1 つをラベル イメージ内の細胞 1 つとして検出するなど、正しい数の細胞を正確に検出するモデルを選択する必要があります。ズームインされたイメージに基づくと、このイメージ ファミリには cyto2 モデルが最も適しています。
xlim([200 370]) ylim([190 320])
モデルを選択したら、細胞しきい値やフロー誤差しきい値などのパラメーターを調整して境界の精度を改善できます。詳細については、Refine Cellpose Segmentation by Tuning Model Parameters (Medical Imaging Toolbox)の例を参照してください。
参照
[1] Stringer, Carsen, Tim Wang, Michalis Michaelos, and Marius Pachitariu. “Cellpose: A Generalist Algorithm for Cellular Segmentation.” Nature Methods 18, no. 1 (January 2021): 100–106. https://doi.org/10.1038/s41592-020-01018-x.
[2] Pachitariu, Marius, and Carsen Stringer. “Cellpose 2.0: How to Train Your Own Model.” Nature Methods 19, no. 12 (December 2022): 1634–41. https://doi.org/10.1038/s41592-022-01663-4.
参考
cellpose (Medical Imaging Toolbox) | segmentCells2D (Medical Imaging Toolbox) | segmentCells3D (Medical Imaging Toolbox) | trainCellpose (Medical Imaging Toolbox) | downloadCellposeModels (Medical Imaging Toolbox)
トピック
- Refine Cellpose Segmentation by Tuning Model Parameters
- Train Custom Cellpose Model
- Detect Nuclei in Large Whole Slide Images Using Cellpose