glmfit
一般化線形回帰モデルの当てはめ
構文
説明
b = glmfit(X,y,distr,Name,Value)'Constant','off' を指定するとモデルから定数項を省略できます。
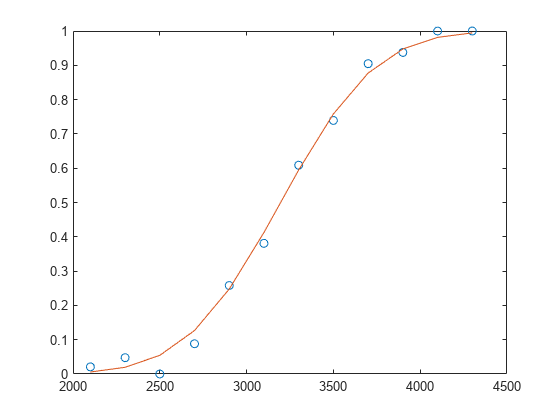
例
入力引数
名前と値の引数
出力引数
詳細
ヒント
glmfitは、Xまたはy内のNaNを欠損値として扱い、無視します。
代替機能
glmfit は、関数の出力引数のみが必要である場合、またはループ内でモデルの当てはめを複数回繰り返す場合に便利です。当てはめたモデルをさらに調べる必要がある場合は、fitglm または stepwiseglm を使用して一般化線形回帰モデル オブジェクト GeneralizedLinearModel を作成します。GeneralizedLinearModel オブジェクトは、glmfit より多くの機能を提供します。
当てはめたモデルを調べるには、
GeneralizedLinearModelのプロパティを使用します。オブジェクト プロパティには、係数推定値、要約統計量、当てはめ手法および入力データに関する情報が含まれています。応答の予測と、一般化線形回帰モデルの修正、評価および可視化を行うには、
GeneralizedLinearModelのオブジェクト関数を使用します。glmfitの出力に含まれる情報は、GeneralizedLinearModelのプロパティとオブジェクト関数を使用して取得できます。glmfitの出力GeneralizedLinearModelの同等の値bCoefficientsプロパティのEstimate列を参照してください。devDevianceプロパティを参照してください。statsコマンド ウィンドウのモデル表示を参照してください。統計量はモデルのプロパティ (
CoefficientCovariance、Coefficients、Dispersion、DispersionEstimated、およびResiduals) から取得できます。glmfitのstats.sDispersionプロパティの分散パラメーターは応答の分散のスケール係数です。したがって、stats.sはDispersionの値の平方根です。
参照
[1] Dobson, A. J. An Introduction to Generalized Linear Models. New York: Chapman & Hall, 1990.
[2] McCullagh, P., and J. A. Nelder. Generalized Linear Models. New York: Chapman & Hall, 1990.
[3] Collett, D. Modeling Binary Data. New York: Chapman & Hall, 2002.
拡張機能
バージョン履歴
R2006a より前に導入