MapReduce フレームワークと Hadoop を使用した大規模なイメージ セットの処理
この例では、Image Processing Toolbox™ と共に MATLAB® の MapReduce およびデータストアを使用して、大量のイメージに対して細胞カウント アルゴリズムを実行する方法を説明します。MapReduce とは、メモリに収まらないデータセットを解析するプログラミング手法です。また、この例では MATLAB Parallel Server™ を使用して、並列 MapReduce プログラムを Hadoop® クラスターで実行します。この例では、Hadoop クラスターにアルゴリズムを移動する前に、一部のイメージについてローカル システムでアルゴリズムをテストする方法を説明します。
サンプル データのダウンロード
Broad Bioimage Benchmark Collection から BBBC005v1 データセットをダウンロードします。このデータセットはテストや検証のために作られている注釈の付いた生体イメージ セットです。このイメージ セットでは合焦点および焦点外の合成イメージの例が提供されており、フォーカス メトリクスの検証に使用できます。データセットには約 2 万ファイルが格納されています。詳細については、こちらの Introduction to the Data Set を参照してください。
Linux® システムのシステム プロンプトで wget コマンドを使用し、BBBC データセットを格納している zip ファイルをダウンロードします。このコマンドを実行する前に、zip ファイル (1.8 GB) と解凍したイメージ (2.6 GB) を十分保持できるスペースがターゲットの場所にあることを確認してください。
wget https://data.broadinstitute.org/bbbc/BBBC005/BBBC005_v1_images.zip
Linux システムのシステム プロンプトで zip ファイルからファイルを解凍します。
unzip BBBC005_v1_images.zip
このデータセット内のイメージ ファイル名を確認してください。この名前は、各イメージについての有効な情報を含んだ特定の形式で構成されています。たとえば、BBBC005_v1_images/SIMCEPImages_A05_C18_F1_s16_w1.TIF というファイル名は、イメージに 18 個の細胞 (C18) が含まれており、焦点のブレのシミュレーションを行うために直径が 1、シグマが直径の 0.25 倍のガウス ローパス フィルターでフィルター処理された (F1) ことを示しています。w1 は使用された染色を識別します。たとえば、w1 染色を使用するデータセット内のイメージの数を特定します。
d = dir('C:\Temp\BBBCdata\BBBC005_v1_images\*w1*');
numel(d)ans = 9600
サンプル イメージでのアルゴリズムのテスト
イメージのバッチ処理アプリを使用して BBBC データセット内のファイルを表示し、少量のファイルのサブセットでアルゴリズムをテストします。この例はイメージ内の細胞をセグメント化する簡単なアルゴリズムをテストします (この例では、この細胞セグメンテーション アルゴリズムを変更したバージョンを使用して、MapReduce の実装で使用する細胞カウント アルゴリズムを作成します)。
イメージのバッチ処理アプリへのイメージ ファイルの読み込み
"イメージのバッチ処理" アプリを開きます。MATLAB のツールストリップの [アプリ] タブにある [イメージ処理とコンピューター ビジョン] セクションで、[イメージのバッチ処理] をクリックします。imageBatchProcessor コマンドを使用して、コマンド ラインからアプリを開くこともできます。
"イメージのバッチ処理" アプリで、[イメージのインポート] をクリックし、ダウンロードしたデータセットが格納されているフォルダーに移動します。
"イメージのバッチ処理" アプリでは、フォルダー内にあるイメージのサムネイルが左ペインに表示され、[入力イメージ] タブには現在選択中のイメージの高解像度バージョンが表示されます。イメージをいくつか表示して、データセットについての理解を深めます。

セグメンテーション関数の指定
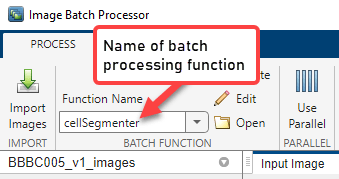
細胞セグメンテーション アルゴリズムを実装する関数の名前を指定します。既存の関数を指定するには、[関数名] フィールドに名前を入力するか、フォルダー アイコンをクリックして参照し、関数を選択します。新しいバッチ処理関数を作成するには、[作成] をクリックします。MATLAB® エディターでバッチ関数テンプレートが開きます。この例では、以下のイメージ セグメンテーション コードを含む新しい関数を作成します。[保存] をクリックしてバッチ関数を作成します。アプリが更新され、アプリのツールストリップの [バッチ関数] セクションに、作成した関数の名前が表示されます。
function imout = cellSegmenter(im) % A simple cell segmenter % Otsu thresholding bw = imbinarize(im); % Show thresholding result in app imout = imfuse(im,bw); % Find area of blobs stats = regionprops('table',bw,{'Area'}); % Average cell diameter is about 33 pixels (based on random inspection) cellArea = pi*(33/2)^2; % Estimate cell count based on area of blobs cellsPerBlob = stats.Area/cellArea; cellCount = sum(round(cellsPerBlob)); disp(cellCount); end
サンプル イメージでのセグメンテーション関数のテスト
アプリに表示されたイメージのサムネイルを選択し、[選択対象を処理] をクリックしてアルゴリズムのテストを実行します。この例では、"w1" 染色 (ファイル名で識別可) を含むイメージのみを選択します。セグメンテーション アルゴリズムはこれらのイメージに最適です。
アルゴリズムの実行結果を調べ、セグメンテーション アルゴリズムでイメージ内の細胞の数が正確に検出されたことを確認します。イメージの名前には C の数字により細胞数が示されています。たとえば、SIMCEPImages_A05_C18_F1_s05_w1.TIF という名前のイメージには 18 個の細胞が含まれています。この個数とコマンド ラインに返されたサンプル イメージの結果を比較します。
MapReduce フレームワークでのアルゴリズムのローカル テスト
作成したセグメンテーション コードが 1 つのイメージで期待どおりに動作することを確認したら、大規模な処理の実行にあたり、小規模なテスト バージョンをローカル システムで設定します。大量のファイルで実行する前に、処理フレームワークをテストする必要があります。
イメージ データストアへのイメージ ファイルの読み込み
まず、関数imageDatastoreを使用して、イメージの小規模なサブセットを含むイメージ データストアを作成します。MapReduce は、データ ストアを使用して、メモリに収まる小さいチャンクごとにデータを処理します。イメージが格納されているフォルダーに移動し、イメージ データストアを作成します。cellSegmenter.m に実装される細胞セグメンテーション アルゴリズムは細胞体染色で最適に機能するため、ファイル名のインジケーターが w1 のファイルのみを選択します。
localimds = imageDatastore(fullfile('/your_data/broad_data/BBBC005_v1-images','*w1*'));
ファイル名に "w1" が含まれるファイルに限定して選択する場合でも、イメージ データストアにはまだ 9000 個を超えるファイルが含まれています。データセットに含まれる大量のファイルから 100 個ごとにファイルを選択し、イメージのリストのサブセットを絞り込みます。
localimds.Files = localimds.Files(1:100:end);
Hadoop シーケンス ファイルへのサンプル セットの再パッケージ化
イメージ データストアを作成したら、イメージのサブセットのサンプルを、Hadoop クラスターで使用される形式である Hadoop シーケンス ファイルに変換します。この手順は、あるストレージ形式から別の形式にデータを変更するだけでデータ値は変更されません。シーケンス ファイルの詳細については、MapReduce 入門を参照してください。
イメージ データストアを Hadoop シーケンス ファイルに変換するには、関数 mapreduce に渡す "map" 関数および "reduce" 関数を作成します。イメージ ファイルを Hadoop シーケンス ファイルに変換するには、map 関数をノーオペレーション関数にする必要があります。この例では、map 関数はファイル名をキーとして使用し、イメージ データをそのまま単純に保存します。
function identityMap(data, info, intermKVStore) add(intermKVStore, info.Filename, data); end
reduce 関数を作成します。この関数は、イメージ ファイルをシーケンス ファイルのキーと値のデータストアに変換します。
function identityReduce(key, intermValueIter, outKVStore) while hasnext(intermValueIter) add(outKVStore, key, getnext(intermValueIter)); end end
mapreduce を呼び出し、map 関数と reduce 関数を渡します。この例では、まず関数 mapreducer を呼び出し、処理の場所を指定します。ローカル システムで設定をテストして処理を実行するには、0 を指定します
mapreducer(0);
ローカルで実行すると、mapreduce は MAT ファイルのキーと値のデータストアを作成します。
localmatds = mapreduce(localimds,@identityMap,@identityReduce,'OutputFolder',pwd);
MapReduce フレームワークのローカル テスト
イメージ ファイルのサブセットをテスト用に作成し、キーと値のデータストアに変換したら、いつでもアルゴリズムをテストできます。元の細胞セグメンテーション アルゴリズムを、細胞数を返すように変更します(この例で最初にアルゴリズムをテストした、イメージのバッチ処理アプリは、処理されたイメージのみを返し、細胞数などの値を返すことができません)。
細胞数を返し、イメージの表示を削除するように、細胞セグメンテーション関数を変更します。
function cellCount = cellCounter(im) % Otsu thresholding bw = imbinarize(im); % Find area of blobs stats = regionprops('table',bw,{'Area'}); % Average cell diameter is about 33 pixels (based on random inspection) cellArea = pi*(33/2)^2; % Estimate cell count based on area of blobs cellsPerBlob = stats.Area/cellArea; cellCount = sum(round(cellsPerBlob)); end
特定のイメージのエラー数を計算する map 関数を作成します。この関数はファイル名のコーディング (C の数字) からイメージの実際の細胞数を取得し、セグメンテーション アルゴリズムで返された細胞数と比較します。
function mapImageToMisCountError(data, ~, intermKVStore) % Extract the image im = data.Value{1}; % Call the cell counting algorithm actCount = cellCounter(im); % The original file name is available as the key fileName = data.Key{1}; [~, name] = fileparts(fileName); % Extract expected cell count and focus blur from the file name strs = strsplit(name, '_'); expCount = str2double(strs{3}(2:end)); focusBlur = str2double(strs{4}(2:end)); diffCount = abs(actCount-expCount); % Note: focus blur is the key add(intermKVStore, focusBlur, diffCount); end
フォーカス値ごとに細胞数のエラー平均値を計算する reduce 関数を作成します。
function reduceErrorCount(key, intermValueIter, outKVStore) focusBlur = key; % Compute the sum of all differences in cell count for this value of % focus blur count = 0; totalDiff = 0; while hasnext(intermValueIter) diffCount = getnext(intermvalueIter); count = count + 1; totalDiff = totalDiff+diffCount; end % Average meanDiff = totalDiff/count; add(outKVStore, focusBlue, meanDiff); end
mapreduce ジョブをローカル システムで実行します。
focusErrords = mapreduce(localmatds,@mapImageToMisCountError,@reduceErrorCount);
結果を収集します。
focusErrorTbl = readall(focusErrords);
誤差の平均値を取得します。
averageErrors = cell2mat(focusErrorTbl.Value);
ここで使用する簡単な細胞カウント アルゴリズムは、細胞または細胞群の平均の面積に依存します。焦点のブレが大きくなると、細胞の境界が拡散するため、面積も広がります。結果のプロットに見られるように、焦点のブレが大きくなると、誤差が増加するという結果が予測されます。
function plot_errors() bar(focusErrorTbl.Key, averageErrors); ha = gca; ha.XTick = sort(focusErrorTbl.Key); ha.XLim = [min(focusErrorTbl.Key)-2 max(focusErrorTbl.Key)+2]; title('Cell counting result on a test data set'); xlabel('Focus blur'); ylabel('Average error in cell count'); end

Hadoop クラスターでの MapReduce フレームワークの実行
データのサブセットに対してアルゴリズムの処理を検証したところで、Hadoop クラスターで全データセットに対してアルゴリズムを実行します。
Hadoop ファイル システムへのデータの読み込み
すべてのイメージ データを Hadoop ファイル システムに読み込み、次のシェル コマンドを使用して MapReduce フレームワークを Hadoop クラスターで実行します。このコマンドを実行するには、your_data をお使いのコンピューター上の場所に置き換えます。
hadoop fs -mkdir /user/broad_data/
hadoop fs -copyFromLocal /your_data/broad_data/BBBC005_v1_images /user/broad_data/BBBC005_v1_images
MATLAB Parallel Server クラスターへのアクセスの設定
MATLAB Parallel Server クラスターへのアクセスを設定します。このコマンドを実行するには、'your/hadoop/install' をお使いのコンピューター上の場所に置き換えます。
setenv('HADOOP_HOME','/your/hadoop/install');
cluster = parallel.cluster.Hadoop;
cluster.HadoopProperties('mapred.job.tracker') = 'hadoop01glnxa64:54311';
cluster.HadoopProperties('fs.default.name') = 'hdfs://hadoop01glnxa64:54310';
disp(cluster);
Mapreduce 実行環境のリモート クラスターへの変更
mapreduce 実行環境を、リモート クラスターを指すように変更します。
mapreducer(cluster);
すべてのイメージ データの Hadoop シーケンス ファイルへの変換
すべてのイメージ データを Hadoop シーケンス ファイルに変換します。これは、プロトタイピング用のイメージのサブセットを変換したときにローカル システムで実行した内容と同様のものです。前に使用した map 関数と reduce 関数を再利用できます。内部 Hadoop クラスターを使用します。
broadFolder = 'hdfs://hadoop01glnxa64:54310/user/broad_data/BBBC005_v1_images';
処理する細胞体染色 (w1) のファイルのみを選択します。
w1Files = fullfile(broadFolder,'*w1*.TIF');
これらのファイルすべてを表す ImageDatastore を作成します。
imageDS = imageDatastore(w1Files);
出力フォルダーを指定します。
seqFolder = 'hdfs://hadoop01glnxa64:54310/user/datasets/images/broad_data/broad_sequence';
イメージをキーと値のデータストアに変換します。
seqds = mapreduce(imageDS,@identityMap,@identityReduce,'OutputFolder',seqFolder);
データセット全体での細胞カウント アルゴリズムの実行
MapReduce フレームワークを使用して、Hadoop ファイル システムに格納されたデータセット全体に細胞カウント アルゴリズムを実行します。この場合、入力位置と出力位置が Hadoop ファイル システムになるという点が、ローカル システムでフレームワークを実行する場合と唯一異なります。
まず、誤差数の出力位置を指定します。
output = 'hdfs://hadoop01glnxa64:54310/user/broad_data/BBBC005_focus_vs_errorCount';
Mapreduce フレームワークでアルゴリズムを実行します。関数 tic および関数 toc を使用して、イメージ セットの処理にかかる時間を記録します。
tic
focusErrords = mapreduce(seqds,@mapImageToMisCountError,@reduceErrorCount,'OutputFolder',output);
toc
結果を収集します。
focusErrorTbl = readall(focusErrords);
averageErrors = cell2mat(focusErrorTbl.Value);
前と同様に結果をプロットします。
function reduceErrorCountAll(key, intermValueIter, outKVStore) bar(focusErrorTbl.Key, averageErrors); ha = gca; ha.XTick = sort(focusErrorTbl.Key); ha.XLim = [min(focusErrorTbl.Key)-2 max(focusErrorTbl.Key)+2]; title('Cell counting result on the entire data set'); xlabel('Focus blur'); ylabel('Average error in cell count'); end
