複素数値データを使用したネットワークの学習
この例では、1 次元畳み込みニューラル ネットワークを使用して複素数値波形の周波数を予測する方法を示します。
複素数値データをニューラル ネットワークに渡すには、入力層を使用して複素数値を実数部と虚数部に分割した後、ネットワーク内の後続の層にデータを渡します。入力層がこの方法で入力データを分割する場合、層は分割されたデータを追加のチャネルとして出力します。次の図は、複素数値データが畳み込みニューラル ネットワークをどのように流れるかを示しています。

複素数値データをネットワークへの入力として実数部と虚数部に分割するには、ネットワーク入力層の SplitComplexInputs オプションを 1 (true) に設定します。
この例では、複素数波形のデータ セットを使用して sequence-to-one 回帰ネットワークに学習させます。このデータ セットには、2 つのチャネルの異なる長さの複素数値の合成生成波形が 500 個含まれます。この例で学習させたネットワークは、波形の周波数を予測します。
シーケンス データの読み込み
サンプル データを ComplexWaveformData.mat から読み込みます。データは、numObservations 行 1 列のシーケンスの cell 配列です。ここで、numObservations はシーケンスの数です。各シーケンスは numChannels 行 numTimeSteps 列の複素数値配列です。ここで、numChannels はシーケンスのチャネル数、numTimeSteps はシーケンスに含まれるタイム ステップ数です。対応するターゲットは、波形の周波数から成る numObservations 行 numResponses 列の数値配列です。ここで、numResponses はターゲットのチャネル数です。
load ComplexWaveformData観測値の数を表示します。
numObservations = numel(data)
numObservations = 500
最初のいくつかのシーケンスのサイズ、および対応する周波数を表示します。
data(1:4)
ans=4×1 cell array
{2×157 double}
{2×112 double}
{2×102 double}
{2×146 double}
freq(1:4,:)
ans = 4×1
5.6232
2.1981
4.6921
4.5805
シーケンスのチャネル数を表示します。ネットワークに学習させるには、各シーケンスに同じ数のチャネルが含まれていなければなりません。
numChannels = size(data{1},1)numChannels = 2
応答の数 (ターゲットのチャネル数) を表示します。
numResponses = size(freq,2)
numResponses = 1
最初のいくつかのシーケンスをプロットに可視化します。
displayLabels = [ ... "Real Part" + newline + "Channel " + string(1:numChannels), ... "Imaginary Part" + newline + "Channel " + string(1:numChannels)]; figure tiledlayout(2,2) for i = 1:4 nexttile stackedplot([real(data{i}') imag(data{i}')],DisplayLabels=displayLabels) xlabel("Time Step") title("Frequency: " + freq(i)) end

学習用データの準備
検証用とテスト用のデータを残しておきます。データの 80% を含む学習セット、データの 10% を含む検証セット、およびデータの残りの 10% を含むテスト セットにデータを分割します。データを分割するには、この例にサポート ファイルとして添付されている関数 trainingPartitions を使用します。このファイルにアクセスするには、例をライブ スクリプトとして開きます。
[idxTrain,idxValidation,idxTest] = trainingPartitions(numObservations, [0.8 0.1 0.1]); XTrain = data(idxTrain); XValidation = data(idxValidation); XTest = data(idxTest); TTrain = freq(idxTrain); TValidation = freq(idxValidation); TTest = freq(idxTest);
短い学習シーケンスに対してネットワークが有効であることを確認するには、最短シーケンスの長さをネットワークのシーケンス入力層に渡します。最短の学習シーケンスの長さを計算します。
for n = 1:numel(XTrain) sequenceLengths(n) = size(XTrain{n},2); end minLength = min(sequenceLengths)
minLength = 76
1 次元畳み込みネットワーク アーキテクチャの定義
1 次元畳み込みニューラル ネットワーク アーキテクチャを定義します。
入力データの特徴の数に一致する入力サイズでシーケンス入力層を指定します。
入力データを実数部と虚数部に分割するために、入力層の
SplitComplexInputsオプションを1(true) に設定します。短い学習シーケンスに対してネットワークが有効であることを確認するために、
MinLengthオプションを最短の学習シーケンスの長さに設定します。畳み込み層のフィルター サイズが 5 である 1 次元畳み込み層、ReLU 層、およびレイヤー正規化層から成るブロックを 2 つ指定します。32 個のフィルターと 64 個のフィルターを最初と 2 番目の畳み込み層にそれぞれ指定します。どちらの畳み込み層に対しても、出力の長さが同じになるように入力を左パディングします (因果的パディング)。
畳み込み層の出力を単一のベクトルに減らすために、1 次元グローバル平均プーリング層を使用します。
予測する値の数を指定するために、応答の数に一致するサイズの全結合層を含めます。
filterSize = 5; numFilters = 32; layers = [ ... sequenceInputLayer(numChannels,SplitComplexInputs=true,MinLength=minLength) convolution1dLayer(filterSize,numFilters,Padding="causal") reluLayer layerNormalizationLayer convolution1dLayer(filterSize,2*numFilters,Padding="causal") reluLayer layerNormalizationLayer globalAveragePooling1dLayer fullyConnectedLayer(numResponses)];
学習オプションの指定
学習オプションを指定します。
Adam オプティマイザーを使用して学習させます。
入力データの形式が
"CTB"(チャネル、時間、バッチ) であることを指定します。学習を 250 エポック行います。より大きなデータ セットでは、良好な適合を実現させるために多くのエポックを学習させる必要がない場合があります。
検証に使用するシーケンスと応答を指定します。
検証損失が最も少ないネットワークを出力します。
学習プロセスをプロットに表示します。
詳細出力を無効にします。
options = trainingOptions("adam", ... InputDataFormats="CTB", ... MaxEpochs=250, ... ValidationData={XValidation, TValidation}, ... OutputNetwork="best-validation-loss", ... Plots="training-progress", ... Verbose=false);
ネットワークの学習
関数trainnetを使用してニューラル ネットワークに学習させます。回帰の場合は、平均二乗誤差損失を使用します。既定では、関数 trainnet は利用可能な GPU がある場合にそれを使用します。GPU を使用するには、Parallel Computing Toolbox™ ライセンスとサポートされている GPU デバイスが必要です。サポートされているデバイスの詳細については、GPU 計算の要件 (Parallel Computing Toolbox)を参照してください。そうでない場合、関数は CPU を使用します。実行環境を指定するには、ExecutionEnvironment 学習オプションを使用します。
net = trainnet(XTrain,TTrain,layers,"mse",options);
ネットワークのテスト
関数minibatchpredictを使用し、テスト データで予測を実行します。既定では、関数 minibatchpredict は利用可能な GPU がある場合にそれを使用します。データを左パディングし、形式が "CTB" (チャネル、時間、バッチ) であることを指定します。
YTest = minibatchpredict(net,XTest, ... SequencePaddingDirection="left", ... InputDataFormats="CTB");
最初のいくつかの予測をプロットに可視化します。
displayLabels = [ ... "Real Part" + newline + "Channel " + string(1:numChannels), ... "Imaginary Part" + newline + "Channel " + string(1:numChannels)]; figure tiledlayout(2,2) for i = 1:4 nexttile stackedplot([real(XTest{i}') imag(XTest{i}')], DisplayLabels=displayLabels); xlabel("Time Step") title("Frequency: " + YTest(i)) end

ヒストグラムで平均二乗誤差を可視化します。
figure histogram(mean((TTest - YTest).^2,2)) xlabel("Error") ylabel("Number of Predictions")
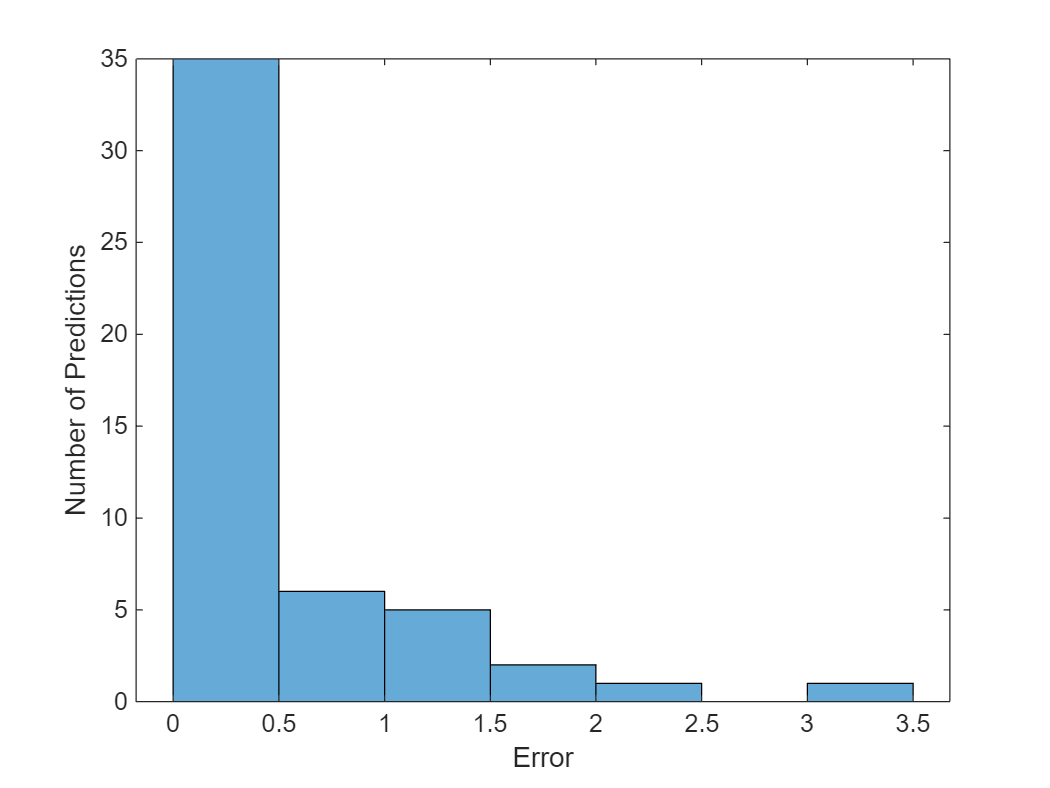
全体の平方根平均二乗誤差を計算します。
rmse = sqrt(mean((YTest-TTest).^2))
rmse = single
0.6751
ターゲット周波数に対する予測周波数をプロットします。
figure scatter(YTest,TTest,"+"); xlabel("Predicted Frequency") ylabel("Target Frequency") hold on m = min(freq); M = max(freq); xlim([m M]) ylim([m M]) plot([m M], [m M], "--")

参考
trainnet | trainingOptions | dlnetwork | testnet | minibatchpredict | predict | scores2label | convolution1dLayer | sequenceInputLayer