フーリエ ニューラル演算子を使用した PDE の求解
この例では、偏微分方程式 (PDE) の解を出力するフーリエ ニューラル演算子 (FNO) ニューラル ネットワークに学習させる方法を示します。
"ニューラル演算子" [1] は、関数空間の間をマッピングするニューラル ネットワークの一種です。たとえば、システムの初期条件が与えられたときに PDE の解を出力するように学習できます。"フーリエ ニューラル演算子" (FNO) [2] は、フーリエ変換を活用することで、これらのマッピングをより効率的に学習できるニューラル演算子です。具体的には、空間領域における入力関数 に対して、ニューラル ネットワークの層は、 となる変換 を学習します。ここで は のフーリエ変換です。これにより、FNO は周波数領域で動作できるようになります。ニューラル ネットワークを使用して PDE の解を予測すると、数値的な計算よりも高速に解を得られる場合があります。
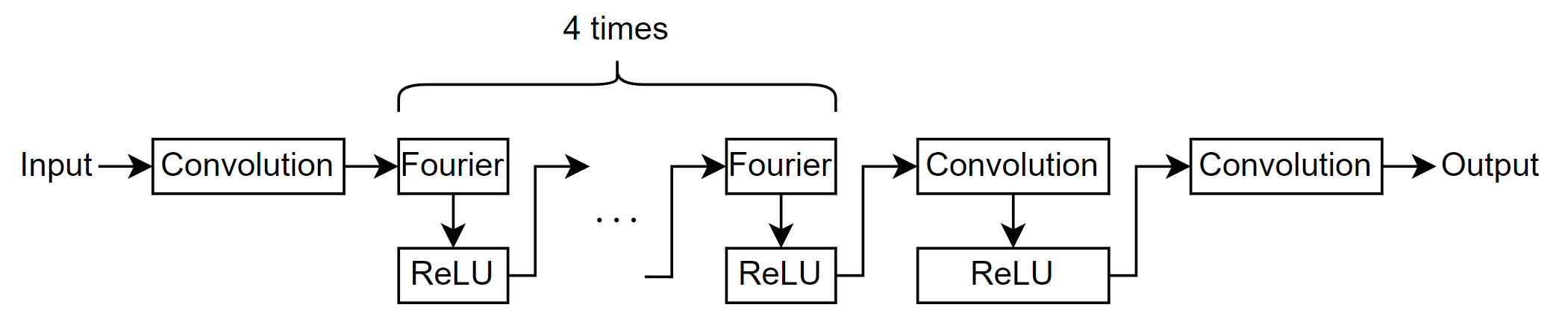
次の図は、FNO を通るデータの流れを示しています。
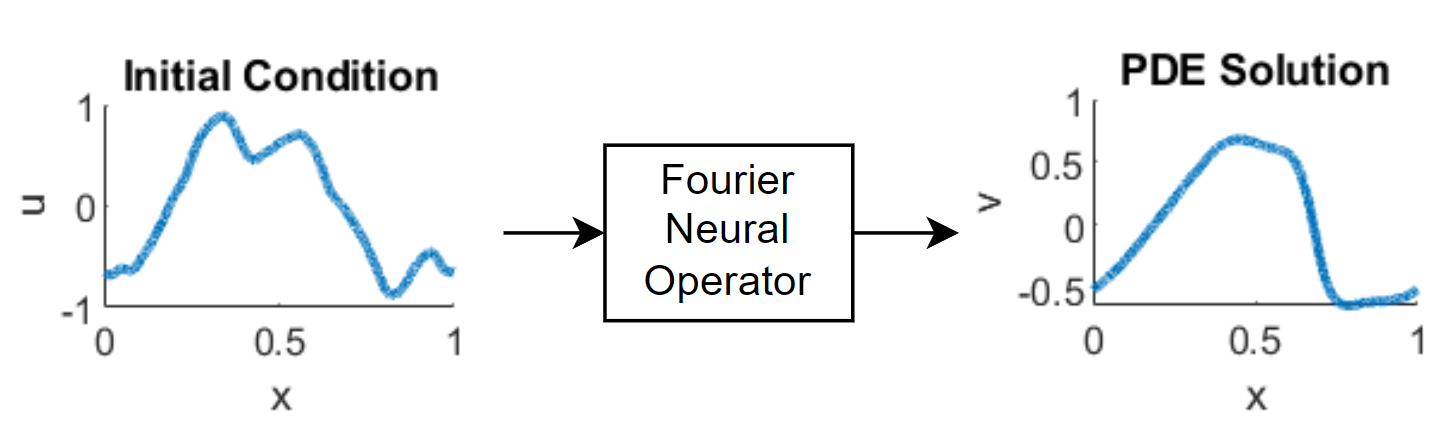
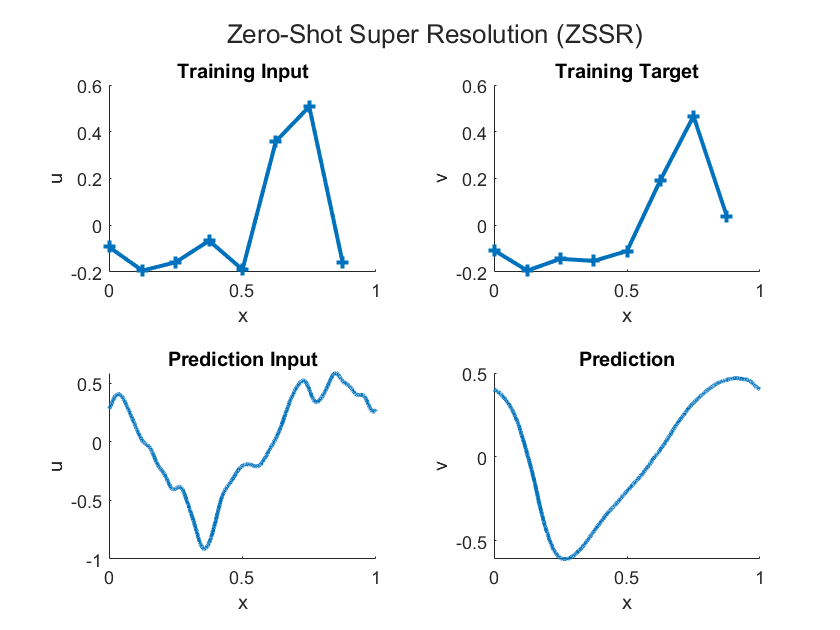
FNO は関数空間の間のマッピングを学習するため、学習データの解像度よりも高い解像度で出力関数を評価できます。この機能はゼロショット超解像 (ZSSR) として知られています。つまり、ZSSR は、追加の高解像度の例を必要とせずに、入力データと出力データの解像度を元の学習データを超えて向上させる機能です。次の図は、予測のための低解像度の学習データと高解像度の入力、およびそれらの出力の違いを示しています。
この例では、1 次元バーガーズ方程式の解を出力するように FNO に学習させます。
学習データの読み込み
1 次元バーガーズ方程式のデータを読み込みます。
バーガーズ方程式は、流体力学、非線形音響学、気体力学、交通流など、応用数学のさまざまな分野で用いられる PDE です。その PDE は次のようなものです。
ここで、 はレイノルズ数です。初期条件は次のとおりです。
この方程式は、空間領域全体にわたって周期的境界条件を課し、初期条件 から時刻 における解を示しています。
1 次元バーガーズ方程式のデータを burgers_data_R10.mat から読み込みます。このデータには、バーガーズ方程式の初期速度 と解 が含まれています。
dataFile = "burgers_data_R10.mat"; if ~exist(dataFile,"file") location = "https://ssd.mathworks.com/supportfiles/nnet/data/burgers1d/burgers_data_R10.mat"; websave(dataFile,location); end data = load(dataFile,"a","u"); u0 = data.a; u1 = data.u;
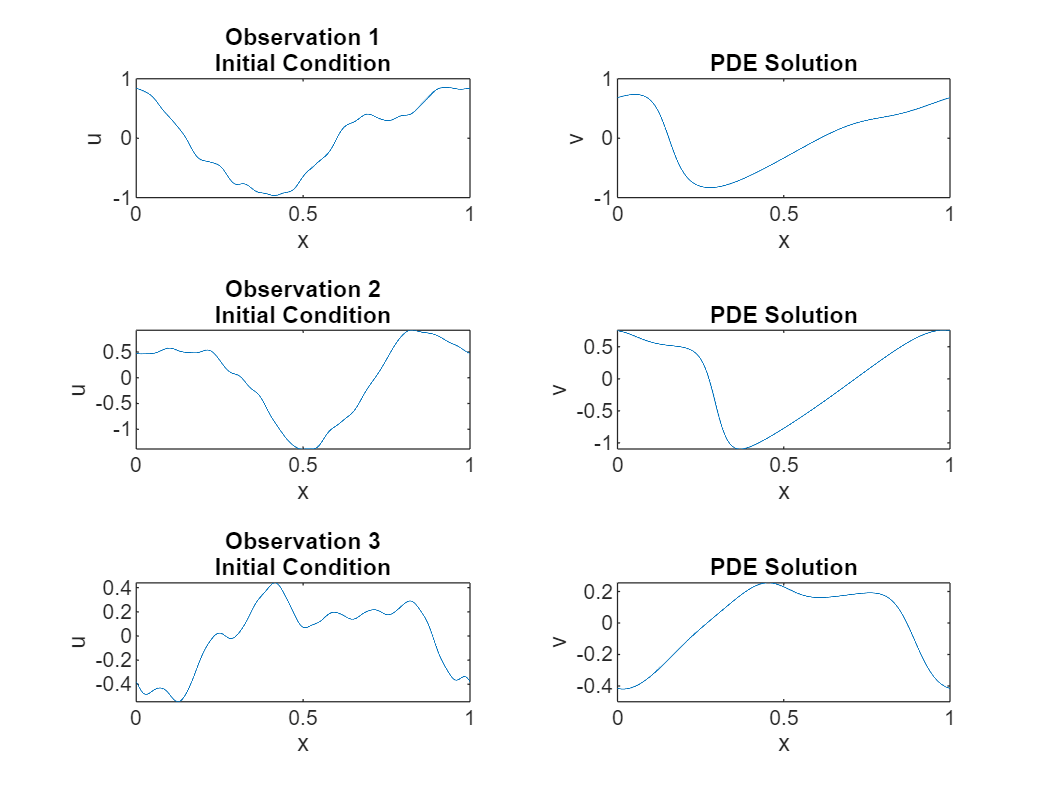
学習データをいくつか表示します。
numPlots = 3; gridSize = size(u0,2); figure tiledlayout(numPlots,2) for i = 1:numPlots nexttile plot(linspace(0,1,gridSize),u0(i,:)); title("Observation " + i + newline + "Initial Condition") xlabel("x") ylabel("u") nexttile plot(linspace(0,1,gridSize),u1(i,:)); title("PDE Solution") xlabel("x") ylabel("v") end
学習データの準備
この例にサポート ファイルとして添付されている trainingPartitions 関数を使用して、学習データとテスト データを分割します。この関数にアクセスするには、例をライブ スクリプトとして開きます。データの 80% は学習に使用し、10% は検証に使用し、残りの 10% はテストに使用します。
numObservations = size(u0,1); [idxTrain,idxValidation,idxTest] = trainingPartitions(numObservations,[0.8 0.1 0.1]); U0Train = u0(idxTrain,:); U1Train = u1(idxTrain,:); U0Validation = u0(idxValidation,:); U1Validation = u1(idxValidation,:); U0Test = u0(idxTest,:);
FNO での処理量を減らすには、学習データと検証データをダウンサンプリングして、グリッド サイズが 1024 になるようにします。
gridSizeDownsampled = 1024; downsampleFactor = floor(gridSize./gridSizeDownsampled); U0Train = U0Train(:,1:downsampleFactor:end); U1Train = U1Train(:,1:downsampleFactor:end); U0Validation = U0Validation(:,1:downsampleFactor:end); U1Validation = U1Validation(:,1:downsampleFactor:end);
FNO は、空間座標 (グリッド) に対応するチャネルをもつ入力データを必要とします。グリッドを作成し、入力データと連結します。
xMin = 0; xMax = 1; xGrid = linspace(xMin,xMax,gridSizeDownsampled); xGridTrain = repmat(xGrid, [numel(idxTrain) 1]); xGridValidation = repmat(xGrid, [numel(idxValidation) 1]); U0Train = cat(3,U0Train,xGridTrain); U0Validation = cat(3,U0Validation,xGridValidation);
学習セットと検証セットのサイズを表示します。1 番目、2 番目、および 3 番目の次元は、それぞれバッチ次元、空間次元、チャネル次元です。
size(U0Train)
ans = 1×3
1638 1024 2
size(U0Validation)
ans = 1×3
204 1024 2
ニューラル ネットワーク アーキテクチャの定義
ニューラル ネットワーク アーキテクチャを定義します。ネットワークは複数のフーリエ層と畳み込み層で構成されています。
フーリエ層の定義
周波数領域で動作するネットワーク層を作成する関数を定義します。
この層は、入力に畳み込みとスペクトル畳み込みを適用して処理し、それらの出力を合計します。次の図はこの層のアーキテクチャを示しています。
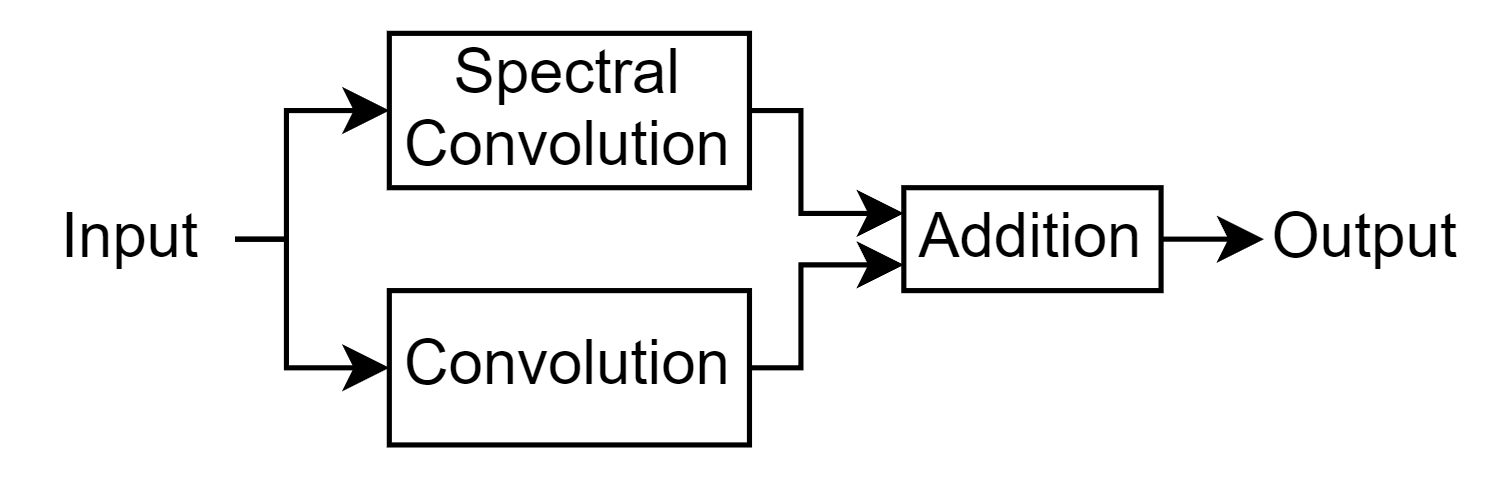
スペクトル畳み込み層は周波数領域で畳み込みを適用するもので、ネットワークが複雑な空間依存性を学習できるため、PDE の求解に特に有用です。フーリエ層は、スペクトル畳み込み演算を適用するために、この例にサポート ファイルとして添付されているカスタム層 spectralConvolution1dLayer を使用します。この層にアクセスするには、この例をライブ スクリプトとして開きます。
function layer = fourierLayer(spatialWidth,numModes,args) arguments spatialWidth numModes args.Name = "" end name = args.Name; net = dlnetwork; layers = [ identityLayer(Name="in") spectralConvolution1dLayer(spatialWidth,numModes,Name="specConv") additionLayer(2,Name="add")]; net = addLayers(net,layers); layer = convolution1dLayer(1,spatialWidth,Name="fc"); net = addLayers(net,layer); net = connectLayers(net,"in","fc"); net = connectLayers(net,"fc","add/in2"); layer = networkLayer(net,Name=name); end
ニューラル ネットワークの作成
ニューラル ネットワーク アーキテクチャを定義します。この例のネットワークは、直列に接続された複数の Fourier-ReLU ブロックで構成されています。
次のオプションを指定します。
フーリエ層には、空間の幅が 16 である 16 モードを指定します。
サイズ
[NaN spatialWidth 2]、形式"BSC"(バッチ、空間、チャネル) の入力層を使用します。ここで、spatialWidthはフーリエ層の空間の幅です。convolution-ReLU ブロックには、128 個の 1 行 1 列のフィルターを指定します。
最後の畳み込み層には、1 行 1 列のフィルターを 1 つ指定します。
numModes = 16;
spatialWidth = 64;
layers = [
inputLayer([NaN spatialWidth 2],"BSC");
convolution1dLayer(1,spatialWidth,Name="fc0")
fourierLayer(spatialWidth,numModes,Name="fourier1");
reluLayer
fourierLayer(spatialWidth,numModes,Name="fourier2")
reluLayer
fourierLayer(spatialWidth,numModes,Name="fourier3")
reluLayer
fourierLayer(spatialWidth,numModes,Name="fourier4")
reluLayer
convolution1dLayer(1,128)
reluLayer
convolution1dLayer(1,1)];学習オプション
学習オプションを指定します。オプションの中から選択するには、経験的解析が必要です。実験を実行してさまざまな学習オプションの構成を調べるには、実験マネージャーアプリを使用できます。
SGDM ソルバーを使用して学習させます。
初期学習率 0.001、低下係数 0.5 の区分的学習率スケジュールを使用して学習させます。
ミニバッチ サイズを 20 として 30 エポック学習させます。
すべてのエポックでデータをシャッフルします。
入力データにはバッチ次元、空間次元、チャネル次元に対応する次元があるため、入力データ形式を
"BSC"として指定します。学習の進行状況をプロットで監視し、検証データを指定します。
詳細出力を無効にします。
schedule = piecewiseLearnRate(DropFactor=0.5); options = trainingOptions("sgdm", ... InitialLearnRate=1e-3, ... LearnRateSchedule=schedule, ... MaxEpochs=30, ... MiniBatchSize=20, ... Shuffle="every-epoch", ... InputDataFormats="BSC", ... Plots="training-progress", ... ValidationData={U0Validation,U1Validation}, ... Verbose=false);
相対 損失関数の定義
予測とターゲットを入力として受け取り、相対的な 損失を返す relativeL2Loss 関数を作成します。相対 損失関数は、予測値とターゲットの差のユークリッド ノルムを使用してスケールを考慮し、それをターゲットのユークリッド ノルムで割る 損失関数のバリエーションです。
ここで、
は予測を表します。
はターゲットを表します。
は観測数を表します。
function loss = relativeL2Loss(Y,T) Y = stripdims(Y,"BSC"); T = stripdims(T,"BSC"); p = vecnorm(T-Y,2,2); q = vecnorm(T,2,2); loss = sum(p./q, 1); end
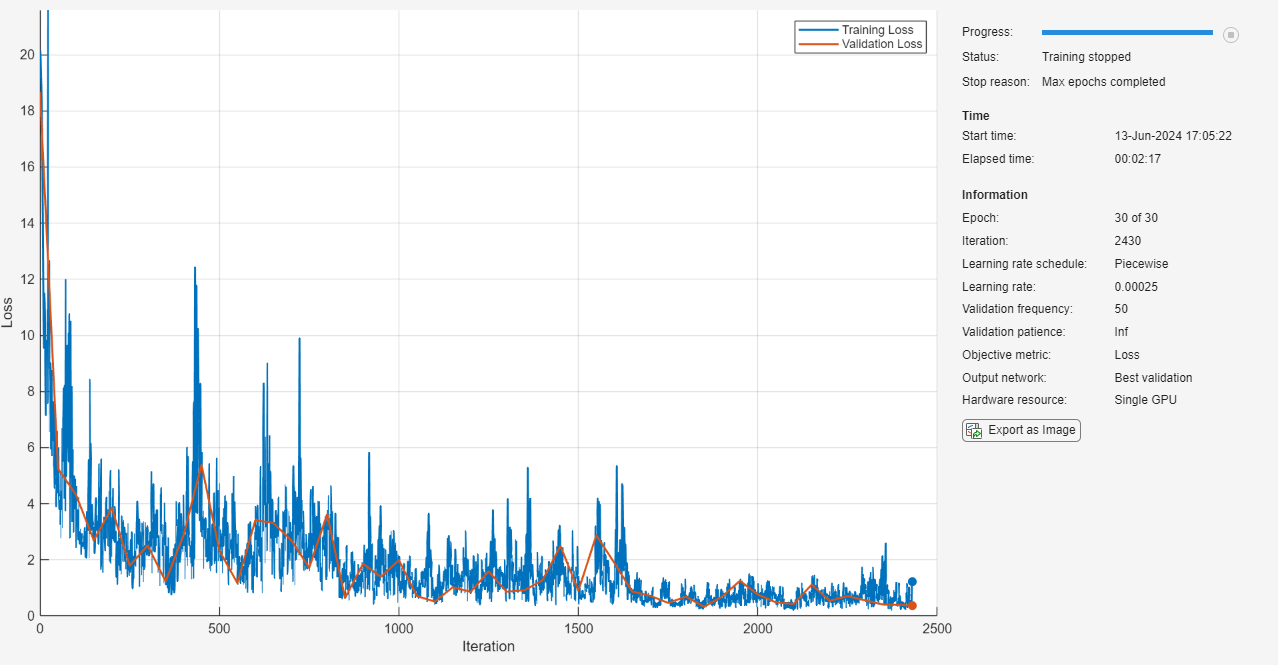
ネットワークの学習
関数trainnetを使用してニューラル ネットワークに学習させます。カスタム損失関数 relativeL2Loss を使用します。既定では、関数 trainnet は利用可能な GPU がある場合にそれを使用します。GPU を使用するには、Parallel Computing Toolbox™ ライセンスとサポートされている GPU デバイスが必要です。サポートされているデバイスの詳細については、GPU 計算の要件 (Parallel Computing Toolbox)を参照してください。そうでない場合、関数は CPU を使用します。実行環境を手動で選択するには、ExecutionEnvironment 学習オプションを使用します。
net = trainnet(U0Train,U1Train,layers,@relativeL2Loss,options);
ネットワークのテスト
真のラベルをもつホールドアウトされたテスト セットの予測を比較して、その平方根平均二乗誤差 (RMSE) を計算することで、モデルの予測パフォーマンスをテストします。
学習データの場合と同じ手順を使い、テスト データをダウンサンプリングして、グリッドを連結します。
xGridTest = repmat(xGrid, [numel(idxTest) 1]); U0Test = U0Test(:,1:downsampleFactor:end); U0Test = cat(3,U0Test,xGridTest); U1Test = u1(idxTest,:); U1Test = U1Test(:,1:downsampleFactor:end);
testnet関数を使用してニューラル ネットワークをテストします。既定では、testnet 関数は利用可能な GPU がある場合にそれを使用します。そうでない場合、関数は CPU を使用します。実行環境を手動で選択するには、testnet 関数の ExecutionEnvironment 引数を使用します。
rmseTest = testnet(net,U0Test,U1Test,"rmse")rmseTest = 0.0112
ZSSR の精度を評価するには、ダウンサンプリングせずにテスト データを使用してネットワークをテストします。
xGridZSSR = linspace(xMin,xMax,gridSize);
xGridTestZSSR = repmat(xGridZSSR, [numel(idxTest) 1]);
U0TestZSSR = u0(idxTest,:);
U0TestZSSR = cat(3,U0TestZSSR,xGridTestZSSR);
U1TestZSSR = u1(idxTest,:);
rmseTestZSSR = testnet(net,U0TestZSSR,U1TestZSSR,"rmse")rmseTestZSSR = 0.0113
新しいデータでの予測の実行
ニューラル ネットワークを使用して予測を行います。
新しいデータを読み込みます。例として、最初の 3 つのテスト観測値を使用します。
dataNew = u0(idxTest(1:3),:); numObservationsNew = size(dataNew,1);
グリッド サイズを計算し、それをデータと連結します。
gridSizeNew = size(dataNew,2); xGridNew = linspace(xMin,xMax,gridSizeNew); xGridNew = repmat(xGridNew, [size(dataNew,1) 1]); U0New = cat(3,dataNew,xGridNew);
関数minibatchpredictを使用して予測を行います。既定では、関数 minibatchpredict は利用可能な GPU がある場合にそれを使用します。そうでない場合、関数は CPU を使用します。実行環境を手動で選択するには、ExecutionEnvironment オプションを使用します。
Y = minibatchpredict(net,U0New);
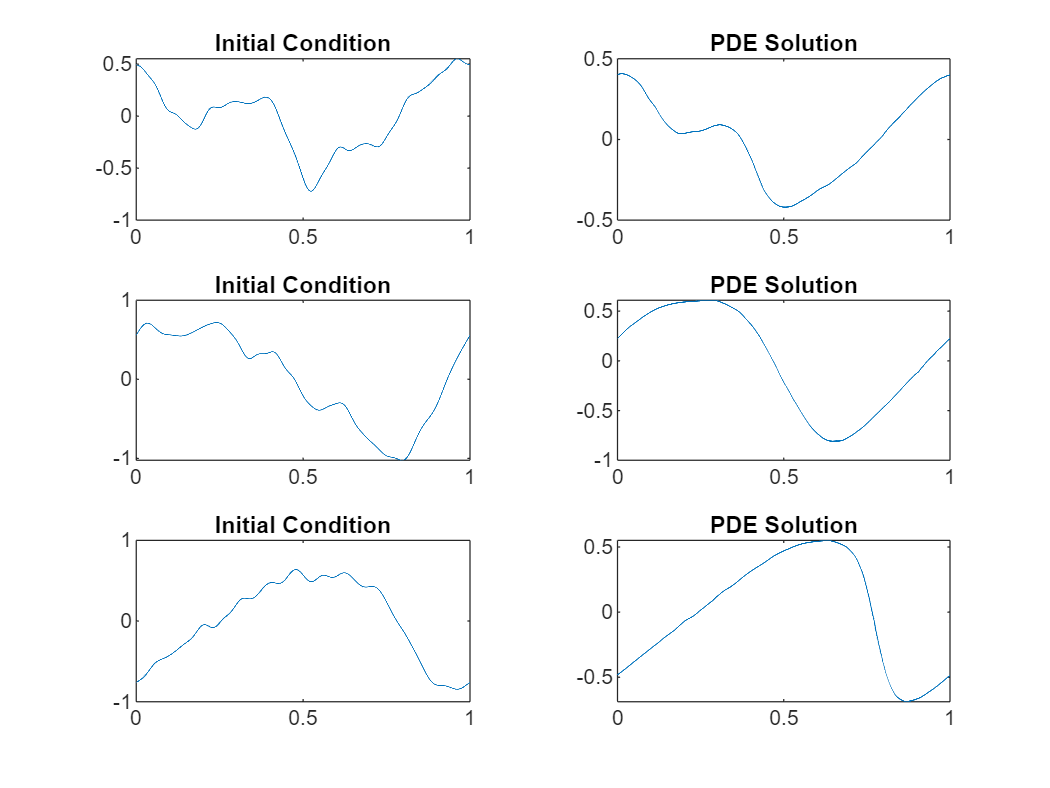
プロットで予測を可視化します。
figure tiledlayout("flow") for i = 1:numObservationsNew nexttile plot(xGridNew(i,:),dataNew(i,:)) title("Initial Condition") nexttile plot(xGridNew(i,:),Y(i,:)) title("PDE Solution") end
参考文献
Li, Zongyi, Nikola Kovachki, Kamyar Azizzadenesheli, Burigede Liu, Kaushik Bhattacharya, Andrew Stuart, and Anima Anandkumar. “Neural Operator: Graph Kernel Network for Partial Differential Equations.” arXiv, March 6, 2020. http://arxiv.org/abs/2003.03485.
Li, Zongyi, Nikola Kovachki, Kamyar Azizzadenesheli, Burigede Liu, Kaushik Bhattacharya, Andrew Stuart, and Anima Anandkumar. “Fourier Neural Operator for Parametric Partial Differential Equations,” 2020. https://openreview.net/forum?id=c8P9NQVtmnO.
参考
trainingOptions | trainnet | dlnetwork