plotconfusion
分類の混同行列のプロット
構文
説明
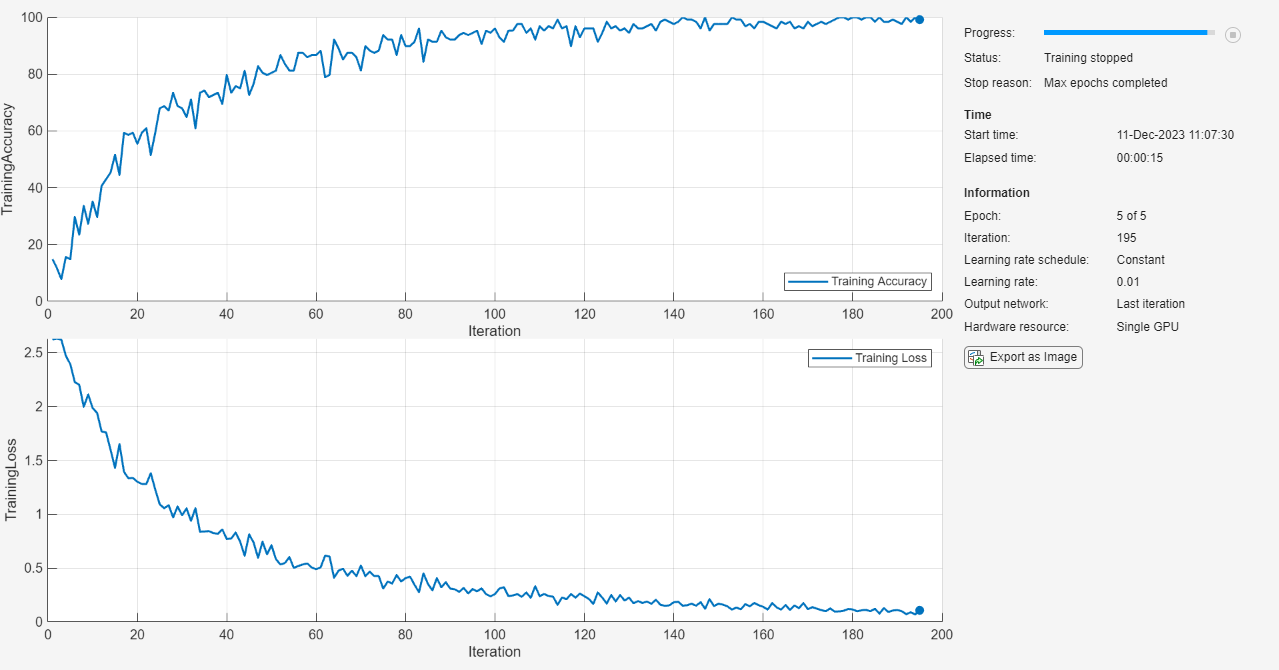
plotconfusion( は、真のラベル targets,outputs)targets および予測ラベル outputs の混同行列をプロットします。ラベルは、categorical ベクトルまたは one-of-N (one-hot) 表現で指定します。
ヒント
plotconfusion は、カテゴリカル ラベルでは推奨されません。代わりに confusionchart を使用してください。
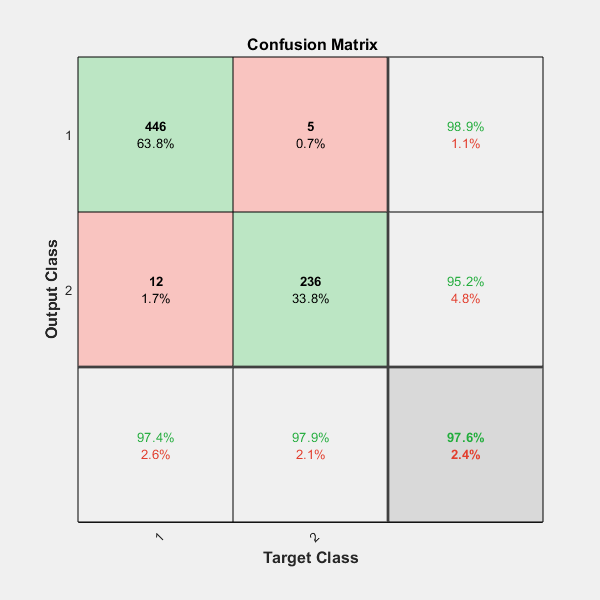
混同行列プロットでは、行は予測されたクラス (出力クラス) に対応し、列は真のクラス (ターゲット クラス) に対応します。対角線上のセルは、正しく分類された観測値に対応します。対角線外のセルは、誤分類された観測値に対応します。観測値の数と観測値の総数に対する割合の両方が各セルに表示されます。
プロットの右端の列は、各クラスに属していると予測されたすべての例について正しく分類された例と誤分類された例の割合を示します。これらのメトリクスは通常、それぞれ精度 (または陽性の予測値) および偽発見率と呼ばれます。プロットの下部の行は、各クラスに属しているすべての例について正しく分類された例と誤分類された例の割合を示します。これらのメトリクスは通常、それぞれ再現率 (または真陽性率) および偽陰性率と呼ばれます。プロットの右下のセルは、全体の精度を示します。
plotconfusion(targets1,outputs1,name1,targets2,outputs2,name2,...,targetsn,outputsn,namen) は、1 つの Figure に複数の混同行列をプロットし、対応するプロットのタイトルの先頭に引数 name を追加します。
例
入力引数
バージョン履歴
R2008a で導入