ECG 信号分類のウェーブレット時間散乱
この例では、ウェーブレット時間散乱とサポート ベクター マシン (SVM) 分類器を使用して人間の心電図 (ECG) 信号を分類する方法を示します。ウェーブレット散乱で、データは、時系列の低分散表現を作成するために一連のウェーブレット変換、非線形性、および平均化全体に伝播されます。ウェーブレット時間散乱によって、クラス判別性を犠牲にすることなく入力信号へのシフトに影響しない信号表現が得られます。この例を実行するには、Wavelet Toolbox™ および Statistics and Machine Learning Toolbox™ が必要です。この例で使用されているデータは、PhysioNet から公的に入手可能です。この分類問題への深層学習的アプローチはこの例ウェーブレット解析と深層学習を使用した時系列の分類にあり、機械学習的アプローチはこの例ウェーブレットベースの特徴とサポート ベクター マシンを使用した信号分類にあります。
用語に関する注意: ウェーブレット散乱のコンテキストにおいて "時間枠" という用語は、平滑化処理の出力をダウンサンプリングした後に取得されるサンプルの数を指します。詳細については、Time Windowsを参照してください。
データの説明
この例では、人の 3 つのグループ (クラス) から取得された ECG データを使用します。3 つのグループとは、心不整脈の患者、鬱血性心不全の患者、および正常洞調律の患者です。この例では、次の 3 つの PhysioNet データベースから 162 個の ECG 記録を使用します。MIT-BIH Arrhythmia Database [3][5]、MIT-BIH Normal Sinus Rhythm Database [3]、The BIDMC Congestive Heart Failure Database [2][3]。合計で、不整脈の患者の記録は 96 個、鬱血性心不全の患者の記録は 30 個、正常洞調律の患者の記録は 36 個あります。目的は、不整脈 (ARR)、鬱血性心不全 (CHF)、および正常洞調律 (NSR) 間を区別できるように分類器に学習させることです。
データのダウンロード
1 番目のステップは、GitHub リポジトリからデータをダウンロードすることです。データをダウンロードするには、[Code] をクリックして [Download ZIP] を選択します。書き込み権限のあるフォルダーに、ファイル physionet_ECG_data-main.zip を保存します。この例の手順では、ファイルを一時ディレクトリ (MATLAB® の tempdir) にダウンロードしているものと仮定します。tempdir とは異なるフォルダーにデータをダウンロードすることを選択した場合は、データの解凍および読み込みに関する後続の手順を変更してください。
ファイル physionet_ECG_data-main.zip には次のものが含まれています。
ECGData.zipREADME.md
また、ECGData.zip には次のものが含まれています。
ECGData.matModified_physionet_data.txtLicense.txt
ECGData.mat は、この例で使用されるデータを保持します。.txt ファイルの Modified_physionet_data.txt は PhysioNet のコピー ポリシーで必要になり、データのソース属性、および ECG の各記録に適用される前処理手順の説明を提供します。
ファイルの読み込み
前の節のダウンロード手順に従った場合、次のコマンドを入力して 2 つのアーカイブ ファイルを解凍します。
unzip(fullfile(tempdir,"physionet_ECG_data-main.zip"),tempdir) unzip(fullfile(tempdir,"physionet_ECG_data-main","ECGData.zip"), ... fullfile(tempdir,"ECGData"))
ECGData.zip ファイルを解凍したら、データをワークスペースに読み込みます。
load(fullfile(tempdir,"ECGData","ECGData.mat"))
ECGData は 2 つのフィールド (Data と Labels) を持つ構造体配列です。Data は 162 行 65,536 列の行列で、各行は 128 Hz でサンプリングした ECG 記録です。各 ECG 時系列の持続時間の合計は 512 秒です。Labels は 162 行 1 列の診断ラベルの cell 配列で、それぞれが Data の各行に対応します。3 つの診断カテゴリは、'ARR' (心不整脈)、'CHF' (鬱血性心不全)、および 'NSR' (正常洞調律) です。
学習データとテスト データの作成
データを 2 つのセット (学習データセットとテスト データセット) に無作為に分割します。補助関数 helperRandomSplit は、無作為の分割を実行します。helperRandomSplit は、学習データと ECGData に対して希望する分割割合を受け入れます。関数 helperRandomSplit は、2 つのデータセットと、それぞれのラベル セットを出力します。trainData と testData の各行は ECG 信号です。trainLabels と testLabels の各要素には、データ行列の対応する行のクラス ラベルが含まれています。この例では、各クラスのデータの 70% を無作為に学習セットに割り当てます。残りの 30% はテスト (予測) 用に取り分けられ、テスト セットに割り当てられます。
percent_train = 70;
[trainData,testData,trainLabels,testLabels] = ...
helperRandomSplit(percent_train,ECGData);trainData セットには 113 個のレコード、testData には 49 個のレコードがあります。計画的に、学習データにはデータの 69.75% (113/162) が含まれています。ARR クラスはデータの 59.26% (96/162)、CHF クラスは 18.52% (30/162)、そして NSR クラスは 22.22% (36/162) を示すことを思い出してください。学習セットとテスト セットに含まれる各クラスの割合を調べます。各クラスの割合はデータセットに含まれるクラスの全体的な割合と一致します。
Ctrain = countcats(categorical(trainLabels))./numel(trainLabels).*100
Ctrain = 3×1
59.2920
18.5841
22.1239
Ctest = countcats(categorical(testLabels))./numel(testLabels).*100
Ctest = 3×1
59.1837
18.3673
22.4490
サンプルのプロット
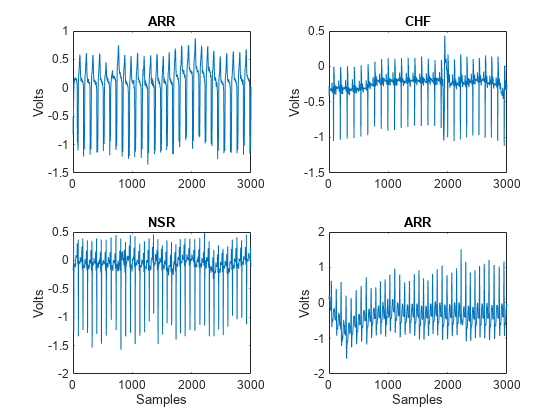
ECGData から無作為に選択した 4 個のレコードのうちの最初の数千個のサンプルをプロットします。補助関数 helperPlotRandomRecords がこれを実行します。helperPlotRandomRecords は ECGData とランダムシードを入力として受け入れます。各クラスの少なくとも 1 つのレコードがプロットされるように、初期シードは 14 で設定されます。各クラスに関連付けられたさまざまな ECG 波形を把握するために、必要に応じて何度でも、ECGData を唯一の入力引数として指定して helperPlotRandomRecords を実行することができます。この補助関数およびすべての補助関数のソース コードは、この例の最後にある「サポート関数」の節で見つけることができます。
helperPlotRandomRecords(ECGData,14)
ウェーブレット時間散乱
ウェーブレット時間散乱ネットワークで指定するキー パラメーターは、時不変のスケール、ウェーブレット変換の数、および各ウェーブレット フィルター バンクのオクターブあたりのウェーブレットの数です。多くのアプリケーションの場合、優れたパフォーマンスを達成するには 2 つのフィルター バンクのカスケードで十分です。この例では、ウェーブレット時間散乱ネットワークを次の既定のフィルター バンクで構成します。最初のフィルター バンクにオクターブあたり 8 ウェーブレット、2 つ目のフィルター バンクにオクターブあたり 1 ウェーブレット。不変スケールは 150 秒に設定されています。
N = size(ECGData.Data,2);
sn = waveletScattering(SignalLength=N,InvarianceScale=150, ...
SamplingFrequency=128);2 つのフィルター バンクのウェーブレット フィルターは以下を使用して可視化できます。
[fb,f,filterparams] = filterbank(sn);
figure
tiledlayout(2,1)
nexttile
plot(f,fb{2}.psift)
xlim([0 128])
grid on
title("1st Filter Bank Wavelet Filters")
nexttile
plot(f,fb{3}.psift)
xlim([0 128])
grid on
title("2nd Filter Bank Wavelet Filters")
xlabel("Hz")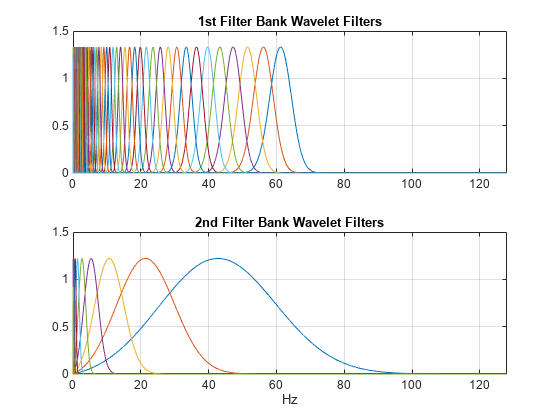
不変スケールを示すには、スケーリング関数の逆フーリエ変換を取得し、時間で 0 秒にセンタリングします。2 つの黒の垂直線は、-75 と 75 秒の境界をマークします。また、最初のフィルター バンクからの最も粗いスケール (最も低い周波数) のウェーブレットについて、実数部と虚数部をプロットします。最も粗いスケール ウェーブレットは、スケーリング関数の時間サポートによって決定される不変スケールを超えません。これは、ウェーブレット時間散乱の重要なプロパティです。
figure
phi = ifftshift(ifft(fb{1}.phift));
psiL1 = ifftshift(ifft(fb{2}.psift(:,end)));
t = (-2^15:2^15-1).*1/128;
scalplt = plot(t,phi);
hold on
grid on
ylim([-1.5e-4 1.6e-4])
plot([-75 -75],[-1.5e-4 1.6e-4],"k--")
plot([75 75],[-1.5e-4 1.6e-4],"k--")
xlabel("Seconds")
ylabel("Amplitude")
wavplt = plot(t,[real(psiL1) imag(psiL1)]);
legend([scalplt wavplt(1) wavplt(2)], ...
{"Scaling Function","Wavelet-Real Part","Wavelet-Imaginary Part"})
title({"Scaling Function";"Coarsest-Scale Wavelet First Filter Bank"})
hold off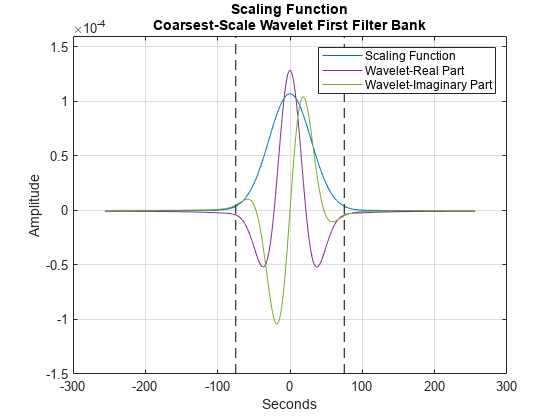
散乱ネットワークを構成した後、学習データの散乱係数を行列として取得します。featureMatrix を複数の信号で実行すると、各列は単一信号として取り扱われます。
scat_features_train = featureMatrix(sn,trainData');
この場合、featureMatrix の出力は、409×16×113 です。各ページのテンソル scat_features_train は、1 つの信号の散乱変換です。ウェーブレット散乱変換は、スケーリング関数の帯域幅に基づいて時間で大きくダウンサンプリングされます。この場合、409 個の散乱パスのそれぞれに対して 16 個の時間枠になります。
SVM 分類器と互換性のある行列を取得するために、各列が散乱パスに対応し、各行が散乱時間枠である行列に、多重信号の散乱変換の形状を変更します。この場合、学習データで 113 個の各信号に対して 16 個の時間枠があるため、1808 行を取得します。
Nwin = size(scat_features_train,2);
scat_features_train = permute(scat_features_train,[2 3 1]);
scat_features_train = reshape(scat_features_train, ...
size(scat_features_train,1)*size(scat_features_train,2),[]);テスト データに対して手順を繰り返します。最初に、学習セットに 49 ECG 波形があるため、scat_features_test は 409×16×49 です。SVM 分類器の形状を変更すると、特徴量行列は 784 行 416 列です。
scat_features_test = featureMatrix(sn,testData');
scat_features_test = permute(scat_features_test,[2 3 1]);
scat_features_test = reshape(scat_features_test, ...
size(scat_features_test,1)*size(scat_features_test,2),[]);各信号に対して 16 個の散乱時間枠を取得したため、枠数に一致させるためラベルを作成する必要があります。補助関数 createSequenceLabels は時間枠数を基にこれを実行します。
[sequence_labels_train,sequence_labels_test] = createSequenceLabels(Nwin,trainLabels,testLabels);
交差検証
分類の場合、2 つの解析が実行されます。最初に、散乱データすべてを使用して、マルチクラス SVM を 2 次カーネルに近似します。合計で、データセット全体で 2592 個の散乱シーケンス (162 信号のそれぞれに 16) があります。誤り率、または損失率は、5 分割交差検証を使用して推定されます。
scat_features = [scat_features_train; scat_features_test]; allLabels_scat = [sequence_labels_train; sequence_labels_test]; rng(1); template = templateSVM(... KernelFunction="polynomial", ... PolynomialOrder=2, ... KernelScale="auto", ... BoxConstraint=1, ... Standardize=true); classificationSVM = fitcecoc(... scat_features, ... allLabels_scat, ... Learners=template, ... Coding="onevsone", ... ClassNames={'ARR';'CHF';'NSR'}); kfoldmodel = crossval(classificationSVM,KFold=5);
損失と混同行列を計算します。精度を表示します。
predLabels = kfoldPredict(kfoldmodel); loss = kfoldLoss(kfoldmodel)*100; confmatCV = confusionmat(allLabels_scat,predLabels)
confmatCV = 3×3
1535 0 1
1 479 0
0 0 576
fprintf("Accuracy is %2.2f percent.\n",100-loss);Accuracy is 99.92 percent.
精度は 99.88% です。これは、非常に良好ですが、実際の結果は各時間枠が個別に分類されるものよりも良いと考えられます。各信号に対して 16 個の個別の分類があります。簡単な多数決を使用して、各散乱表現に対して単一のクラス予測を取得します。
classes = categorical({'ARR','CHF','NSR'});
[ClassVotes,ClassCounts] = helperMajorityVote(predLabels,[trainLabels; testLabels],classes);散乱時間枠の各設定に対してクラス予測のモードに基づいて実際の交差検証の精度を決定します。モードが指定された設定に対して一意でない場合、補助関数 helperMajorityVote は、'NoUniqueMode' によって指定された分類誤差を返します。これは、混同行列の余分の列になります。この場合、散乱予測の各設定に一意のモードが存在するためにすべてゼロになります。
CVaccuracy = sum(eq(ClassVotes,categorical([trainLabels; testLabels])))/162*100;
fprintf("True cross-validation accuracy is %2.2f percent.\n",CVaccuracy);True cross-validation accuracy is 100.00 percent.
MVconfmatCV = confusionmat(categorical([trainLabels; testLabels]),ClassVotes); MVconfmatCV
MVconfmatCV = 4×4
96 0 0 0
0 30 0 0
0 0 36 0
0 0 0 0
散乱は、交差検証済みモデルですべての信号を正しく分類しています。ClassCounts を調べる場合、confmatCV の 2 つの誤分類された時間枠は、16 個の散乱時間枠のうち 15 個が正しく分類された 2 つの信号に起因することがわかります。
SVM 分類
次の解析では、マルチクラスの 2 次 SVM を学習データのみ (70%) に当てはめてから、そのモデルを使用してテスト用に取り分けられた 30% のデータに対して予測を行います。テスト セットには 49 個のデータ レコードがあります。個々の散乱時間枠で多数決を使用します。
model = fitcecoc(... scat_features_train, ... sequence_labels_train, ... Learners=template, ... Coding="onevsone", ... ClassNames={'ARR','CHF','NSR'}); predLabels = predict(model,scat_features_test); [TestVotes,TestCounts] = helperMajorityVote(predLabels,testLabels,classes); testaccuracy = sum(eq(TestVotes,categorical(testLabels)))/numel(testLabels)*100; fprintf("The test accuracy is %2.2f percent. \n",testaccuracy);
The test accuracy is 97.96 percent.
confusionchart(categorical(testLabels),TestVotes)
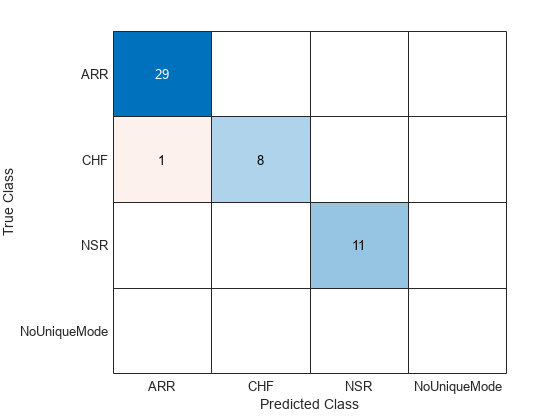
テスト データセットに関する分類精度は約 98% です。混同行列は 1 つの CHF レコードが ARR と誤分類されていることを示しています。48 個のその他の信号はすべて正しく分類されています。
まとめ
この例では、3 つの診断クラスのうち 1 つに ECG 波形を分類するためにウェーブレット時間散乱と SVM 分類器を使用しました。強力な特徴抽出器であることを実証するウェーブレット散乱です。これには、分類に対してロバスト特徴のセットを得られるようにユーザー指定のパラメーターの最小設定のみが必要でした。これを例ウェーブレットベースの特徴とサポート ベクター マシンを使用した信号分類と比較します。この例では分類で使用するための特徴量の作成に多くの専門知識が必要でした。ウェーブレット時間散乱では、時間不変性のスケール、フィルター バンク (またはウェーブレット変換) の数、およびオクターブあたりのウェーブレットの数のみを指定する必要があります。ウェーブレット散乱変換と SVM 分類器の組み合わせによって、交差検証済みモデルで 100% の分類と、ホールドアウトのテスト セットの散乱変換に SVM を適用したときに 98% の正しい分類が得られました。
参考文献
Anden, J., Mallat, S. 2014. Deep scattering spectrum, IEEE Transactions on Signal Processing, 62, 16, pp. 4114-4128.
Baim DS, Colucci WS, Monrad ES, Smith HS, Wright RF, Lanoue A, Gauthier DF, Ransil BJ, Grossman W, Braunwald E. Survival of patients with severe congestive heart failure treated with oral milrinone. J American College of Cardiology 1986 Mar; 7(3):661-670.
Goldberger AL, Amaral LAN, Glass L, Hausdorff JM, Ivanov PCh, Mark RG, Mietus JE, Moody GB, Peng C-K, Stanley HE. PhysioBank, PhysioToolkit,and PhysioNet: Components of a New Research Resource for Complex Physiologic Signals. Circulation. Vol. 101, No. 23, 13 June 2000, pp. e215-e220.
http://circ.ahajournals.org/content/101/23/e215.fullMallat, S., 2012. Group invariant scattering. Communications in Pure and Applied Mathematics, 65, 10, pp. 1331-1398.
Moody GB, Mark RG. The impact of the MIT-BIH Arrhythmia Database. IEEE Eng in Med and Biol 20(3):45-50 (May-June 2001). (PMID: 11446209)
サポート関数
helperPlotRandomRecords - ECGData から無作為に選択した 4 個の ECG 信号をプロットします。
function helperPlotRandomRecords(ECGData,randomSeed) % This function is only intended to support the XpwWaveletMLExample. It may % change or be removed in a future release. if nargin==2 rng(randomSeed) end M = size(ECGData.Data,1); idxsel = randperm(M,4); tiledlayout(2,2) for numplot = 1:4 nexttile plot(ECGData.Data(idxsel(numplot),1:3000)) ylabel('Volts') if numplot > 2 xlabel('Samples') end title(ECGData.Labels{idxsel(numplot)}) end end
helperMajorityVote - 散乱時間枠の各セットに予測したクラス ラベルでモードを探します。関数は、散乱時間枠の各セットにクラス ラベル モードとクラス予測の数の両方を返します。固有のモードがない場合、helperMajorityVote は散乱時間枠のセットが分類誤差であることを示す "エラー" のクラス ラベルを返します。
function [ClassVotes,ClassCounts] = helperMajorityVote(predLabels,origLabels,classes) % This function is in support of ECGWaveletTimeScatteringExample. It may % change or be removed in a future release. % Make categorical arrays if the labels are not already categorical predLabels = categorical(predLabels); origLabels = categorical(origLabels); % Expects both predLabels and origLabels to be categorical vectors Npred = numel(predLabels); Norig = numel(origLabels); Nwin = Npred/Norig; predLabels = reshape(predLabels,Nwin,Norig); assert(size(predLabels,2) == length(origLabels)); ClassCounts = countcats(predLabels); [~,idx] = max(ClassCounts); ClassVotes = classes(idx); % Check for any ties in the maximum values and ensure they are marked as % error if the mode occurs more than once modecnt = modecount(predLabels,string(classes)); ClassVotes(modecnt>1) = categorical({'NoUniqueMode'}); ClassVotes = ClassVotes(:); %------------------------------------------------------------------------- function modecnt = modecount(predlabels,classes) % Ensure there is a unique mode modecnt = zeros(size(predlabels,2),1); for nc = 1:size(predlabels,2) hc = histcounts(predlabels(:,nc),classes); hc = hc-max(hc); if sum(hc == 0) > 1 modecnt(nc) = 1; end end end end