loss
学習した特徴量の重みの精度をテスト データに対して評価
構文
説明
err = loss(mdl,Tbl,ResponseVarName)Tbl 内の予測子と table Tbl 内の変数 ResponseVarName に対するモデル mdl の誤分類誤差を計算します。この構文は、mdl が元々は table で学習させたものである場合に使用します。
例
レーダー信号のデータを含む ionosphere データ セットを読み込みます。X に予測子データが格納されており、応答変数の Y にレーダー信号が良好 ("g") か不良 ("b") かを表す値が格納されます。
load ionosphere近傍成分分析 (NCA) を実行して、レーダー反射を正しく分類するために重要な予測子を特定します。
mdl = fscnca(X,Y)
mdl =
FeatureSelectionNCAClassification
NumObservations: 351
ModelParameters: [1×1 struct]
Lambda: 0.0028
FitMethod: 'exact'
Solver: 'lbfgs'
GradientTolerance: 1.0000e-06
IterationLimit: 1000
PassLimit: 5
InitialLearningRate: []
Verbose: 0
InitialFeatureWeights: [34×1 double]
FeatureWeights: [34×1 double]
FitInfo: [1×1 struct]
Mu: []
Sigma: []
X: [351×34 double]
Y: {351×1 cell}
W: [351×1 double]
ClassNames: {2×1 cell}
Properties, Methods
mdl は、NCA モデルのデータ、当てはめの情報、特徴量の重み、その他のパラメーターが格納された FeatureSelectionNCAClassification モデル オブジェクトです。特徴量の重みをプロットします。重要でない特徴量の重みはゼロに近くなります。
figure() plot(mdl.FeatureWeights,"o") grid on xlabel("Feature Index") ylabel("Feature Weight")
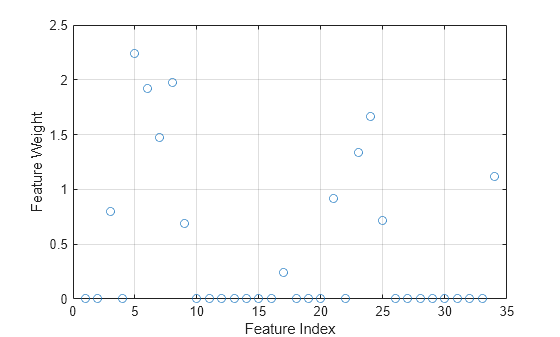
NCA モデルの誤分類誤差を計算します。
L = loss(mdl,X,Y)
L = 0.0142
標本データを読み込みます。
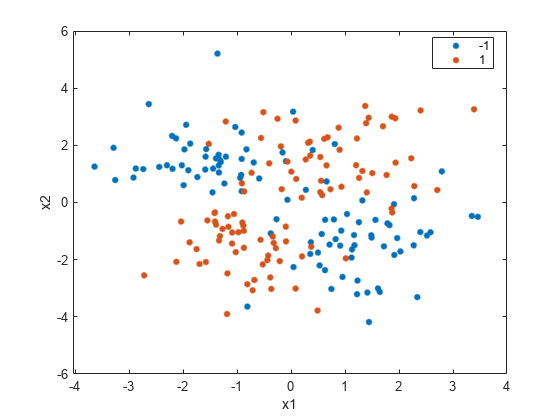
load("twodimclassdata.mat")このデータ セットは、[1] で説明されている方法を使用してシミュレートしたものです。これは 2 次元の 2 クラス分類問題です。1 番目のクラス (クラス -1) のデータは、2 つの二変量正規分布 または から同じ確率で抽出されたものです。ここで、、 および です。同様に、2 番目のクラス (クラス 1) のデータは、2 つの二変量正規分布 または から同じ確率で抽出されたものです。ここで、、 および です。このデータ セットの作成に使用した正規分布のパラメーターにより、[1] で使用されているデータよりデータのクラスターが緊密になります。
クラス別にグループ化したデータの散布図を作成します。
gscatter(X(:,1),X(:,2),y) xlabel("x1") ylabel("x2")
100 個の無関係な特徴量を に追加します。はじめに、平均が 0、分散が 20 の正規分布からデータを生成します。
n = size(X,1);
rng("default")
XwithBadFeatures = [X,randn(n,100)*sqrt(20)];すべての点が 0 と 1 の間になるようにデータを正規化します。
XwithBadFeatures = (XwithBadFeatures-min(XwithBadFeatures,[],1))./ ...
range(XwithBadFeatures,1);
X = XwithBadFeatures;既定値の Lambda (正則化パラメーター ) を使用して近傍成分分析 (NCA) モデルをデータに当てはめます。LBFGS ソルバーを使用し、収束情報を表示します。
ncaMdl = fscnca(X,y,FitMethod="exact",Verbose=1, ... Solver="lbfgs");
o Solver = LBFGS, HessianHistorySize = 15, LineSearchMethod = weakwolfe
|====================================================================================================|
| ITER | FUN VALUE | NORM GRAD | NORM STEP | CURV | GAMMA | ALPHA | ACCEPT |
|====================================================================================================|
| 0 | 9.519258e-03 | 1.494e-02 | 0.000e+00 | | 4.015e+01 | 0.000e+00 | YES |
| 1 | -3.093574e-01 | 7.186e-03 | 4.018e+00 | OK | 8.956e+01 | 1.000e+00 | YES |
| 2 | -4.809455e-01 | 4.444e-03 | 7.123e+00 | OK | 9.943e+01 | 1.000e+00 | YES |
| 3 | -4.938877e-01 | 3.544e-03 | 1.464e+00 | OK | 9.366e+01 | 1.000e+00 | YES |
| 4 | -4.964759e-01 | 2.901e-03 | 6.084e-01 | OK | 1.554e+02 | 1.000e+00 | YES |
| 5 | -4.972077e-01 | 1.323e-03 | 6.129e-01 | OK | 1.195e+02 | 5.000e-01 | YES |
| 6 | -4.974743e-01 | 1.569e-04 | 2.155e-01 | OK | 1.003e+02 | 1.000e+00 | YES |
| 7 | -4.974868e-01 | 3.844e-05 | 4.161e-02 | OK | 9.835e+01 | 1.000e+00 | YES |
| 8 | -4.974874e-01 | 1.417e-05 | 1.073e-02 | OK | 1.043e+02 | 1.000e+00 | YES |
| 9 | -4.974874e-01 | 4.893e-06 | 1.781e-03 | OK | 1.530e+02 | 1.000e+00 | YES |
| 10 | -4.974874e-01 | 9.404e-08 | 8.947e-04 | OK | 1.670e+02 | 1.000e+00 | YES |
Infinity norm of the final gradient = 9.404e-08
Two norm of the final step = 8.947e-04, TolX = 1.000e-06
Relative infinity norm of the final gradient = 9.404e-08, TolFun = 1.000e-06
EXIT: Local minimum found.
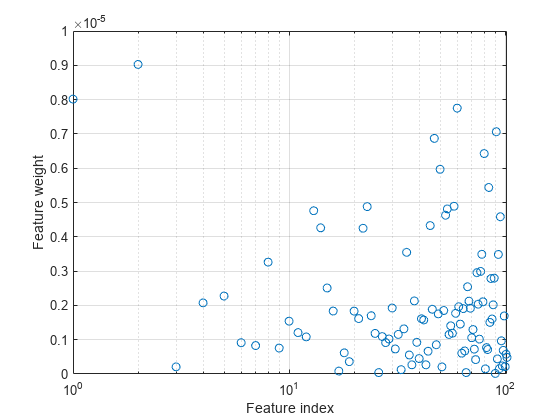
特徴量の重みをプロットします。無関係な特徴量の重みはゼロに非常に近いはずです。
semilogx(ncaMdl.FeatureWeights,"o") xlabel("Feature index") ylabel("Feature weight") grid on
NCA モデルを使用してクラスを予測し、混同行列を計算します。
ypred = predict(ncaMdl,X); confusionchart(y,ypred)
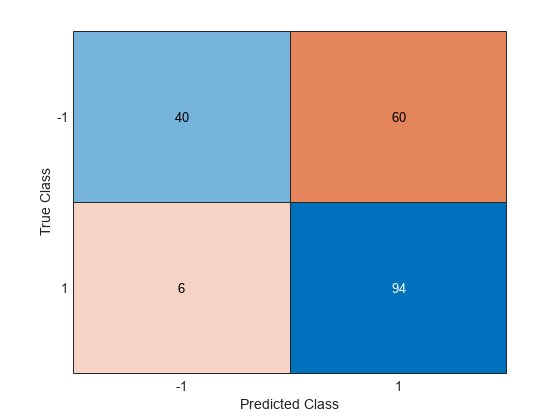
混同行列は、クラス –1 に属しているデータのうち 40 個がクラス –1 に属すという予測を示しています。クラス –1 のデータのうち 60 個は、クラス 1 に属すことが予測されています。同様に、クラス 1 のデータのうち 94 個はクラス 1 に、6 個はクラス –1 に属すことが予測されています。クラス -1 の予測精度が良くありません。
すべての重みがゼロに非常に近くなっています。これは、モデルに学習させるときに使用した の値が大きすぎることを示します。 では、すべての特徴量の重みがゼロに近づきます。したがって、関連がある特徴量を判別するには、ほとんどのケースで正則化パラメーターを調整することが重要です。
5 分割交差検証を使用して、fscnca を使用する特徴選択用に を調整します。 の調整とは、分類損失が最小になる の値を求めることを意味します。交差検証を使用して を調整するため、以下を行います。
1.データを 5 つの分割に分割します。各分割について、cvpartition はデータの 4/5 を学習セットとして、1/5 をテスト セットとして割り当てます。さらに各分割について、クラスの比率がほぼ等しい層化区分を cvpartition で作成します。
cvp = cvpartition(y,"KFold",5);
numtestsets = cvp.NumTestSets;
lambdavalues = linspace(0,2,20)/length(y);
lossvalues = zeros(length(lambdavalues),numtestsets);2.各分割の学習セットを使用して、 の各値について近傍成分分析 (NCA) モデルに学習させます。
3.NCA モデルを使用して、分割内の対応するテスト セットの分類損失を計算します。損失の値を記録します。
4.このプロセスをすべての分割およびすべての の値に対して繰り返します。
for i = 1:length(lambdavalues) for k = 1:numtestsets % Extract the training set from the partition object Xtrain = X(cvp.training(k),:); ytrain = y(cvp.training(k),:); % Extract the test set from the partition object Xtest = X(cvp.test(k),:); ytest = y(cvp.test(k),:); % Train an NCA model for classification using the training set ncaMdl = fscnca(Xtrain,ytrain,FitMethod="exact", ... Solver="lbfgs",Lambda=lambdavalues(i)); % Compute the classification loss for the test set using the NCA % model lossvalues(i,k) = loss(ncaMdl,Xtest,ytest, ... LossFunction="quadratic"); end end
分割の平均損失値を の値についてプロットします。最小の損失に対応する の値が のテスト済みの値の境界に位置する場合、 の値の範囲を再検討する必要があります。
plot(lambdavalues,mean(lossvalues,2),"o-") xlabel("Lambda values") ylabel("Loss values") grid on
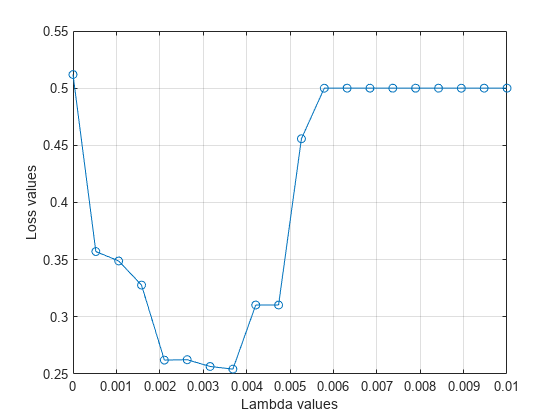
最小の平均損失に対応する の値を求めます。
[~,idx] = min(mean(lossvalues,2)); % Find the index bestlambda = lambdavalues(idx) % Find the best lambda value
bestlambda = 0.0037
最適な の値を使用して、すべてのデータに NCA モデルを当てはめます。LBFGS ソルバーを使用し、収束情報を表示します。
ncaMdl = fscnca(X,y,FitMethod="exact",Verbose=1, ... Solver="lbfgs",Lambda=bestlambda);
o Solver = LBFGS, HessianHistorySize = 15, LineSearchMethod = weakwolfe
|====================================================================================================|
| ITER | FUN VALUE | NORM GRAD | NORM STEP | CURV | GAMMA | ALPHA | ACCEPT |
|====================================================================================================|
| 0 | -1.246913e-01 | 1.231e-02 | 0.000e+00 | | 4.873e+01 | 0.000e+00 | YES |
| 1 | -3.411330e-01 | 5.717e-03 | 3.618e+00 | OK | 1.068e+02 | 1.000e+00 | YES |
| 2 | -5.226111e-01 | 3.763e-02 | 8.252e+00 | OK | 7.825e+01 | 1.000e+00 | YES |
| 3 | -5.817731e-01 | 8.496e-03 | 2.340e+00 | OK | 5.591e+01 | 5.000e-01 | YES |
| 4 | -6.132632e-01 | 6.863e-03 | 2.526e+00 | OK | 8.228e+01 | 1.000e+00 | YES |
| 5 | -6.135264e-01 | 9.373e-03 | 7.341e-01 | OK | 3.244e+01 | 1.000e+00 | YES |
| 6 | -6.147894e-01 | 1.182e-03 | 2.933e-01 | OK | 2.447e+01 | 1.000e+00 | YES |
| 7 | -6.148714e-01 | 6.392e-04 | 6.688e-02 | OK | 3.195e+01 | 1.000e+00 | YES |
| 8 | -6.149524e-01 | 6.521e-04 | 9.934e-02 | OK | 1.236e+02 | 1.000e+00 | YES |
| 9 | -6.149972e-01 | 1.154e-04 | 1.191e-01 | OK | 1.171e+02 | 1.000e+00 | YES |
| 10 | -6.149990e-01 | 2.922e-05 | 1.983e-02 | OK | 7.365e+01 | 1.000e+00 | YES |
| 11 | -6.149993e-01 | 1.556e-05 | 8.354e-03 | OK | 1.288e+02 | 1.000e+00 | YES |
| 12 | -6.149994e-01 | 1.147e-05 | 7.256e-03 | OK | 2.332e+02 | 1.000e+00 | YES |
| 13 | -6.149995e-01 | 1.040e-05 | 6.781e-03 | OK | 2.287e+02 | 1.000e+00 | YES |
| 14 | -6.149996e-01 | 9.015e-06 | 6.265e-03 | OK | 9.974e+01 | 1.000e+00 | YES |
| 15 | -6.149996e-01 | 7.763e-06 | 5.206e-03 | OK | 2.919e+02 | 1.000e+00 | YES |
| 16 | -6.149997e-01 | 8.374e-06 | 1.679e-02 | OK | 6.878e+02 | 1.000e+00 | YES |
| 17 | -6.149997e-01 | 9.387e-06 | 9.542e-03 | OK | 1.284e+02 | 5.000e-01 | YES |
| 18 | -6.149997e-01 | 3.250e-06 | 5.114e-03 | OK | 1.225e+02 | 1.000e+00 | YES |
| 19 | -6.149997e-01 | 1.574e-06 | 1.275e-03 | OK | 1.808e+02 | 1.000e+00 | YES |
|====================================================================================================|
| ITER | FUN VALUE | NORM GRAD | NORM STEP | CURV | GAMMA | ALPHA | ACCEPT |
|====================================================================================================|
| 20 | -6.149997e-01 | 5.764e-07 | 6.765e-04 | OK | 2.905e+02 | 1.000e+00 | YES |
Infinity norm of the final gradient = 5.764e-07
Two norm of the final step = 6.765e-04, TolX = 1.000e-06
Relative infinity norm of the final gradient = 5.764e-07, TolFun = 1.000e-06
EXIT: Local minimum found.
特徴量の重みをプロットします。
semilogx(ncaMdl.FeatureWeights,"o") xlabel("Feature index") ylabel("Feature weight") grid on
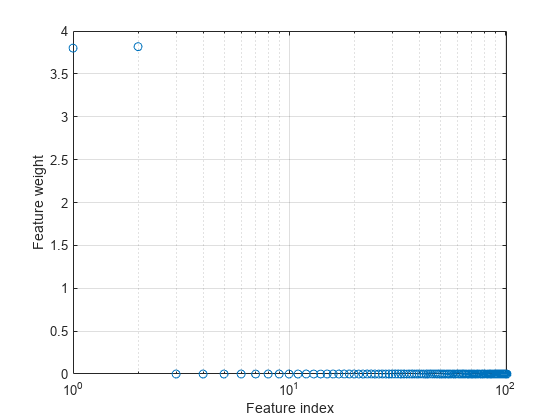
fscnca は、初めの 2 つの特徴量に関連があり残りはそうではないことを正しく判別します。初めの 2 つの特徴量は単独では情報を与えませんが、一緒にすると正確な分類モデルが得られます。
新しいモデルを使用してクラスを予測し、精度を計算します。
ypred = predict(ncaMdl,X); confusionchart(y,ypred)
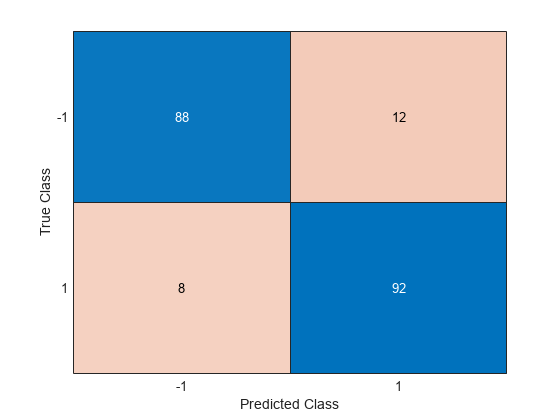
混同行列は、クラス -1 の予測精度が改善されたことを示しています。クラス –1 のデータのうち 88 個はクラス –1 に、12 個はクラス 1 に属すことが予測されています。また、クラス 1 のデータのうち 92 個はクラス 1 に、8 個はクラス –1 に属すことが予測されています。
参考文献
[1] Yang, W., K. Wang, W. Zuo."Neighborhood Component Feature Selection for High-Dimensional Data." Journal of Computers. Vol. 7, Number 1, January, 2012.
入力引数
分類用の近傍成分分析モデル。FeatureSelectionNCAClassification オブジェクトとして指定します。
標本データ。table として指定します。Tbl の各行は 1 つの観測値に、各列は 1 つの予測子変数に対応します。
データ型: table
応答変数の名前。Tbl 内の変数の名前で指定します。table 内の残りの変数は予測子です。
データ型: char | string
予測子変数の値。n 行 p 列の行列を指定します。n は観測値の個数、p は予測子変数の個数です。
データ型: single | double
クラス ラベル。categorical 配列、logical ベクトル、数値ベクトル、string 配列、長さ n の文字ベクトルの cell 配列、または n 行の文字行列として指定します。n は観測値の数です。Y の要素 i または行 i は、X の行 i (観測値 i) に対応するクラス ラベルです。
データ型: single | double | logical | char | string | cell | categorical
損失関数のタイプ。次の値のいずれかとして指定します。
"classiferror"— 次のように定義される 10 進数の誤分類率。は予測されたクラス、 は観測値 i の真のクラスです。 は、 と が同じではないことを示すインジケーターです。
"quadratic"— 次のように定義される二次損失関数。c はクラスの数、 は i 番目の観測値がクラス k に属する推定確率、 は i 番目の観測値がクラス k に属することを示すインジケーターです。
例: "quadratic"
出力引数
学習した特徴量の重みに対する、小さいほど優秀とする精度の尺度。スカラー値として返されます。精度の尺度は、名前と値の引数 LossFunction を使用して指定することができます。
バージョン履歴
R2016b で導入
MATLAB Command
You clicked a link that corresponds to this MATLAB command:
Run the command by entering it in the MATLAB Command Window. Web browsers do not support MATLAB commands.
Web サイトの選択
Web サイトを選択すると、翻訳されたコンテンツにアクセスし、地域のイベントやサービスを確認できます。現在の位置情報に基づき、次のサイトの選択を推奨します:
また、以下のリストから Web サイトを選択することもできます。
最適なサイトパフォーマンスの取得方法
中国のサイト (中国語または英語) を選択することで、最適なサイトパフォーマンスが得られます。その他の国の MathWorks のサイトは、お客様の地域からのアクセスが最適化されていません。
南北アメリカ
- América Latina (Español)
- Canada (English)
- United States (English)
ヨーロッパ
- Belgium (English)
- Denmark (English)
- Deutschland (Deutsch)
- España (Español)
- Finland (English)
- France (Français)
- Ireland (English)
- Italia (Italiano)
- Luxembourg (English)
- Netherlands (English)
- Norway (English)
- Österreich (Deutsch)
- Portugal (English)
- Sweden (English)
- Switzerland
- United Kingdom (English)