異常検出用のコード生成
この例では、学習済みの孤立森モデルまたは 1 クラス サポート ベクター マシン (OCSVM) を使用してデータ内の異常を検出する単精度のコードを生成する方法を示します。
isolationForest の関数isanomalyと ClassificationSVM の関数predictはコード生成をサポートしています。これらのオブジェクト関数には学習済みのモデル オブジェクトが必要ですが、codegen (MATLAB Coder)の -args オプションはそれらのオブジェクトを受け入れません。この例で説明されているように saveLearnerForCoder と loadLearnerForCoder を使用して、この制限に対処します。
次のフロー チャートは、異常検出用のコード生成ワークフローを示しています。
![]()
モデルに学習をさせた後で、saveLearnerForCoder を使用して学習済みモデルを保存します。loadLearnerForCoder を使用して保存済みモデルを読み込んでオブジェクト関数を呼び出す、エントリポイント関数を定義します。次に、codegen を使用してエントリポイント関数のコードを生成し、生成されたコードを確認します。コード生成ワークフローの詳細な例については、機械学習モデルの予測をコマンド ラインで行うコードの生成を参照してください。
データの読み込み
自動車の周囲にある物体の座標が 3 次元の点の集合として格納された LiDAR スキャン データ セットを読み込みます。
load("lidar_subset.mat")
loc = lidar_subset;自動車の周囲の環境を強調するため、道路表面の上部の領域で自動車の左右 20 メートルおよび前後 20 メートルにわたるように関心領域を設定します。
xBound = 20; % in meters yBound = 20; % in meters zLowerBound = 0; % in meters
指定した領域内の点のみが含まれるようにデータをトリミングします。
indices = loc(:,1) <= xBound & loc(:,1) >= -xBound ... & loc(:,2) <= yBound & loc(:,2) >= -yBound ... & loc(:,3) > zLowerBound; loc = loc(indices,:); whos loc
Name Size Bytes Class Attributes loc 19070x3 228840 single
loc は、3 次元の点の 19,070 個の標本を含む単精度の行列です。
2 次元散布図としてデータを可視化します。プロットに注釈を付けて、自動車を強調します。
scatter(loc(:,1),loc(:,2),"."); annotation("ellipse",[0.48 0.48 .1 .1],Color="red")
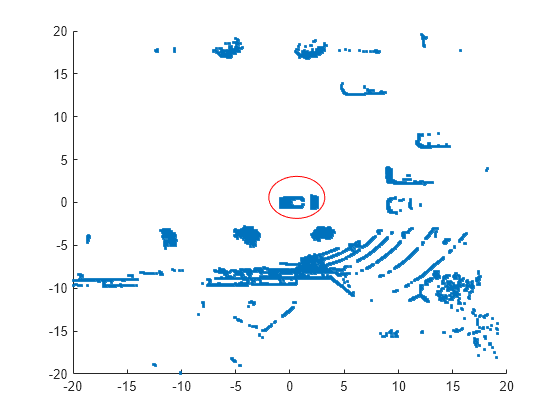
点集合の中心 (赤い丸の部分) には、自動車の屋根とボンネットが含まれています。他のすべての点は障害物です。
データに含まれる外れ値の比率が 0.05 であると仮定します。
contaminationFraction = single(0.05);
孤立森用のコード生成
孤立森モデルの学習
関数iforestを使用して孤立森モデルに学習させます。外れ値の比率 (ContaminationFraction) を 0.05 と指定します。
rng("default") % For reproducibility [forest,tf_forest,s_forest] = iforest(loc,ContaminationFraction=contaminationFraction);
forest はIsolationForestオブジェクトです。iforest は、データ (loc) の異常インジケーター (tf_forest) および異常スコア (s_forest) も返します。iforest は、指定した比率の観測値が外れ値として検出されるようにスコアのしきい値 (forest.ScoreThreshold) を決定します。
saveLearnerForCoder の使用によるモデルの保存
saveLearnerForCoder を使用して、モデル オブジェクトを IsolationForestModel.mat というファイルに保存します。
ForestMdlFileName = "IsolationForestModel";
saveLearnerForCoder(forest,ForestMdlFileName)saveLearnerForCoder は、現在のフォルダーの MATLAB® バイナリ ファイル IsolationForestModel.mat に構造体配列としてオブジェクトを保存します。
エントリポイント関数の定義
入力データの異常インジケーターと異常スコアを返すエントリポイント関数を定義します。この関数では、loadLearnerForCoder を使用して単精度のモデルを読み込み、読み込んだモデルを isanomaly に渡します。
type myIsanomaly.m % Display contents of myIsanomaly.m file
function varargout = myIsanomaly(MdlFileName,x,varargin) %#codegen
%MYISANOMALY Entry-point function for anomaly detection
% This function supports only the example Code Generation for Anomaly
% Detection and might change in a future release.
% This function detects anomalies in new observations x using the saved
% anomaly detection model in the MdlFileName file.
Mdl = loadLearnerForCoder(MdlFileName,DataType="single");
[varargout{1:nargout}] = isanomaly(Mdl,x,varargin{:});
end
コードの生成
4 行 1 列の cell 配列を使用して myIsanomaly の入力引数の型を指定します。エントリポイント関数の各入力引数の型を各 cell に割り当てます。特定のデータ型および配列サイズをもつ一連の値を表す例の値を使用して、データ型と正確な入力配列のサイズを指定します。
ARGS = cell(4,1);
p = numel(forest.PredictorNames);
ARGS{1} = coder.Constant(ForestMdlFileName);
ARGS{2} = coder.typeof(single(0),[Inf,p],[1,0]);
ARGS{3} = coder.Constant("ScoreThreshold");
ARGS{4} = single(0.5);myIsanomaly の 2 番目の入力は可変サイズの入力です。可変サイズの引数の詳細については、コード生成用の可変サイズ引数の指定を参照してください。
エントリポイント関数 myIsanomaly から MEX 関数を生成します。-args オプションおよび cell 配列 ARGS を使用して、入力引数の型を指定します。-nargout オプションを使用して、生成されるエントリポイント関数の出力引数の個数を指定します。
codegen myIsanomaly -args ARGS -nargout 2
Code generation successful.
codegen は、プラットフォームに依存する拡張子をもつ MEX 関数 myIsanomaly_mex を現在のフォルダーに生成します。
生成されたコードの確認
生成された MEX 関数を使用して、学習データ内の異常を検出します。MEX 関数からの異常インジケーターと異常スコアを iforest から返される内容と比較します。
[tf_forest_MEX,s_forest_MEX] = myIsanomaly_mex(ForestMdlFileName,loc,"ScoreThreshold",single(forest.ScoreThreshold));
isequal(tf_forest,tf_forest_MEX)ans = logical
1
max(abs(s_forest-s_forest_MEX))
ans = single
5.9605e-08
isequal は、すべての異常インジケーターが等しいことを意味する logical 1 (true) を返します。異常スコアには有意差はありません。
OCSVM 用のコード生成
OCSVM モデルの学習
関数fitcsvmを使用して 1 クラス学習用のサポート ベクター マシン モデルに学習させます。クラス ラベル変数が 1 のベクトルの場合、関数は 1 クラス学習用にモデルに学習させます。外れ値の比率 (OutlierFraction) を 0.05 と指定します。
MdlOCSVM = fitcsvm(loc,single(ones(size(loc,1),1)),OutlierFraction=contaminationFraction, ...
Standardize=true);MdlOCSVM は、ClassificationSVMオブジェクトです。関数resubPredictを使用して loc の外れ値のスコアを計算します。
[~,s_OCSVM] = resubPredict(MdlOCSVM);
負のスコア値は、対応する観測値が外れ値であることを示します。異常インジケーターを取得します。
tf_OCSVM = s_OCSVM < 0;
saveLearnerForCoder の使用によるモデルの保存
saveLearnerForCoder を使用して、モデル オブジェクトを SVMModel.mat というファイルに保存します。
SVMMdlFileName = "SVMModel";
saveLearnerForCoder(MdlOCSVM,SVMMdlFileName)エントリポイント関数の定義
入力データの異常インジケーターと異常スコアを返すエントリポイント関数を定義します。この関数では、loadLearnerForCoder を使用して単精度のモデルを読み込み、読み込んだモデルを predict に渡して異常スコアを計算します。スコアを使用して異常インジケーターを見つけます。
type myIsanomalySVM.m % Display contents of myIsanomalySVM.m file
function [tf,scores] = myIsanomalySVM(MdlFileName,x,scoreThreshold) %#codegen %MYISANOMALY Entry-point function for anomaly detection % This function supports only the example Code Generation for Anomaly % Detection and might change in a future release. % This function detects anomalies in new observations x using the saved % one-class support vector machine model in the MdlFileName file. Mdl = loadLearnerForCoder(MdlFileName,DataType="single"); [~,scores] = predict(Mdl,x); tf = scores < scoreThreshold; end
コードの生成
3 行 1 列の cell 配列を使用して myIsanomalySVM の入力引数の型を指定します。
ARGS = cell(3,1);
p = numel(MdlOCSVM.PredictorNames);
ARGS{1} = coder.Constant(SVMMdlFileName);
ARGS{2} = coder.typeof(single(0),[Inf,p],[1,0]);
ARGS{3} = single(0);エントリポイント関数 myIsanomalySVM から MEX 関数を生成します。
codegen myIsanomalySVM -args ARGS -nargout 2
Code generation successful.
生成されたコードの確認
生成された MEX 関数を使用して、学習データ内の異常を検出します。MEX 関数からの異常インジケーターと異常スコアを resubPredict から返される内容と比較します。
[tf_OCSVM_MEX,s_OCSVM_MEX] = myIsanomalySVM_mex(SVMMdlFileName,loc,single(0));
isequal(tf_OCSVM,tf_OCSVM_MEX)
ans = logical
1
max(abs(s_OCSVM-s_OCSVM_MEX))
ans = single
0.0133
isequal は、すべての異常インジケーターが等しいことを意味する logical 1 (true) を返します。平均スコア (mean(s_OCSVM)) が約 700 であることを考えれば、異常スコアの差は許容できる大きさです。1 クラス学習用の既定の設定であるガウス カーネルを使用すると、スコアに多少の差が見られます。
検出された外れ値の比較
孤立森モデルおよび 1 クラス SVM モデルで検出された正常な点と外れ値をプロットします。
tiledlayout(2,1) nexttile gscatter(loc(:,1),loc(:,2),tf_forest) legend("Normal Points","Outliers") title("Isolation Forest") nexttile gscatter(loc(:,1),loc(:,2),tf_OCSVM) legend("Normal Points","Outliers") title("One-Class SVM")

2 つの方法で識別された外れ値は互いに似ています。両方の方法の出力に含まれる同じ識別子の比率を計算します。
mean(tf_forest == tf_OCSVM)
ans = 0.9732
参考
codegen (MATLAB Coder) | iforest | isanomaly | fitcsvm | predict