EMG 信号と深層学習を使用した腕の運動の分類
この例では、筋電位 (EMG) 信号に基づいて前腕の運動を分類する方法を説明します。EMG 信号は、筋肉が収縮するときの電気的活動を測定します。
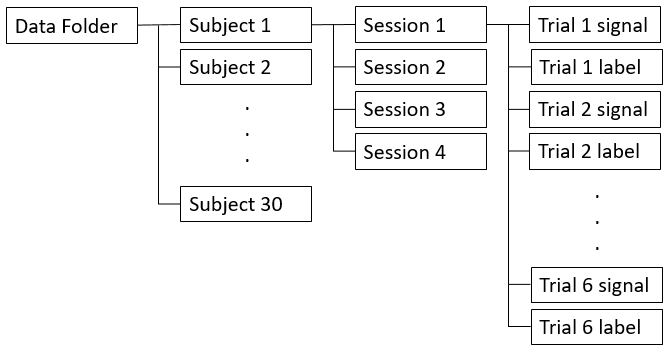
被験者 30 人がそれぞれ 4 回のデータ収集セッションに参加し、8 つの筋肉から EMG 信号を記録しながら、異なる前腕運動を個別に 6 回試行しました。データ セットは 1440 個の MAT ファイルで構成されています。このうち 720 個のファイルには信号データ、残りの 720 個のファイルには対応するラベル データが含まれています。ラベル データは運動変数 motion とインデックス変数 data_indx で構成されます。このデータ セットは、セッションごとのサブフォルダーを格納する被験者フォルダーに分かれています。各セッション サブフォルダーには、各試行に対応する 6 個の信号データ ファイルと 6 個のラベル データ ファイルが格納されています。
変数 motion は、7 つの異なる運動を表す数値配列です。
手を開く
手を閉じる
手首を曲げる
手首を伸ばす
回外
回内
休止
それぞれの運動は 3 秒間保持され、ランダムな順序で 4 回繰り返されました。motion の最初と最後の要素は -1 に等しく、各試行の開始と終了時に取られた長めの休止時間に対応します。変数 data_indx には、各運動の開始インデックスが格納されます。
ファイルは https://ssd.mathworks.com/supportfiles/SPT/data/MyoelectricData.zip からダウンロードできます。
信号およびラベル データを読み取るためのデータストアの作成
ファイルにアクセスするには、ファイルがダウンロードされた場所を指す信号データストアを作成します。信号データを格納するファイルの名前は "d" で終わり、ラベル データを格納するファイルの名前は "i" で終わります。サンプル レートは 3000 Hz です。信号データのみを含むデータストアのサブセットを作成します。
fs = 3000; localfile = matlab.internal.examples.downloadSupportFile("SPT","data/MyoelectricData.zip"); datasetFolder = fullfile(fileparts(localfile),"MyoelectricData"); if ~exist(datasetFolder,"dir") unzip(localfile,datasetFolder) end sds1 = signalDatastore(datasetFolder,IncludeSubFolders=true,SampleRate=fs); p = endsWith(sds1.Files,"d.mat"); sdssig = subset(sds1,p);
同じファイルの場所を指す 2 番目のデータストアを作成し、ラベル ファイルに 2 つの変数の名前を指定します。ラベル データのみを含むこのデータストアのサブセットを作成します。
sds2 = signalDatastore(datasetFolder,SignalVariableNames=["motion";"data_indx"],IncludeSubfolders=true); p = endsWith(sds2.Files,"i.mat"); sdslbl = subset(sds2,p);
最初の EMG 信号の 8 つのチャネルをすべてプロットして、1 回の試行における各筋肉の活性化を可視化します。
signal = preview(sdssig); for i = 1:8 ax(i) = subplot(4,2,i); plot(signal(:,i)) title("Channel"+i) end linkaxes(ax,"y")
ROI table の作成
data_indx のインデックスに基づき、各運動の関心領域 (ROI) の範囲を定義します。最初と最後のラベル値 (= -1) を削除し、残りの数値ラベルを categorical 配列に変換します。1 列目に ROI の範囲、2 列目にラベルを格納する table を作成します。
lbls = {};
i = 1;
while hasdata(sdslbl)
label = read(sdslbl);
idx_start = label{2}(2:end-1)';
idx_end = [idx_start(2:end)-1;idx_start(end)+(3*fs)];
val = categorical(label{1}(2:end-1)',[1 2 3 4 5 6 7], ...
["HandOpen" "HandClose" "WristFlexion" "WristExtension" "Supination" "Pronation" "Rest"]);
ROI = [idx_start idx_end];
% In some cases, the number of label values and ROIs are not equal.
% To eliminate these inconsistencies, remove the extra label value or ROI limits.
if numel(val) < size(ROI,1)
ROI(end,:) = [];
elseif numel(val) > size(ROI,1)
val(end) = [];
end
lbltable = table(ROI,val);
lbls{i} = {lbltable};
i = i+1;
endデータストアの準備
変更後のラベル データを含む新しいデータストアを作成し、最初の観測値から得られた ROI table を表示します。
lblDS = signalDatastore(lbls);
lblstable = preview(lblDS);
lblstable{1}信号およびラベル データを 1 つのデータストアに結合します。
DS = combine(sdssig,lblDS); combinedData = preview(DS)
信号マスクを作成し、plotsigroi を呼び出して最初の信号の最初のチャネルのラベル付き運動領域を表示します。黒で示された信号の開始および終了部分は長めの休止時間を表し、この部分は次の前処理手順で削除されます。
figure
msk = signalMask(combinedData{2});
plotsigroi(msk,combinedData{1}(:,1))データの前処理
次の前処理タスクを実行する関数 preprocess を使用して、結合したデータストアを変換します。
各信号の開始および終了部分の長めの休止時間を削除します。
回内および回外運動を削除します。前腕の回内を可能にする主な筋肉からの EMG を記録せず、回外に関与する筋肉の 1 つのみから EMG を記録しているため、これらの運動は関数によりデータから除外されます。
休止時間を削除します。
低域カットオフ周波数 10 Hz と高域カットオフ周波数 400 Hz をもつバンドパス フィルターを使用して信号のフィルター処理を行います。
信号およびラベル データを 1000 Hz までダウンサンプリングします。
関心領域 (運動) とラベルの信号マスクを作成します。ここで、sequence-to-sequence 分類ができるように、各信号サンプルは対応するラベルをもちます。
信号を、長さ 12000 サンプルの短いセグメントに分割します。
tDS = transform(DS,@preprocess); transformedData = preview(tDS)
学習セットとテスト セットへのデータの分割
データの 80% をネットワークの学習用に、20% をネットワークのテスト用に使用します。ランダムなインデックスを 24 (6 回の試行 x 4 回のセッション = 被験者 1 人につき 24 個のファイル) で乗算して、単一の被験者からのデータを学習セットとテスト セットの両方に含めないようにします。
rng default [trainIdx,~,testIdx] = dividerand(30,0.8,0,0.2); trainIdx_all = {}; m = 1; for k = trainIdx if k == 1 start = k; else start = ((k-1)*24)+1; end l = start:k*24; trainIdx_all{m} = l; m = m+1; end trainIdx_all = cell2mat(trainIdx_all)'; trainDS = subset(tDS,trainIdx_all); testIdx_all = {}; m = 1; for k = testIdx if k == 1 start = k; else start = ((k-1)*24)+1; end l = start:k*24; testIdx_all{m} = l; m = m+1; end testIdx_all = cell2mat(testIdx_all)'; testDS = subset(tDS,testIdx_all);
ネットワークの学習
学習エポックごとに前処理手順を繰り返さないようにすることで学習時間を短縮するには、ネットワークの学習を行う前にすべてのデータをメモリに読み取ります。データを並列で読み取ることでこのプロセスを高速化できます (これには Parallel Computing Toolbox™ が必要です)。
traindata = readall(trainDS,"UseParallel",true);畳み込みニューラル ネットワークを定義します。出力サイズが 4 の fullyConnectedLayer を運動タイプごとに 1 つ指定します。
layers = [ ... sequenceInputLayer(8) convolution1dLayer(8,32,Stride=2,Padding="same") reluLayer layerNormalizationLayer convolution1dLayer(8,16,Stride=2,Padding="same") reluLayer layerNormalizationLayer transposedConv1dLayer(8,16,Stride=2,Cropping="same") reluLayer layerNormalizationLayer transposedConv1dLayer(8,32,Stride=2,Cropping="same") reluLayer layerNormalizationLayer fullyConnectedLayer(4) softmaxLayer ];
ネットワーク学習のオプションを指定します。Adam オプティマイザーと 32 のミニバッチ サイズを使用します。初期学習率を 0.001 に設定し、エポックの最大数を 100 に設定します。すべてのエポックでデータをシャッフルします。
options = trainingOptions("adam", ... MaxEpochs=100, ... MiniBatchSize=32, ... Plots="training-progress",... InitialLearnRate=0.001,... Verbose=0,... Shuffle="every-epoch",... Metric="accuracy");
rawNet = trainnet(traindata(:,1),traindata(:,2),layers,"crossentropy",options);
テスト信号の分類
学習済みネットワークを使用してテスト データ セットの運動を分類します。混同チャートを使用して結果を表示します。
testdata = readall(testDS);
scores = minibatchpredict(rawNet,testdata(:,1),MiniBatchSize=128);
classNames = categories(traindata{1,2});
predTest = scores2label(scores,classNames);confusionchart(vertcat(testdata{:,2}),[predTest(:)],Normalization="column-normalized")
まとめ
この例では、sequence-to-sequence 分類を実行して、EMG 信号に基づいて異なる腕の運動を検出する方法を示しました。80 個の隠れユニットをもつ畳み込みネットワークを使用した場合の全体的な精度は約 84% でした。手を開く運動と手首を伸ばす運動との間、および手を閉じる運動と手首を曲げる運動との間にいくつかの誤分類が発生しました。手を開く運動では、手首を伸ばす運動で使う筋肉の 1 つと同じ筋肉を使います。同様に、手を閉じる運動と手首を曲げる運動では、同じ筋肉が活性化されることがあります。さらに、腕への EMG 電極の配置では、分類精度が最も高い手首を曲げる運動に使われる筋肉が主な対象となりました。
この例のデータはカールトン大学の Chan 教授が収集したもので、https://www.sce.carleton.ca/faculty/chan/index.php?page=matlab[1] で確認できます。
参考文献
[1] Chan, Adrian D.C., and Geoffrey C. Green. 2007. "Myoelectric Control Development Toolbox." Paper presented at 30th Conference of the Canadian Medical & Biological Engineering Society, Toronto, Canada, 2007.
関数 preprocess
function Tsds = preprocess(inputDS) sig = inputDS{1}; roiTable = inputDS{2}; % Remove first and last rest periods from signal sig(roiTable.ROI(end,2):end,:) = []; sig(1:roiTable.ROI(1,1),:) = []; % Shift ROI limits to account for deleting start and end of signal roiTable.ROI = roiTable.ROI-(roiTable.ROI(1,1)-1); % Create signal mask m = signalMask(roiTable); L = length(sig); % Obtain sequence of category labels and remove pronation, supination, and rest motions mask = catmask(m,L); idx = ~ismember(mask,{'Pronation','Supination','Rest'}); mask = mask(idx); sig = sig(idx,:); % Create new signal mask without pronation and supination categories m2 = signalMask(mask); m2.SpecifySelectedCategories = true; % m2.SelectedCategories = [1 2 3 4 7]; m2.SelectedCategories = [1 2 3 4]; mask = catmask(m2); % Filter and downsample signal data sigfilt = bandpass(sig,[10 400],3000); downsig = downsample(sigfilt,3); % Downsample label data downmask = downsample(mask,3); targetLength = 12000; % Get number of chunks numChunks = floor(size(downsig,1)/targetLength); % Truncate signal and mask to integer number of chunks sig = downsig(1:numChunks*targetLength,:); mask = downmask(1:numChunks*targetLength); % Create a cell array containing signal chunks sigOut = {}; step = 0; for i = 1:numChunks sigOut{i,1} = sig(1+step:i*targetLength,:); step = step+targetLength; end % Create a cell array containing mask chunks lblOut = reshape(mask,targetLength,numChunks); lblOut = num2cell(lblOut,1)'; % Output a two-column cell array with all chunks Tsds = [sigOut,lblOut]; end