勾配分布のプロットによる深層ニューラル ネットワークの勾配消失の検出
この例では、深層ニューラル ネットワークの学習中に勾配消失を監視する方法を説明します。
深いネットワークの学習では、"勾配消失" の問題がよく発生します。深層学習の学習アルゴリズムは、学習中にネットワークの学習可能なパラメーターを調整して損失を最小にすることを目的としています。勾配ベースの学習アルゴリズムは、現在の学習可能なパラメーターについての損失関数の勾配を使用して調整レベルを決定します。初期の層では、前の層から伝播された勾配を使用して勾配を計算します。そのため、常に 1 未満の勾配値を生成する活性化関数がネットワークに含まれている場合、更新アルゴリズムが初期の層に移動するにつれて、勾配の値が次第に小さくなります。その結果、ネットワークの初期の層はほとんど 0 に近い小さな勾配を受け取ることになり、ネットワークは学習ができなくなってしまいます。しかし、活性化関数の勾配が常に 1 以上であれば、勾配がネットワーク内をくまなく伝播でき、勾配消失が発生する可能性を減らすことができます。
この例では、異なる活性化関数をもつ 2 つのネットワークの学習を行い、勾配分布を比較します。
活性化関数の比較
活性化関数の特性の違いを説明するため、一般的な 2 つの深層学習活性化関数である ReLU およびシグモイドを比較します。
ReLU 活性化関数とシグモイド活性化関数の勾配を評価します。
x = linspace(-5,5,1000); reluActivation = max(0,x); reluGradient = gradient(reluActivation,0.01); sigmoidActivation = 1./(1 + exp(-x)); sigmoidGradient = gradient(sigmoidActivation,0.01);
ReLU 活性化関数とシグモイド活性化関数、ならびにそれらの勾配をプロットします。
figure tiledlayout(1,2) nexttile plot(x,[reluActivation;reluGradient]) legend("ReLU","Gradient of ReLU") nexttile plot(x,[sigmoidActivation;sigmoidGradient]) legend("Sigmoid","Gradient of Sigmoid")

ReLU の勾配は、全範囲で 0 または 1 です。そのため、ネットワーク内で勾配が逆伝播しても勾配が次第に小さくなることはなく、勾配消失が発生する可能性は減ります。シグモイドの勾配曲線は、全範囲で 1 未満です。そのため、シグモイド活性化層を含むネットワークは、勾配消失問題の影響を受ける可能性があります。
データの読み込み
digitTrain4DArrayData を使用して、5000 個の手書き数字の合成イメージとそのラベルから成るサンプル データを読み込みます。
[XTrain,TTrain] = digitTrain4DArrayData; numObservations = length(TTrain);
学習イメージのサイズを自動的に変更するには、拡張イメージ データストアを使用します。
inputSize = [28,28,1]; augimdsTrain = augmentedImageDatastore(inputSize(1:2),XTrain,TTrain);
学習データ内のクラスの数を決定します。
classes = categories(TTrain); numClasses = numel(classes);
ネットワークの定義
活性化層の効果を比較するため、2 つのネットワークを構築します。各ネットワークには、4 つの全結合層を隔てる ReLU 活性化層またはシグモイド活性化層を含めます。これら 2 つのネットワークの学習の進行状況を比較することで、学習時における活性化層の影響を確認できます。これらのネットワークは、あくまで説明のためのものです。シンプルなイメージ分類ネットワークの作成および学習を行う方法を示す例については、分類用のシンプルな深層学習ニューラル ネットワークの作成を参照してください。
activationTypes = ["ReLU","Sigmoid"]; numNetworks = length(activationTypes); for i = 1:numNetworks activationType = activationTypes(i); switch activationType case "ReLU" activationLayer = reluLayer; case "Sigmoid" activationLayer = sigmoidLayer; end layers = [ imageInputLayer(inputSize,Normalization="none") fullyConnectedLayer(10) activationLayer fullyConnectedLayer(10) activationLayer fullyConnectedLayer(10) activationLayer fullyConnectedLayer(numClasses) softmaxLayer]; % Create a dlnetwork object from the layers. networks{i} = dlnetwork(layers); end
モデル損失関数の定義
この例の最後にリストされている関数 modelLoss を作成します。この関数は dlnetwork オブジェクト、入力データのミニバッチとそれに対応するラベルを入力として受け取り、ネットワークの学習可能なパラメーターについての損失とその損失の勾配を返します。
学習オプションの指定
ミニバッチ サイズを 128 として 50 エポック学習させます。
numEpochs = 50; miniBatchSize = 128;
モデルの学習
2 つのネットワークを比較するため、各ネットワークの各層の損失と平均勾配を追跡します。各ネットワークには 4 つの学習可能なパラメーターの層が含まれています。
numIterations = numEpochs*ceil(numObservations/miniBatchSize); numLearnableLayers = 4; losses = zeros(numIterations,numNetworks); meanGradients = zeros(numIterations,numNetworks,numLearnableLayers);
学習中にイメージのミニバッチを処理および管理する minibatchqueue オブジェクトを作成します。各ミニバッチで次を行います。
この例の最後で定義されているカスタム ミニバッチ前処理関数
preprocessMiniBatchを使用して、ラベルを one-hot 符号化変数に変換します。イメージ データを次元ラベル
"SSCB"(spatial、spatial、channel、batch) で形式を整えます。既定では、minibatchqueueオブジェクトは、基となる型が single であるdlarrayオブジェクトにデータを変換します。形式をクラス ラベルに追加しないでください。GPU が利用できる場合、GPU で学習を行います。既定では、
minibatchqueueオブジェクトは、GPU が利用可能な場合、各出力をgpuArrayに変換します。GPU を使用するには、Parallel Computing Toolbox™ とサポートされている GPU デバイスが必要です。サポートされているデバイスの詳細については、GPU 計算の要件 (Parallel Computing Toolbox)を参照してください。
mbq = minibatchqueue(augimdsTrain,... MiniBatchSize=miniBatchSize,... MiniBatchFcn=@preprocessMiniBatch,... MiniBatchFormat=["SSCB",""]);
各ネットワークをループ処理します。各ネットワークで次を行います。
重みのインデックス、および重みをもつ層の名前を見つけます。
この例の終わりで定義されているサポート関数
setupGradientDistributionAxesを使用して、重みの分布のプロットを初期化します。カスタム学習ループを使用してネットワークに学習させます。
カスタム学習ループでは、各エポックについて、データをシャッフルしてデータのミニバッチをループ処理します。各ミニバッチで次を行います。
関数
dlfevalおよびmodelLossを使用してモデルの損失と勾配を評価します。関数
adamupdateを使用してネットワーク パラメーターを更新します。それぞれの反復で各層の平均勾配の値を保存します。
各エポックの最後に、この例の終わりで定義されているサポート関数 plotGradientDistributions を使用して、各学習可能なパラメーターの層の重みの勾配分布をプロットします。
for activationIdx = 1:numNetworks activationName = activationTypes(activationIdx); net = networks{activationIdx}; % Find the indices of the weight learnables. weightIdx = ismember(net.Learnables.Parameter,"Weights"); % Find the names of the layers with weights. weightLayerNames = join([net.Learnables.Layer(weightIdx),... net.Learnables.Parameter(weightIdx)]); % Prepare axes to display the weight distributions for each epoch % using the supporting function setupGradientDistributionAxes. plotSetup = setupGradientDistributionAxes(activationName,weightLayerNames,numEpochs); % Initialize parameters for the Adam training algorithm. averageGrad = []; averageSqGrad = []; % Train the network using a custom training loop. iteration = 0; start = tic; % Reset minibatchqueue to the start of the data. reset(mbq); % Loop over epochs. for epoch = 1:numEpochs % Shuffle data. shuffle(mbq); % Loop over mini-batches. while hasdata(mbq) iteration = iteration + 1; % Read mini-batch of data. [X,T] = next(mbq); % Evaluate the model loss and gradients using dlfeval and the % modelLoss function. [loss,gradients] = dlfeval(@modelLoss,net,X,T); % Update the network parameters using the Adam optimizer. [net,averageGrad,averageSqGrad] = adamupdate(net,gradients,averageGrad,averageSqGrad,iteration); % Record the loss at every iteration. losses(iteration,activationIdx) = loss; % Record the average gradient of each learnable layer at each iteration. gradientValues = gradients.Value(weightIdx); for ii = 1:numLearnableLayers meanGradients(iteration,activationIdx,ii) = mean(gradientValues{ii},"all"); end end % At the end of each epoch, plot the gradient distributions of the weights % of each learnable layer using the supporting function % plotGradientDistributions. gradientValues = gradients.Value(weightIdx); plotGradientDistributions(plotSetup,gradientValues,epoch) end end
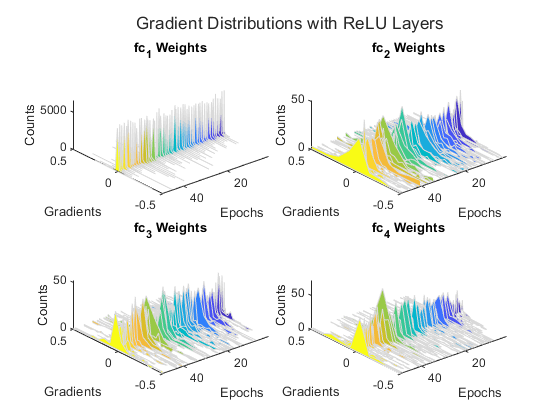

勾配分布のプロットを見ると、シグモイド ネットワークがほとんど 0 に近い小さな勾配の影響を受けていることがわかります。この影響は、初期の層に向かって勾配がネットワーク内を逆伝播するほど顕著になります。
損失の比較
学習済みネットワークの損失を比較します。
figure plot(losses) xlabel("Iteration") ylabel("Loss") legend(activationTypes)

シグモイド ネットワークの損失は、ReLU ネットワークの損失よりも緩やかに減少しています。そのため、このモデルの場合、ReLU 活性化層を使用したほうが高速に学習させることができます。
平均勾配の比較
各学習反復における各層の平均勾配を比較します。
figure tiledlayout("flow") for ii = 1:numLearnableLayers nexttile plot(meanGradients(:,:,ii)) xlabel("Iteration") ylabel("Average Gradient") title(weightLayerNames(ii)) legend(activationTypes) end

平均勾配のプロットは、勾配分布のプロットで見られた結果と一致しています。シグモイド層を含むネットワークの場合、勾配の値は 0 を中心として非常に狭い範囲に分散しています。一方、ReLU 層を含むネットワークの場合、勾配はより広い範囲に分散しているため、勾配消失の可能性が減少して学習速度が増加します。
サポート関数
モデル損失関数
関数 modelLoss は、dlnetwork オブジェクト net、入力データ X のミニバッチ、およびラベルを含む対応するターゲット T を入力として受け取り、学習可能なパラメーターについての損失とその損失の勾配を返します。
function [loss,gradients] = modelLoss(net,X,T) Y = forward(net,X); loss = crossentropy(Y,T); gradients = dlgradient(loss,net.Learnables); end
ミニ バッチ前処理関数
関数 preprocessMiniBatch は、次の手順を使用して予測子とラベルのミニバッチを前処理します。
関数
preprocessMiniBatchPredictorsを使用してイメージを前処理します。入力 cell 配列からラベル データを抽出し、2 番目の次元に沿って categorical 配列にデータを連結します。
カテゴリカル ラベルを数値配列に one-hot 符号化します。最初の次元への符号化は、ネットワーク出力の形状と一致する符号化された配列を生成します。
function [X,T] = preprocessMiniBatch(XCell,TCell) % Preprocess predictors. X = preprocessMiniBatchPredictors(XCell); % Extract label data from cell and concatenate. T = cat(2,TCell{1:end}); % One-hot encode labels. T = onehotencode(T,1); end
ミニバッチ予測子前処理関数
関数 preprocessMiniBatchPredictors は、入力 cell 配列からイメージ データを抽出して数値配列に連結することで、予測子のミニバッチを前処理します。グレースケール入力の場合、4 番目の次元で連結することにより、3 番目の次元が各イメージに追加されます。この次元は、大きさが 1 のチャネル次元として使用されます。
function X = preprocessMiniBatchPredictors(XCell) % Concatenate. X = cat(4,XCell{1:end}); end
分布の計算
関数 gradientDistributions は、ヒストグラムの値を計算し、ビンの中央値とヒストグラムのカウント数を返します。
function [centers,counts] = gradientDistributions(values) % Get the histogram count for the values. [counts,edges] = histcounts(values,30); % histcounts returns edges of the bins. To get the bin centers, % calculate the midpoints between consecutive elements of the edges. centers = edges(1:end-1) + diff(edges)/2; end
勾配分布プロットの軸の作成
関数 setupGradientDistributionAxes は、勾配分布プロットを 3 次元でプロットするのに適した軸を作成します。この関数は、TiledChartLayout オブジェクトとカラーマップを含む構造体配列を返します。この構造体配列は、サポート関数 plotGradientDistributions への入力として機能します。
function plotSetup = setupGradientDistributionAxes(activationName,weightLayerNames,numEpochs) f = figure; t = tiledlayout(f,"flow",TileSpacing="tight"); t.Title.String = "Gradient Distributions with " + activationName + " Layers"; % To avoid updating the same values every epoch, set up axis % information before the training loop. for i = 1 : numel(weightLayerNames) tiledAx = nexttile(t,i); % Set up the label names and titles. xlabel(tiledAx,"Gradients"); ylabel(tiledAx,"Epochs"); zlabel(tiledAx,"Counts"); title(tiledAx,weightLayerNames(i)); % Rotate the view. view(tiledAx, [-130, 50]); xlim(tiledAx,[-0.5,0.5]); ylim(tiledAx,[1,Inf]); end plotSetup.ColorMap = parula(numEpochs); plotSetup.TiledLayout = t; end
勾配分布のプロット
関数 plotGradientDistributions は、TiledChartLayout オブジェクトとカラーマップを含む構造体配列、および特定のエポックにおける値 (層の勾配など) から成る配列を入力として取り、平滑化されたヒストグラムを 3 次元でプロットします。適切な構造体配列の入力を生成するには、サポート関数 setupGradientDistributionAxes を使用します。
function plotGradientDistributions(plotSetup,gradientValues,epoch) for w = 1:numel(gradientValues) nexttile(plotSetup.TiledLayout,w) color = plotSetup.ColorMap(epoch,:); values = extractdata(gradientValues{w}); % Get the centers and counts for the distribution. [centers,counts] = gradientDistributions(values); % Plot the gradient values on the x axis, the epochs on the y axis, and the % counts on the z axis. Set the edge color as white to more easily distinguish % between the different histograms. hold("on"); fill3(centers,zeros(size(counts))+epoch,counts,color,EdgeColor="#D9D9D9"); hold("off") drawnow end end
参考
dlfeval | adamupdate | dlnetwork | minibatchqueue