このページは機械翻訳を使用して翻訳されました。最新版の英語を参照するには、ここをクリックします。
ファイルからgaを実行する
コマンドライン インターフェイスを使用すると、ファイルを使用して、さまざまなオプション設定で遺伝的アルゴリズムを何度も実行できます。たとえば、交叉率を異なる設定で遺伝的アルゴリズムを実行し、どの設定が最良の結果をもたらすかを確認できます。次のコードは、関数 ga を 21 回実行し、options.CrossoverFraction を 0 から 1 まで 0.05 ずつ変化させて、結果を記録します。
options = optimoptions('ga','MaxGenerations',300,'Display','none'); rng default % for reproducibility record=[]; for n=0:.05:1 options = optimoptions(options,'CrossoverFraction',n); [x,fval]=ga(@rastriginsfcn,2,[],[],[],[],[],[],[],options); record = [record; fval]; end
交叉分率に対して fval の値をプロットします。
plot(0:.05:1, record); xlabel('Crossover Fraction'); ylabel('fval')
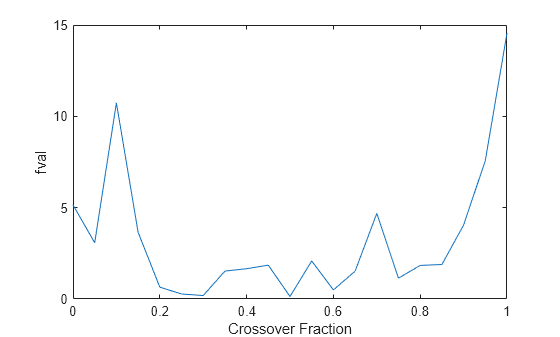
プロットは、options.CrossoverFraction を 0.4 から 0.8 の間の値に設定すると最良の結果が得られることを示唆しています。
ga を 20 回実行し、各交叉分率の fval の値を平均すると、交叉分率の関数として fval のより滑らかなプロットを取得できます。
options = optimoptions('ga','MaxGenerations',300,'Display','none'); rng default % for reproducibility record=[]; fval = zeros(20,1); for n=0:.05:1 options = optimoptions(options,'CrossoverFraction', n); for i = 1:20 [x,fval(i)]=ga(@rastriginsfcn,2,[],[],[],[],[],[],[],options); end meanf = mean(fval); record = [record; meanf]; end plot(0:.05:1, record); xlabel('Crossover Fraction'); ylabel('fval')
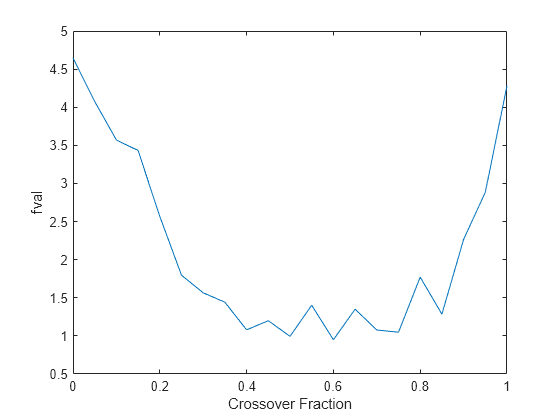
このグラフは、options.CrossoverFraction の最適な選択範囲が 0.4 ~ 0.8 であることを示唆しています。